엽록체기반 SSR marker를 이용한 당귀의 기원 판별
© The Korean Society of Medicinal Crop Science. All rights reserved.
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License ( http://creativecommons.org/licenses/by-nc/3.0 ) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
In the herbal medicinal industry, Angelica gigas Nakai, Angelica sinensis (Oliv.) Diels. and Angelica acutiloba (Siebold & Zucc.) Kitag. are often confused, because the roots of the three species can not be distinguished by their appearance. This confusion can cause serious side effects. In this study, we determined the origins of Angelica roots distributed in the Korean market using the simple sequence repeat (SSR) markers developed based on the A. gigas chloroplast DNA sequence.
We collected twenty seven A. gigas and three A. acutiloba samples from the Seoul, Daegu, and Cheongju herbal medicinal markets. Fifty sections of one collection were mixed and ground to make a powder, which was used for DNA extraction using the cetyl trimethylammonium bromide (CTAB) method. Chloroplast based SSR markers were applied to the DNA for the determination of the species. In addition, polymorphism was found in eight samples. The phylogenetic analysis showed that the A. gigas roots collected from herbal medicinal markets were clearly discriminated from A. sinensis and A. acutiloba even though they were grouped into four clusters.
This study showed that chloroplast based SSR markers would help the discrimination of Angelica roots in the Korean herbal medicinal industry and the markers are useful to prevent confusion between Angelica roots.
Keywords:
Angelica gigas Nakai, Chloroplast, Simple Sequence Repeat, Species Discrimination서 언
참당귀 (Angelica gigas Nakai)는 산형과 당귀속에 속하는 식물로 파종 2 - 3 년 후에 추대가 일어나며 높이는 1 - 2m 정도로 성장하고 자줏빛 줄기를 가지고 있으며 산형화서의 꽃 이 핀다. 약리 활성 성분으로는 coumarine 계의 decursin, decursinol, decursinol angelate 등이 알려져 있다 (Kim et al., 2006b). 약리 작용으로는 혈관 확장 (Rhyu et al., 2005), 기억력 개선 (Kang et al., 2003), 진정 (Choi et al., 2003), 항암 (Lee et al., 2003), 면역세포 활성화 (Han et al., 1998), 항염 (Cho et al., 2015) 등이 보고되고 있다.
한국에서는 참당귀 (A. gigas), 일본에서는 왜당귀 [Angelica acutiloba (Siebold & Zucc.) Kitag.], 중국에서는 중국당귀 [Angelica sinensis (Oliv.) Diels.]를 당귀라고 부르며 사용하고 있어 혼용되기 쉬운데 약리 성분, 약효가 모두 다르기 때문에 오용할 경우 부작용을 일으킬 수 있다 (Kim et al., 2011). 위의 3 종류의 식물처럼 이름이 유사하거나 외양이 유사한 다 른 종의 식물을 혼·오용하는 사례가 발생하고 있다 (Moon et al., 2017). 참당귀와 다른 당귀의 혼용을 막기 위해 해부학적 인 특징을 통해서 세 식물을 구분하기 위한 연구 (Sung et al., 2004)와 성분의 차이로 구분하기 위한 연구 (Jeong et al., 2015)가 보고되었지만 당귀가 유통될 때에는 뿌리를 건조 시켜 절편으로 가공한 상태이기 때문에 외관의 변화에도 변하 지 않는 유전체를 이용한 종판별 방법에 관한 연구가 필요하 다 (Gil et al., 2016).
이렇게 외형적으로 종을 판별하기 힘든 경우 분자마커가 활 용되고 있다 (Jo et al., 2013). 분자 마커란 염기서열 수준에 서 개체 또는 종의 판별을 가능하게 하는 도구이다. Next generation sequencing (NGS)의 개발로 생물의 유전체 정보를 획득하는 것이 용이해짐에 따라 연구가 활발히 진행되고 있으 며 random amplified polymorphic DNA (RAPD) (Bang et al., 2002), amplified fragment length polymorphism (AFLP) (Li et al., 2017), sequence characterized amplified regions (SCARs) (Li et al., 2017), single nucleotide polymorphism (SNP) (Merrill et al., 2016), simple sequence repeat (SSR) (Mohammad-Panah et al., 2017) 등 다양한 분자마커가 개발 되어 사용되고 있다. 당귀의 판별을 위한 RAPD 마커가 보고 되었지만, RAPD 마커는 재현성이 떨어진다는 단점이 있다 (Bang et al., 2002).
SSR 마커는 염기서열에서 단순염기서열의 반복횟수 차이를 이용하여 다형성을 확인하는 마커로써 높은 다형성과 재현성 으로 다양한 식물의 유연관계, 유전적 다양성의 연구 등에 사 용되고 있다 (Kaur et al., 2016). 엽록체의 염기서열은 핵 염 기서열보다 보존되어 있고 모계유전 되기 때문에 종간의 다형 성을 가지는 분자마커를 개발하기에 용이하다고 알려져 있다 (Ebert and Peakall, 2009). 선행 연구에서는 당귀의 엽록체 genome에서 SSR 부위를 조사하고 이 부분을 증폭하는 프라 이머를 제작해서 참당귀, 왜당귀, 중국당귀 등 당귀속 식물 14 종에 적용하여 종간에 다형성을 나타내는 엽록체 기반 SSR 마커를 제작하여 참당귀, 왜당귀, 중국당귀의 종 판별을 수행 한 바 있다 (Park et al., 2016).
이번 연구에서는 서울, 청주, 대구의 한약재 시장에서 유통 되는 당귀 시료 30 점을 수집하여 3 종의 당귀 속 식물을 구 분할 수 있는 2 개의 마커를 활용하여 종을 판별하였다.
재료 및 방법
1. 당귀 종판별 마커
종판별을 위한 마커는 선행 연구 (Park et al., 2016)에서 보고된 (Angelica gigas Nakai), 왜당귀 [Angelica acutiloba (Siebold & Zucc.) Kitag.], 중국당귀 [Angelica sinensis (Oliv.) Diels.]의 PCR product size 비교를 통해 각각의 종을 구분해 낼 수 있는 마커 AGcpSSR2 (AGcpSSR2F: 5’-GCCTTC CAAGCTAACGAT, AGcpSSR2R: 5’-ACCTCTGTCCTATCCA TT)와 AGcpSSR5 (AGcpSSR5F: 5’-GATTCTTTATCCTGTTC GTC, AGcpSSR5R: 5’-CCCACTATTCTAC TATTCTC)의 AGcpSSR2F에 FAM를 부착하고 AGcpSSR5F에 NED를 부 착해서 사용하였다
마커의 종판별 능력을 시험하기 위해 종명을 알고 있는 참 당귀, 왜당귀, 중국당귀 시료 각각 5 개체에서 Plant gDNA Extraction Kit (Biomedic, Bucheon, Korea)를 이용하여 제조 사가 제시한 실험방법에 따라 DNA를 추출하고 마커를 적용하 여 PCR을 수행하였다. PCR mixture의 총 부피는 20㎕로서, 10 ng/㎕ genomic DNA 1㎕, 2 x Taq mix (Dongsheng Biotech, Beijing, China) 10㎕, 5μM forward, reverse primer 각각 2㎕, 멸균 증류수 5㎕로 구성되었다. PCR machine은 Bio-Rad T-100TM Thermal Cycler (Bio-Rad, Hercules, CA, USA)를 이용하였다. PCR 조건은 95℃에서 5 분간 pre-denaturation한 후, 95℃에서 denaturation 30 초, 51℃에서 annealing 30 초, 72℃에서 extention 30 초로 35 cycles로 수행하였고, final extention 과정은 72℃에서 5 분 간 실시하였다. PCR을 수행한 후 4% agarose gel에서 120 V로 30 분 간 전기영동을 수행하여 Gel Doc™ XR+ Gel Documentation System (Bio-Rad, Hercules, CA, USA)에서 결과를 확인하였다 (Fig. 1).
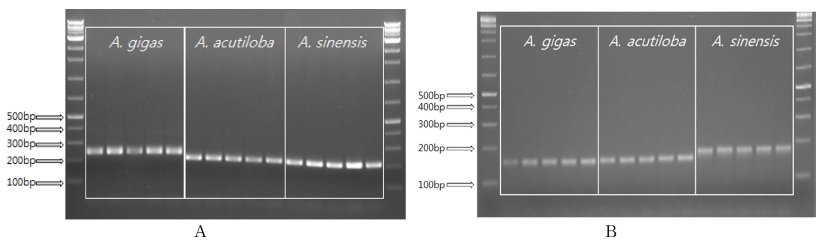
Electrophoretic analysis of the PCR products generated by the polymorphic SSR markers.A; AGcpSSR2, B; AGcpSSR5. PCR analysis was performed to confirm the discrimination function of the Angelica species discrimination markers. PCR was performed by applying the cpSSR markers to 5 samples for each Angelica species and electrophoresis was performed on 2% agarose gel at 100 V for 50 minutes.
2. 시료 수집 및 마커 적용
서울, 청주, 대구의 한약재 시장에서 판매되는 당귀의 뿌리 절편 시료 30 점을 수집하고 참당귀, 왜당귀, 중국당귀 중 어 떤 종의 당귀라고 표시하였는지 기록하였다 (Table 1). 각 시 료 내에서 당귀 절편을 50 개씩 혼합분쇄하고 위에서 제시한 방법으로 DNA를 추출한 후, 추출한 DNA는 Nanodrop (Thermo Fisher Scientific Inc., Waltham, MA, USA) 기기 를 이용하여 농도를 측정하였다. 각각의 DNA 최종농도는 멸 균된 증류수를 이용하여 5 ng/㎕로 조정하였다. 혼합시료의 DNA에 같은 마커를 이용하여 위와 같은 조건으로 PCR을 수 행한 후 PCR 산물을 ABI 3730 DNA Analyzer (Applied Biosystems, Foster, CA, USA)를 이용한 fragment analysis를 통해 product size를 확인하였다 (Table 1). 또한, 종명을 알고 있는 참당귀, 왜당귀, 중국당귀 시료를 포함하여 분석을 실시 하였다.
3. 유전적 다양성 및 유연관계 분석
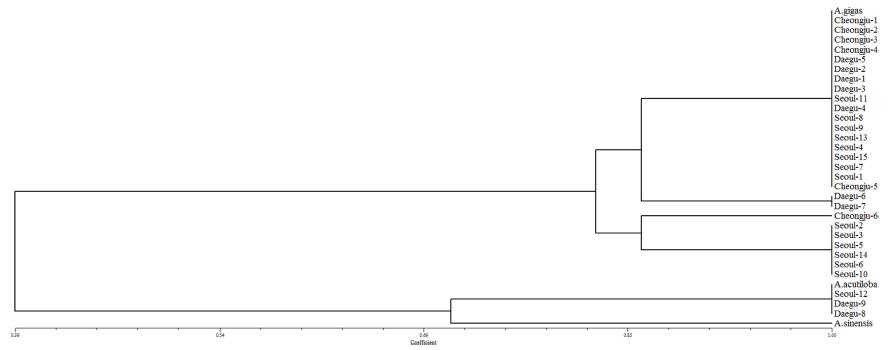
수집된 시료에 대한 다양성 분석은 각 마커를 적용하였을 때의 PCR product size를 분석함으로써 이루어졌다. 위의 방 법으로 확인된 PCR product 중 각 size의 유무에 따라 ‘1’과 ‘0’로 표시하여 매트릭스로 변환하였다. 매트릭스를 이용한 유 연관계 분석은 NTSYS-pc 2.11 프로그램 (Exeter Software, Setauket, NY, USA)을 이용하여 실시하고 phylogenetic tree를 작성하였다. UPGMA 알고리즘을 통해 생산된 데이터를 sequential hierarchical agglomerative non-overlapping (SHAN) 클러스터링을 거쳐 각 샘플간 유연관계를 분석하였다.
결과 및 고찰
1. 수집된 당귀의 기원 판별
서울, 청주, 대구의 시장에서 수집한 30 점의 시료에 마커를 적용하여 PCR을 수행하였고, PCR product는 ABI 3730 DNA Analyzer를 통해 pruduct size를 확인하였다 (Table 1).
AGcpSSR2 마커는 참당귀 (Angelica gigas Nakai), 왜당귀 [Angelica acutiloba (Siebold & Zucc.) Kitag.], 중국당귀 [Angelica sinensis (Oliv.) Diels.] 중에서 참당귀를 구분할 수 있는 마커이다 (Fig. 1A) (Park et al., 2016). AGcpSSR2 마커만으로는 왜당귀와 중국당귀를 구분할 수 없기 때문에 AGcpSSR5 마커를 이용하여 추가로 분석하였다 (Fig. 1B, Table 1).
AGcpSSR5 마커는 위의 세 종류의 당귀 중에서 중국당귀 를 구분할 수 있는 마커로 보고되었는데 (Fig. 1B) (Park et al., 2016) 본 실험의 결과 세 종류의 당귀를 모두 구분할 수 있었다. 그 이유는 선행연구에서는 전기영동 패턴만을 가지고 분석하였으나 이번 연구에서는 프라이머를 형광으로 표지하여 fragment analyzer를 이용한 결과 정밀한 분석이 가능했기 때 문으로 사료된다. 18, 29, 30 번 시료는 AGcpSSR2 마커를 적용한 결과에서 왜당귀 혹은 중국당귀로 추측되었는데, AGcpSSR5 마커의 적용으로 왜당귀로 확정할 수 있었다 (Table 1).
기원 판별 결과 30 개의 시료 중 표기된 식물 종과 PCR 증폭산물의 패턴이 다른 시료가 없었으므로 한약재 시장에서 당귀의 기원 관리가 잘 되고 있다고 할 수 있었다.
Genotyping을 통하여 얻어진 데이터를 기반으로 33 개의 샘 플을 이용하여 계통학적 분석을 실시한 결과 참당귀는 일부 다 형성을 보인 것으로 인하여 4 개의 cluster로 나누어지는 것을 볼 수 있었으나 왜당귀와 중국당귀와는 확실한 구분이 되어 종 을 판별하는 데에는 어려움이 없다고 판단되었다 (Fig. 2).
2. 다형성을 보이는 참당귀 시료의 분석
두 개의 마커를 적용했을 때, 8 점의 참당귀 시료 (8, 9, 11, 12, 16, 20, 27, 28 번 시료)에서 2 개의 band size가 확인되었다. 절편을 혼합 분쇄하여 추출한 DNA를 실험에 사 용했기 때문에 이런 현상이 생겼다고 판단하여 2 점의 시료 (7, 12 번 시료)에서 각 시료 당 절편 10 개씩으로부터 독립 적으로 DNA를 추출하고 AGcpSSR5 마커를 사용하여 위와 같은 실험을 수행하였다. 그 결과 모든 절편에서 각각 1 개의 band size만 증폭되었다 (Table 2).
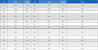
Analysis of the individual sections of the samples showed multi-bands from AGcpSSR5 marker analysis.
절편을 혼합해서 추출한 DNA에 AGcpSSR5 마커를 적용하 였을 때 2 개의 band가 검출되었고, 각 절편에서 따로 DNA 를 추출하여 동일한 적용하였을 때는 각각 1 band가 검출 된 것으로 보아 혼합시료 안에 두 가지의 변이를 가진 당귀가 혼 합되어 있다고 생각된다. 당귀는 한국에서 오래전부터 사용해 온 약초이지만 대부분 야생종을 채집하여 사용했고, 현대에 와 서도 주요 작물이 아니라는 이유로 계통이 확립되지 않았으며 타가수정 작물로 알려져 있고 (Kim et al., 2006a), 타가수정 식물은 기존의 형질이 유지되기 힘든 것으로 보고되고 있다 (Shim et al., 1998). 이러한 이유로 현재 농가에서는 다양한 모본을 가진 계통을 구분 없이 재배하고 소비자에게 판매하기 때문에 유통되는 당귀 내에 다양한 변이가 존재하게 되었다고 사료된다.
3. 분자마커를 이용한 기원판별
한국, 일본, 중국에서 오래전부터 서로 다른 3 종의 식물을 당귀라는 같은 이름으로 불러왔으나 기원 식물이 서로 다르기 때문에 세 종류의 당귀를 구분하려는 다양한 연구가 수행되었 다 (Bang et al., 2002; Sung et al., 2004; Jeong et al., 2015). 그러나 관능검사나 성분분석을 이용해서 세 종을 구분 하는 방법은 재배 환경, 가공 방법에 따라 오류가 생길 수 있 다는 단점이 언급되었고 환경에 영향을 받지 않는 DNA를 이 용한 마커에 대한 연구에서도 RAPD, AFLP, SCAR등의 기술 은 시료의 상태, 조직 부위별 및 분석 조건에 따라 밴드 패턴 이 다르게 나타난다는 점이 실제 유통과정에서 사용되기에 큰 단점으로 작용한다 (Lee et al., 2017). 반면에 SSR 마커는 높은 다형성과 재현성으로 RAPD 등의 단점을 극복할 수 있기 때문 에 이를 이용한 연구가 보고 되고 있다 (Belaj et al., 2003).
본 연구에서 당귀 속 식물에서 엽록체 DNA를 기반으로 개 발된 SSR 마커를 이용해서 국내 한약재 시장에서 유통되는 당귀 속 식물의 혼 오용실태를 확인하였다. 이 연구결과를 통 해 당귀 속 기원식물을 판별하고 한약재 혼·오용 방지에 기여 할 수 있을 것으로 생각된다.
감사의 글
본 연구는 농촌진흥청 연구사업(과제번호: PJ01102201)의 지원에 의해 이루어진 결과로 이에 감사드립니다.
References
- K.H. Bang, H.S. Yu, D.H. Koo, J.H. Cho, H.W. Park, N.S. Seong, S.I. Park, H.S. Kim, Selection of RAPD marker to discriminate the bolting-resistant varieties and commercial dried medicinal materials of Angelica species., Hanguk Yakyong Changmul Hakhoe Chi, (2002), 10, p46-50.
-
A. Belaj, Z. Satovic, G. Cipriani, L. Baldoni, R. Testolin, L. Rallo, I. Trujillo, Comparative study of the discriminating capacity of RAPD, AFLP and SSR markers and of their effectiveness in establishing genetic relationships in olive., Theor. Appl. Genet, (2003), 107, p736-744.
[https://doi.org/10.1007/s00122-003-1301-5]

-
J.H. Cho, J.E. Kwon, Y. Cho, I. Kim, S.C. Kang, Antiinflammatory effect of Angelica gigas via heme oxygenase (HO)-1 expression., Nutrients, (2015), 7, p4862-4874.
[https://doi.org/10.3390/nu7064862]

-
S.S. Choi, K.J. Han, H.K. Lee, E.J. Han, H.W. Suh, Antinociceptive profiles of crude extract from roots of Angelica gigas Nakai in various pain models., Biol. Pharm. Bull, (2003), 26, p1283-1288.
[https://doi.org/10.1248/bpb.26.1283]

-
D. Ebert, R. Peakall, Chloroplast simple sequence repeats(cpSSRs): Technical resources and recommendations forexpanding cpSSR discovery and applications to a wide array of plant species., Mol. Ecol. Resour, (2009), 9, p673-690.
[https://doi.org/10.1111/j.1755-0998.2008.02319.x]

- J. Gil, S.I. Park, Y. Lee, H.B. Kim, S.C. Kim, O.T. Kim, S.W. Cha, C.S. Jung, Y. Um, Current status and prospects ofthe authentication of Angelica species., J. Plant Biotechnol, (2016), 43, p151-156.
-
S.B. Han, Y.H. Kim, C.W. Lee, S.M. Park, H.Y. Lee, K.S. Ahn, I.H. Kim, H.M. Kim, Characteristic immunostimulation by angelan isolated from Angelica gigas Nakai., Immunopharmacology, (1998), 40, p39-48.
[https://doi.org/10.1016/s0162-3109(98)00026-5]

- S.Y. Jeong, H.M. Kim, K.H. Lee, K.Y. Kim, D.S. Huang, J.H. Kim, R.S. Seong, Quantitative analysis of marker compounds in Angelica gigas, Angelica sinensis, and Angelica acutiloba by HPLC/DAD., Chem. Pharm. Bull. (Tokyo), (2015), 63, p504-511.
-
I.H. Jo, K.H. Bang, Y.C. Kim, J.U. Kim, M.R. Shin, J.Y. Moon, B.S. Noh, D.Y. Hyun, D.H. Kim, S.W. Cha, H.S. Kim, Analysis of mitochondrial DNA sequence and molecular marker development for identification of Panax species., Hanguk Yakyong Changmul Hakhoe Chi, (2013), 21, p91-96.
[https://doi.org/10.7783/kjmcs.2013.21.2.91]

-
S.Y. Kang, K.Y. Lee, M.J. Park, Y.C. Kim, G.J. Markelonis, T.H. Oh, Y.C. Kim, Decursin from Angelica gigas mitigates amnesia induced by scopolamine in mice., Neurobiol. Learn. Mem, (2003), 79, p11-18.
[https://doi.org/10.1016/s1074-7427(02)00007-2]

- K. Kaur, V. Sharma, V. Singh, M.S. Wani, R.C. Gupta, (2016), https://link.springer.com/content/pdf/10.1007%2Fs13205-016-0469-8.pdf.
- D.H. Kim, H.W. Park, C.G. Park, J.S. Sung, N.S. Seong, Effects of insects on pollination in Angelica gigas Nakai and Angelica acutiloba Kitagawa., Hanguk Yakyong Changmul Hakhoe Chi, (2006), a 14, p217-220.
-
M.R. Kim, A.M. Abd El-Aty, J.H. Choi, K.B. Lee, J.H. Shim, Identification of volatile components in Angelica species using supercritical-CO2 fluid extraction and solid phase microextraction coupled to gas chromatography-mass spectrometry., Biomed. Chromatogr, (2006), b 20, p1267-1273.
[https://doi.org/10.1002/bmc.696]

- S.A. Kim, H.K. Oh, J.Y. Kim, J.W. Hong, S.I. Cho, A review of pharmacological effects of Angelica gigas, Angelica sinensis, Angelica acutiloba and their bioactive compounds., Journal of Korean Oriental Medicine, (2011), 32, p1-24.
-
S. Lee, Y.S. Lee, S.H. Jung, K.H. Shin, B.K. Kim, S.S. Kang, Anti-tumor activities of decursinol angelate and decursin from Angelica gigas., Arch. Pharm. Res, (2003), 26, p727-730.
[https://doi.org/10.1007/bf02976682]

-
S.W. Lee, S.J. Lee, E.H. Han, E.C. Sin, K.M. Cho, Y.H. Kim, Current status on the development of molecular markers for differentiation of the origin of Angelica spp., J. Plant Biotechnol, (2017), 44, p12-18.
[https://doi.org/10.5010/jpb.2017.44.1.012]

-
S.F. Li, L.J. Wang, C.L. Deng, W.J. Gao, Identification of male-specific AFLP and SCAR markers in the dioecious plant Humulus scandens., Mol. Cell. Probes, (2017), 34, p68-70.
[https://doi.org/10.1016/j.mcp.2017.05.007]

- K.R. Merrill, C.E. Coleman, S.E. Meyer, E.A. Leger, K.A. Collins, (2016), http://www.bioone.org/doi/full/10.3732/apps.1600068.
-
N. Mohammad-Panah, N. Shabanian, A. Khadivi, M-S. Rahmani, A. Emami, Genetic structure of gall oak(Quercus infectoria) characterized by nuclear and chloroplast SSR markers., Tree Genet. Genomes, (2017), 13, p70-81.
[https://doi.org/10.1007/s11295-017-1146-8]

- B.C. Moon, W.J. Kim, G. Choi, A case report of new commercial adulterant of Araliae continentalis radix based on the analysis of ITS2 DNA barcode sequences., Korean Herbal Medicine Informatics, (2017), 5, p33-39.
-
S.I. Park, S. Kim, J. Gil, Y. Lee, H.B. Kim, J.H. Lee, S.C. Kim, C.S. Jung, Y. Um, Development of chloroplast DNAbased simple sequence repeat markers for Angelica species differentiation., Hanguk Yakyong Changmul Hakhoe Chi, (2016), 24, p317-322.
[https://doi.org/10.7783/kjmcs.2016.24.4.317]

-
M.R. Rhyu, J.H. Kim, E.Y. Kim, Radix Angelica elicits both nitric oxide-dependent and calcium influx-mediated relaxation in rat aorta., J. Cardiovasc. Pharmacol, (2005), 46, p99-104.
[https://doi.org/10.1097/01.fjc.0000164092.88821.49]

- T.H. Shim, H.H. Lee, S.Y. Lee, Y.S. Choi, Composition of buckwheat(Fagopyrum esculentum Moench) cultivars fromKorea., Korean J. Food Sci. Technol, (1998), 30, p1259-1266.
- J.S. Sung, K.H. Bang, C.H. Park, C.G. Park, H.S. Yu, H.S. Park, H.W. Park, N.S. Seong, Discrimination of Angelicae radix based on anatomical characters., Hanguk Yakyong Changmul Hakhoe Chi, (2004), 12, p67-72.