SSR 마커를 이용한 산양삼의 유전적 다양성 분석
†Corresponding author: +82-43-261-2519 chanmoon@chungbuk.ac.kr
© The Korean Society of Medicinal Crop Science. All rights reserved.
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License ( http://creativecommons.org/licenses/by-nc/3.0 ) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Panax ginseng C. A. Meyer is wood-cultivated ginseng (WCG) in Korea which depends on an artificial forest growth method. To produce this type of ginseng, various P. ginseng cultivars can be used. To obtain a WCG similar to wild ginseng (WG), this method is usually performed in a mountain using seeds or seedlings of cultivated ginseng (CG) and WG. Recently, the WCG industry is suffering a problem in that Panax notoginseng (Burk.) F. H. Chen or Panax quinquefolium L. are being sold as WCG Korean market; These morphological similarities have created confusion among customers.
WCG samples were collected from five areas in Korea. After polymerase chain reaction (PCR) amplification using the primer pair labeled with fluorescence dye (FAM, NED, PET, or VIC), fragment analysis were performed. PCR products were separated by capillary electrophoresis with an ABI 3730 DNA analyzer. From the results, WCG cultivated in Korea showed very diverse genetic background.
In this study, we tried to develop a method to discriminate between WCG, P. notoginseng or P. quinquefolium using simple sequence repeat (SSR) markers. Furthermore, we analyzed the genetic diversity of WCG collected from five cultivation areas in Korea.
Keywords:
Panax ginseng, Simple Sequence Repeat, Wood-Cultivated Ginseng서 언
인삼 (Panax ginseng C. A. Meyer)은 다년생의 반음지성 숙근초로서, 식물학적으로 두릅나무과 인삼속 (Panax)에 속 한다. 대표적으로 알려져 있는 다양한 인삼속 식물 중 약 용으로 이용되는 것은 고려인삼 (P. ginseng), 죽절삼 (Panax japonicum C. A. Meyer), 전칠삼 [Panax notoginseng (Burk.) F. H. Chen], 화기삼 (Panax quinquefolium L.) 등 4 종이 있으며 그 중에서도 일반적으로 인삼은 “Panax ginseng” 을 지칭한다 (Kim et al., 2009).
인삼은 생육환경에 따라 재배삼, 산양삼, 산삼으로 분류한다. 재배삼은 밭이나 논에서 재배하는 인삼을 말하고, 산양삼은 산 삼의 씨앗이나 인삼묘종을 산으로 옮겨 인위적으로 산에서 재 배한 삼을 일컬으며 산삼은 인삼이 야생상태에서 자연 발아하 여 성장한 것을 말한다 (Shin et al., 2001).
인삼의 경우 재배년근 및 근중에 따른 체계적인 편급, 등급 이 설정되어 있다. 그러한 이유는 인삼에 대한 재배적지 기준 설정 (Hyun et al., 2009a), 재배지의 토양 화학성에 따른 생 리장해 유형 (Hyun et al., 2009b), 직파와 이식재배에 따른 인삼의 부위별 생육특성 (Li et al., 2010) 등의 재배 및 생육 에 관해 오랜 기간 많은 연구가 수행되어 그에 따른 품질지표 설정이 가능했기 때문이다. 산양삼의 경우에도 임간재배 인삼 의 직파 및 이식 재배에 따른 생육 (Lim et al., 2003)에 관 한 연구 등이 이루어지긴 하였으나 아직까지도 산양삼의 재배 는 지역별, 경작자별로 상이하다. 이러한 이유로 산양삼의 재 배는 전국적으로 이루어지고 있으나 시중에 유통되는 산양삼 은 재배기간이 3 - 10 년에 해당하는 다양한 년근의 산양삼이 판매되고 있으며 년근에 따른 차이와 뿌리의 형태, 근중에 따 라 판매되는 가격의 편차가 크다. 또한 국내 산양삼은 고려인 삼을 이용하여 재배하는데 전칠삼, 화기삼 등의 유사종이 한 국 산양삼으로 둔갑하여 유통되고 이러한 고려인삼과 유사종 의 경우 일반 소비자들이 판별하기 어려워 문제가 제기되고 있다.
인삼 판별법 중에서 최근 개발된 분자생물학적 판별법은 형 태적 및 이화학적 판별에서의 한계를 보완하고 결과의 신뢰도 를 향상시켜 종 분류 및 계통분석에 관한 연구에 널리 활용되 고 있다 (Kim et al., 2007). 특히 polymerase chain reaction (PCR) 기법을 이용한 random amplified polymorphic DNA (RAPD), single nucleotide polymorphism (SNP), simple sequence repeat (SSR) 등은 소량의 DNA를 이용하여 식물의 생장과 관계없이 모든 조직에서 안정적으로 탐색할 수 있으며 비교적 적은 비용으로 빠른 시간 안에 분석할 수 있다는 장점 을 가진다 (Jo et al., 2013).
DNA 표지인자 중 SSR은 genome상에 존재하는 단순반복 염기서열의 반복횟수의 차이로 인해 다형성이 나타난다. 또한, 높은 재현성을 보이고 분석이 비교적 간편하며 근연 종 내에 서의 호환성을 갖는다. 이로 인해 종 내에서의 품종, 개체에 대한 유전적 다양성과 유연관계를 평가하는데 많이 이용되고 있다 (Li et al., 2002; Um et al., 2016). 지금까지의 인삼 및 산양삼 관련 연구는 유전적 형질에 대한 감별 (Choi et al., 2008), 추출물에 대한 품질 특성 (Bae et al., 2009), 부 위별 성분과 이화학적 특성 (Hong et al., 2010) 등에 대한 연구가 주로 많이 이루어졌으며, 국내 산양삼의 유전적 다양 성에 대한 연구는 부족한 실정이다.
따라서 본 실험은 국내 5 개 지역에서 재배되는 산양삼 80 자원에 대해 선행연구자들에 의해 개발된 인삼품종 식별이 가 능한 SSR 마커로 유전적 다양성을 분석하고 외국종과의 유연 관계를 분석하여 계통도를 작성해 차후 국내 유통되는 산양삼 의 혼·오용 방지를 위하여 수행하였다.
재료 및 방법
1. 시료 수집
본 실험에 사용된 산양삼은 강원도 횡성, 충청북도 주덕, 충 청북도 청주, 경기도 부천, 울산 지역에 있는 5 개의 산양삼 농가에서 재배되어 유통되는 3 - 10 년근 산양삼 (Panax ginseng C. A. Meyer)을 수집하여 사용하였다. 총 80 자원의 산양삼은 강원도 횡성에서 15 자원, 충청북도 주덕에서 17 자 원, 충청북도 청주에서 15 자원, 경기도 부천에서 15 자원, 울 산에서 18 자원을 수집하였다. 산양삼과 전칠삼 [Panax notoginseng (Burk.) F. H. Chen], 화기삼 (Panax quinquefolium L.)의 유연관계를 확인하기 위해 국립원예특작과학원에서 전칠삼과 화기삼을 수집하였다 (Table 1).
2. DNA 추출 및 PCR 반응
DNA 분석을 위하여 수집된 산양삼과 인삼 (Panax ginseng C. A. Meyer) 잎 조직 100㎎을 사용하였다. DNA 추출은 DNeasy plant mini kit (QIAGEN GmbH, Hilden, Germany)을 사용하였다. 추출된 genomic DNA는 Nanodrop (Thermo Fisher Scientific Inc., Waltham, MA, USA)을 이 용하여 농도를 측정한 후 멸균된 증류수를 이용하여 최종농도 를 5 ng/㎕로 조정하여 PCR에 사용하였다.
실험에 사용된 마커는 Um 등 (2016)의 선행연구에서 개발 된 SSR 마커를 사용하였다 (Table 2). 선행연구자들의 실험방 법에 따라 프라이머에 형광물질을 합성하여 사용하였고 PCR 반응액의 조성은 5 x Phusion HF buffer 5㎕, 10 mM dNTPs 0.5㎕, 10 μM primer F 1.25㎕, 10 μM primer R 1.25㎕, Template DNA 3㎕, DW 13.75㎕, 그리고 Phusion DNA polymerase (NEW ENGLAND BioLabs, Ipswich, MA, USA) 0.25㎕로 총 25㎕로 하였다. PCR은 T100™ Thermal Cycler (Bio-Rad, Hercules, CA, USA)를 사용해 pre-denaturation 94℃에서 5 분, denaturation 94℃에 서 30 초, annealing 51℃ - 62℃에서 1 분, extension 72℃에 서 1 분으로 35 회 반복 수행하고, 마지막 extension 72℃에 서 5 분을 실행하였다.
3. 유전형 및 데이터 분석
PCR 수행 후 생성된 증폭 산물은 ABI3730xl DNA 분석기 와 GeneScan™-500 LIZ size standard (Applied Biosystems, Foster, CA, USA)를 사용하여 분석하였다. 각 마커에 대한 주 요 대립 유전자 빈도 (major allele frequency, MAF), 유전자형 의 수 (number of genotypes, NG), 대립 유전자의 수 (number of alleles, NA), 예상되는 이형 접합성 (expected heterozygosity, HE), 관찰된 이형 접합성 (observed heterozygosity, HO) 및 다 형성 지수 (polymorphic information content, PIC)를 통계 프로그램인 PowerMarker ver. 3.23 ( http://statgen.ncsu.edu/ powermarker/index.html )을 사용하여 분석하였다 (Liu and Muse, 2005).
PowerMarker ver. 3.23의 Shared Allele distance method 를 이용하여 자원의 유전적 거리를 분석하였고 MEGA ver. 5.05 (Arizona State University, Tempe, AZ, USA) 프로그램 을 사용하여 unweighted pair group method with arithmetic mean (UPGMA) phylogenetic tree를 작성하였다. 지역별 산양 삼과 전칠삼, 화기삼의 집단별 대립유전자 분포 및 특정 대립 유전자는 GenAlEx ver. 6.5 ( http://biology-assets.anu.edu.au/ GenAlEx/Welcome.html )를 사용하여 분석하였다.
결과 및 고찰
1. SSR 마커 분석
실험에 사용된 유전자원의 유전형 데이터를 분석한 결과 주 요 대립 유전자 빈도 (MAF)는 평균 0.649로 나타났으며 PgSSR07과 PgSSR53 마커의 경우 각각 0.99, 0.98로 매우 높은 빈도수로 대립유전자가 증폭되었다. 유전자형의 수 (NG) 는 평균 9.5 개로 PgSSR40, PgSSR51이 15 개로 가장 많은 유전자형이 존재하였고 PgSSR07이 2 개로 가장 적었다. 대립 유전자의 수 (NA)는 PgSSR51이 10 개, PgSSR07이 2 개로 평균 6.25 개의 대립유전자를 갖는 것으로 확인되었다.
예상되는 이형접합성 (HE)의 평균은 0.462이었고 PgSSR51 이 0.75로 가장 높았으며 PgSSR07이 0.02로 가장 낮았다. 관 찰된 이형 접합성 (HO)의 평균은 0.299로 분석되었다. 마커의 다형성 지수는 값이 높을수록 해당 마커의 다형성이 높아 개 체의 식별 또는 유전적 다양성을 평가하기에 유용하며 자원의 다양성 분석에 유용한 마커라고 보고된 바 있다 (Kim et al., 2016). 본 실험 결과에서 마커의 다형성 지수 (PIC)는 PgSSR51이 0.71로 가장 높고, PgSSR07이 0.02로 가장 낮은 다형성 지수를 나타내어 평균 0.427로 분석되었고 실험에 사 용된 산양삼 (Panax ginseng C. A. Meyer) 유전자원의 유전 적 다양성 분석 및 전칠삼 [Panax notoginseng (Burk.) F. H. Chen], 화기삼 (Panax quinquefolium L.)과의 유연관계를 확인하는데 사용되었다 (Table 3).
2. 자원의 특정 대립유전자 및 집단별 대립유전자의 분포
집단별 대립유전자의 분포 및 특정 대립유전자를 분석하기 위해 산양삼 유전자원을 수집한 5 개 지역 및 전칠삼, 화기삼 을 집단으로 하여 6 개의 집단에 대해 분석하였다. 그 결과 PgSSR37을 제외한 PgSSR07, PgSSR09, PgSSR08, PgSSR17, PgSSR40, PgSSR51, PgSSR53등 7 개의 SSR 마커에서 전칠 삼과 화기삼에 특정 대립유전자를 나타내어 산양삼과 전칠 삼, 화기삼의 판별이 가능함을 확인할 수 있었다. PgSSR17, PgSSR40은 부천지역 집단에서 특정 대립유전자를 확인할 수 있었고 PgSSR37, PgSSR51은 부천지역 집단과 전칠삼, 화기 삼 집단에서만 존재하는 대립유전자를 확인하였다 (Table 4). PgSSR07과 PgSSR53의 경우 SSR 마커의 분석 결과 PIC 값 은 각각 0.02, 0.05로 매우 낮고 대립유전자의 수 (NA)는 각 각 2 개, 3 개를 나타내어 유전자원 개체간의 다양성을 분석 하는데 유용하지 않았으나 분석결과 전칠삼과 화기삼 집단에 서 특정 대립유전자를 확인 할 수 있었다. 때문에 PgSSR07과 PgSSR53의 경우 마커의 PIC 값은 낮지만 산양삼과 전칠삼, 화기삼에 대한 종 판별을 할 수 있는 유용한 분자표지로 사용 될 수 있다고 판단되었다 (Table 3).
대립유전자의 분포를 집단별로 분석한 결과 횡성 지역에서 수집된 집단이 27 개로 가장 많은 대립유전자 수를 갖고 있으 며 가장 많은 유전적 다양성을 나타내었고 전칠삼과 화기삼 집 단의 경우 13 개의 대립유전자 수를 나타내어 가장 적었다. 울 산지역에서 수집된 집단의 경우 18 개의 대립유전자 수를 나 타내었고 수집된 5 개 지역의 산양삼 집단 중 가장 적은 대 립유전자 수를 나타내었다. 수집된 5 개 지역 산양삼 집단의 분석결과 울산지역 집단이 가장 적은 유전적 다양성을 갖고 있는 것으로 확인되었으며 부천 24 개, 주덕 25 개, 청주 25 개의 대립유전자를 나타내며 비슷한 수준의 유전적 다양성을 확인하였다 (Table 4).
실험에 사용된 산양삼 자원의 집단별 유전자형에 따라 대립 유전자의 분포 및 특정 대립유전자를 확인 할 수 있었고, 이 결과를 바탕으로 각각의 수집된 형태에 따라 수집지역 내에서 의 유전적 다양성 및 집단 사이의 차이점과 전칠삼 및 화기삼 과의 유전적 차이를 식별할 수 있었다.
3. 계통분석을 통한 산양삼의 유연관계 분석
Phylogenetic tree를 이용하여 5 개 지역에서 수집된 산양삼 유전자원 및 전칠삼, 화기삼에 대해 개체별, 집단별 유연관계 를 확인하였다 (Fig. 1, 2). 개체별 유연관계를 확인한 결과 수집된 산양삼 80 자원은 전칠삼, 화기삼과 다른 집단을 형성 하였다. 8 개의 SSR 분자표지를 사용한 유전적 다양성 분석 에서 5 개 지역의 산양삼 유전자원은 울산지역 유전자원의 다 양성이 가장 적고 18 개의 유전자원 중 13 개 자원이 가까운 군집을 형성하였다. 부천지역에서 수집된 15 개의 유전자원의 경우 7 번, 11 번 자원을 제외한 13 개 자원이 군집을 형성 하였다. 청주, 주덕, 횡성 지역에서 수집된 유전자원들은 phylogenetic tree에서 군집을 형성하지 않고 다양하게 분포하 고 있었다. 실험에 사용된 유전자원의 집단별 분석 결과에서 는 청주, 주덕 지역의 집단이 충청북도 내에서 지리적으로 가 까운 집단이며 집단별 phylogenetic tree 분석 결과에서도 가 까운 유연관계를 나타내었다.
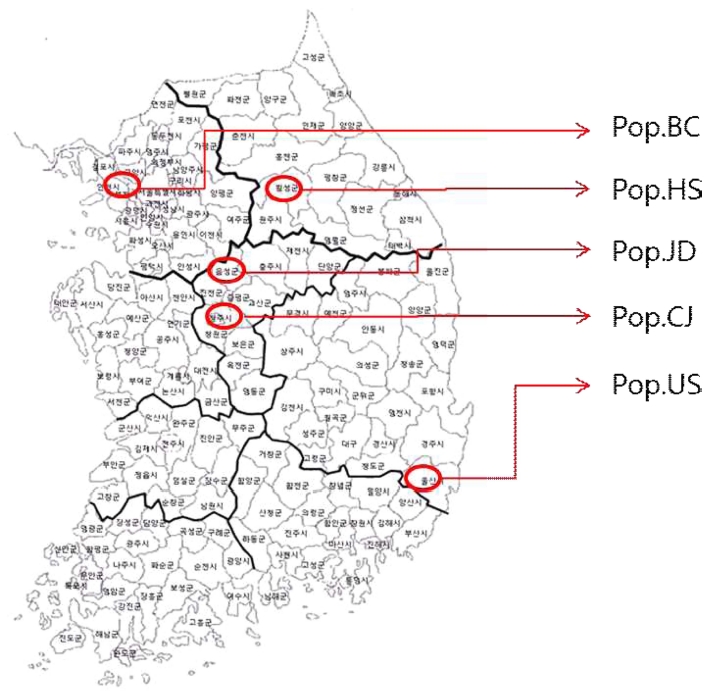
Geographic distribution of the wood-cultivated ginseng collected from South Korea.Pop.BC; Bucheon regional groups, Pop.CJ; Cheongju regional groups, Pop.HS; Hoengseong regional groups, Pop.JD; Judeok regional groups, Pop.US; Ulsan regional groups.
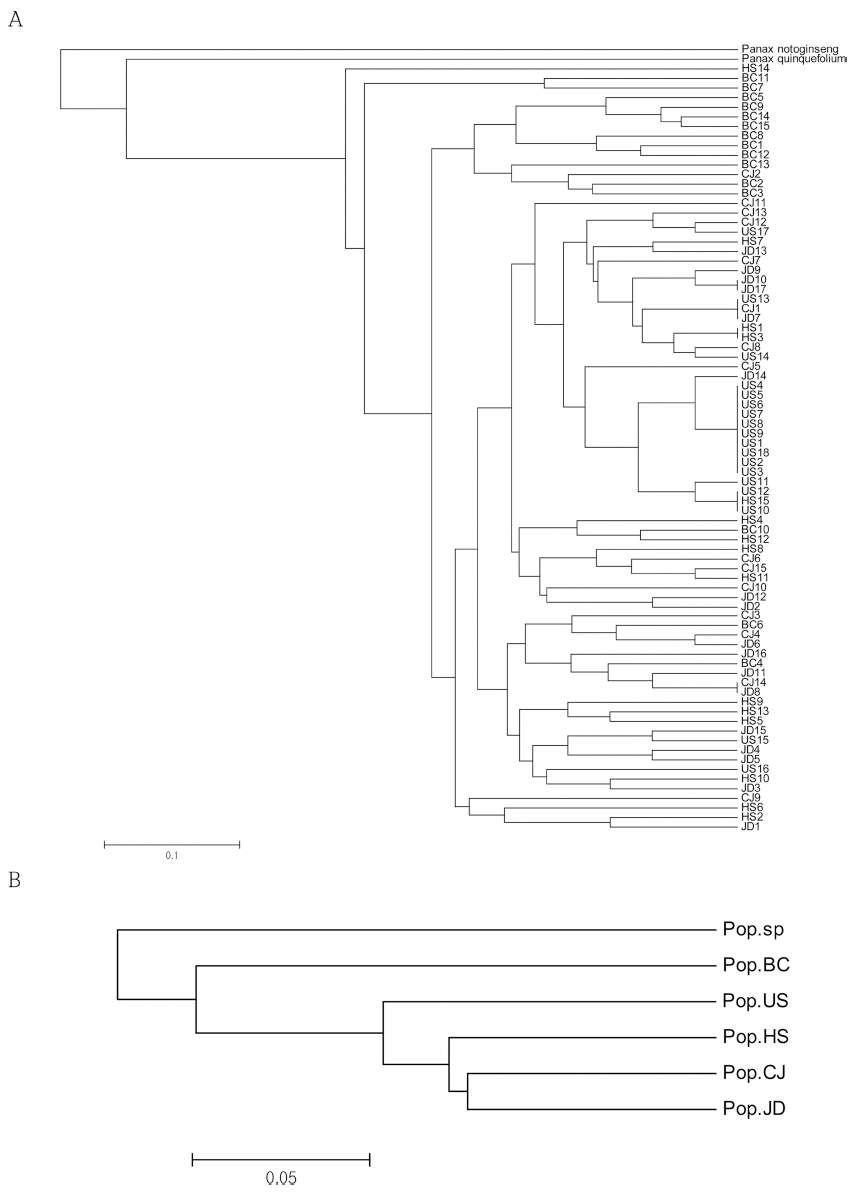
A UPGMA tree based on genetic distances from wood-cultivated ginseng, variety of Panax ginseng, Panax notoginseng and Panax quinquefolium.A; genetic distances of individual genetic resource, BC; Bucheon, CJ; Cheongju, HS; Hoengseong, JD; Judeok, US; Ulsan, B; genetic distances of 7 collected groups, Pop.BC; Bucheon regional groups, Pop.CJ; Cheongju regional groups, Pop.HS; Hoengseong regional groups, Pop.JD; Judeok regional groups, Pop.US; Ulsan regional groups, Pop.sp; Panax notoginseng and Panax quinquefolium groups.
본 연구에서는 기존에 고려인삼의 품종판별을 위해 개발된 8 개의 SSR 분자표지를 이용하여 국내에서 유통되는 산양삼 유전자원을 5 개 지역에서 수집하여 유전적 다양성을 확인하 였고 전칠삼, 화기삼과의 유연관계를 확인하였다. 그 결과 실 험에 사용된 산양삼 유전자원은 고려인삼의 종자, 또는 묘삼 을 이용해 임간재배를 통해 생산되었고 전칠삼, 화기삼과 같 은 유사종의 인삼이 불법 유통되어 판매된 자원이 아님을 확 인할 수 있었다.
본 실험에서 5 개 지역 산양삼 유전자원의 다양성을 확인 한 결과, 수집된 산양삼 유전자원이 다양한 유전형을 나타내 었고 현재 국내에서 재배되는 산양삼이 다양한 품종 또는 지 역종의 고려인삼이 이용되고 있음을 확인할 수 있었다. 때문 에 지역별, 농가별로 품질 또는 등급에 대해 편차가 크게 발 생하여 인삼과 같이 품질, 등급 표준 체계의 확립에 큰 어려 움이 존재한다고 판단된다. 산양삼의 체계적인 유통, 판매 및 품질, 등급 지표의 설정을 위해서는 국내 재배되는 다양한 형 태의 산양삼 유전자원에 대한 재배환경, 생육특성 조사 및 이 러한 유전형 결과를 바탕으로 체계적인 연구가 필요하며 본 실험의 결과는 이러한 연구에 기초자료로 활용 될 수 있을 것 이라 사료된다.
감사의 글
본 연구는 산림청 임업기술연구개발사업(과제번호: 2014 108A00-1417-AA02)의 지원에 의해 이루어진 결과로 이에 감 사드립니다.
References
- M.J. Bae, S.J. Kim, E.J. Ye, H.S. Nam, E.M. Park, Antioxidant activity of tea made from Korean mountaincultivated ginseng leaves and its influence on lipid metabolism., Korean Journal of Food Culture, (2009), 24, p77-83.
-
Y.E. Choi, C.H. Ahn, B.B. Kim, E.S. Yoon, Development of species specific AFLP-derived SCAR marker for authentication of Panax japonicus C. A. Meyer., Biol. Pharm. Bull, (2008), 31, p135-138.
[https://doi.org/10.1248/bpb.31.135]

- J.Y. Hong, S.R. Shin, M.J. Bae, J.S. Bae, I.C. Lee, O.J. Kwon, J.W. Jung, Y.H. Kim, T.H. Kim, Pancreatic lipase inhibitors isolated from the leaves of cultivated mountain ginseng(Panax ginseng)., Korean Journal of Food Preservation, (2010), 17, p727-732.
- D.Y. Hyun, G.S. Hyeon, B.Y. Yeon, S.W. Kang, Y.C. Kim, K.W. Lee, S.M. Kim, A guideline to land suitability used soil physical characteristics and yield potential in Panax ginseng C. A. Meyer., Hanguk Yakyong Changmul Hakhoe Chi, (2009), a 17, p421-426.
- D.Y. Hyun, B.Y. Yeon, S.W. Lee, S.W. Kang, G.S. Hyeon, Y.C. Kim, K.W. Lee, S.M. Kim, Analysis of occurrence type of physiological disorder to soil chemical components in ginseng cultivated field., Hanguk Yakyong Changmul Hakhoe Chi, (2009), b 17, p439-444.
-
I.H. Jo, K.H. Bang, Y.C. Kim, J.U. Kim, M.R. Shin, J.Y. Moon, B.S. Noh, D.Y. Hyun, D.H. Kim, S.W. Cha, H.S. Kim, Analysis of mitochondrial DNA sequence and molecular marker development for identification of Panax species., Hanguk Yakyong Changmul Hakhoe Chi, (2013), 21, p91-96.
[https://doi.org/10.7783/kjmcs.2013.21.2.91]

- J. Kim, B.H. Jo, K.L. Lee, E.S. Yoon, G.H. Ryu, K.W. Chung, Identification of new microsatellite markers in Panax ginseng., Mol. Cells, (2007), 24, p60-68.
- S. Kim, J.H. Jeong, H. Chung, J.H. Kim, J. Gil, J. Yoo, Y. Um, O.T. Kim, T.D. Kim, Y.Y. Kim, D.H. Lee, H.B. Kim, Y. Lee, Simple sequence repeat marker development from Codonopsis lanceolata and genetic relation analysis., J. Plant Biotechnol, (2016), 43, p181-188.
- S.T. Kim, J.H. Jang, J.H. Kwon, K.D. Moon, Changes in the chemical components of red and white ginseng after puffing., Korean Journal of Food Preservation, (2009), 16, p355-361.
- X. Li, S.J. Kang, J.S. Han, J.S. Kim, J.E. Choi, Comparison of growth increment and ginsenoside content in different parts of ginseng cultivated by direct seeding and transplanting., Hanguk Yakyong Changmul Hakhoe Chi, (2010), 18, p70-73.
-
Y.C. Li, A.B. Korol, T. Fahima, A. Beiles, E. Nevo, Microsatellites: Genomic distribution, putative functions and mutational mechanisms: A review., Mol. Ecol, (2002), 11, p2453-2456.
[https://doi.org/10.1046/j.1365-294x.2002.01643.x]

- J.R. Lim, J.Y. Kim, C.B. Park, B.K. Choo, D.H. Kim, Y.G. Choi, C.Y. Hwang, Growth of Panax ginseng C. A. Meyer underwood in direct sowing and transplanting., Hanguk Yakyong Changmul Hakhoe Chi, (2003), 11, p216-223.
-
K. Liu, S.V. Muse, Powermarker: An integrated analysis environment for genetic marker analysis., Bioinformatics, (2005), 21, p2128-2129.
[https://doi.org/10.1093/bioinformatics/bti282]

- S.S. Shin, K.C. Kim, Y.H. Choi, Y.T. Lee, H.S. Eom, C.S. Kim, Critic standardization and objectivity of mountain grown ginseng., Journal of Dong-Eui Oriental Medicine, (2001), 5, p107-114.
-
Y. Um, M.L. Jin, O.T. Kim, Y.C. Kim, S.C. Kim, S.W. Cha, K.W. Chung, S. Kim, C.M. Chung, Y. Lee, Identification of Korean ginseng(Panax ginseng) cultivars using simple sequence repeat markers., Plant Breed. Biotechnol, (2016), 4, p71-78.
[https://doi.org/10.9787/pbb.2016.4.1.71]