침엽수림과 혼효림에서 토양특성과 토양세균 군집이 산양삼 초기 생육특성에 미치는 영향
This is an open access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
This study investigated the effect of soil properties and soil bacterial community on early growth characteristics of wild-simulated ginseng (Panax ginseng C. A. Meyer) in coniferous and mixed forest experimental fields.
The soil bacterial community was analyzed using a high throughput sequencing technique (Illumina MiSeq sequencing). The relationship between the soil bacterial community, soil properties, and growth characteristics of wild-simulated ginseng were analyzed using principal coordinate analysis (PCoA) and the Pearson’s correlation analysis. Soil properties and soil bacterial community showed significant difference with forest physiognomy. Results of Pearson’s correlation analysis and PCoA showed that the soil properties (soil pH, organic matter, total nitrogen, and cation exchange capacity) and soil bacterial community had significant correlation with tree species ratio and early growth characteristics of wild-simulated ginseng.
This study clearly demonstrated the effect of soil properties and soil bacterial community on early growth characteristics of wild-simulated ginseng in coniferous and mixed forest. Moreover, these results will help in the selection of suitable cultivation sites for wild-simulated ginseng.
Keywords:
Panax ginseng C. A. Meyer, Wild-simulated Ginseng, Soil Properties, Soil Bacterial Community, Coniferous and Mixed Forest서 언
식물 뿌리의 영향을 받는 근권 (rhizosphere)은 식물과 미생물이 상호작용을 이루는 공간으로 (Philippot et al., 2013; Lee et al., 2015), 근권에서 서식하는 토양미생물은 양분의 순환, 유기물의 분해, 오염물질의 제거, 식물 양분 공급 등 토양의 질과 생산성을 결정하는데 중요한 역할을 한다 (Shade and Handelsman, 2012; Prasad et al., 2015). 최근에는 토양미생물 군집을 분석하는 방법으로 대표적인 비배양적 방법 (culture-independent methods)인 차세대 염기서열 분석 (next generation sequencing; NGS)을 주로 이용하고 있으며, 토양미생물 군집 구조의 분석 외에도 토양특성과 같은 환경요인과의 상관관계를 구명하는 연구에도 이용하고 있다 (Song et al., 2018; Wu et al., 2018).
산양삼 (Panax ginseng C. A. Meyer)은 두릅나무과 (Araliaceae)에 속하는 인삼속 (genus Panax) 식물로서 학명은 Panax ginseng C. A. Meyer로 표기한다. [산양삼에 관한 품질관리 요령]에서 산지관리법 제 2 조 제 1 호에 의거해 산양삼이란 ‘산지에서 차광막 등 인공시설을 설치하지 아니하고 생산되는 삼’을 산양삼으로 정의하여 [임업 및 산촌진흥 촉진에 관한 법률]에 의거 ‘특별관리 임산물’로 지정하여 관리하고 있다. 산양삼은 무농약, 무비료를 원칙으로 산지에서 7 년 이상 20 년까지 장기적으로 재배하기 때문에 재배지의 환경요인, 그 중에서도 임상 (forest physiognomy)과 토양특성은 매우 중요하다고 할 수 있다 (Kim et al., 2019a).
산림청 조사에 의하면 우리나라는 국토 면적의 63.8%가 산림으로 구성되어 있고 (KFS, 2016), 침엽수림 (coniferous forest) 43.5%, 혼효림 (mixed forest) 30.5%, 활엽수림 (broad-leaved forest) 26.0% 순으로 구성되어 있다 (Jeon et al., 2013). 산림환경에서 식생은 서로의 상호작용으로 형성되는데 토양특성은 산지 내 수종 등에 영향을 받아 유의적으로 변화한다고 하였다 (Chung and Moon, 2011). Pettersson (2004)은 토양특성과 같은 환경요인은 ‘조절자 (modulator)’로 불리며, 이러한 환경요인의 변화는 식물과 토양미생물에게 결과적으로 스트레스로 작용하여 식물의 생산성과 토양미생물의 군집에 영향을 주기 때문에 토양미생물을 연구하는데 있어 환경요인과의 상관관계를 구명하는 연구는 필수적이라고 하였다.
이처럼 산지에서 인공시설 없이 자연 상태 그대로 재배하는 산양삼은 노지에서 재배하는 인삼 (재배삼)에 비해 임상, 토양특성, 토양미생물과 같은 재배환경이 매우 중요하고, 이를 통해 재배적지를 선정하는 과학적이고 체계적인 정립이 필요하다. 이전부터 국내외 다양한 산림환경에 따른 토양특성 연구 (Russell et al., 2007; Yun and Moon, 2009), 산림토양과 임상별 토양미생물 군집 연구 (Lee and Shim, 1994; Nacke et al., 2011)가 수행되었고, 인삼 및 산양삼 재배지의 토양미생물 군집 분석 연구도 최근까지 활발히 수행되고 있다 (Nguyen et al., 2016; Dong et al., 2018; Kim et al., 2019b). 그러나 산양삼 재배지의 임상별 토양특성과 토양미생물 군집 연구와 이에 따른 산양삼 생육특성 간의 상관관계 연구는 매우 미흡한 실정이다.
따라서 본 연구는 우리나라 임상 중 가장 비중이 높은 침엽수림과 혼효림을 선정하고 토양특성과 토양세균 군집을 분석하여 산양삼 생육특성에 미치는 영향을 구명하고자 수행하였다.
재료 및 방법
1. 산양삼 시험포지 선정 및 조성
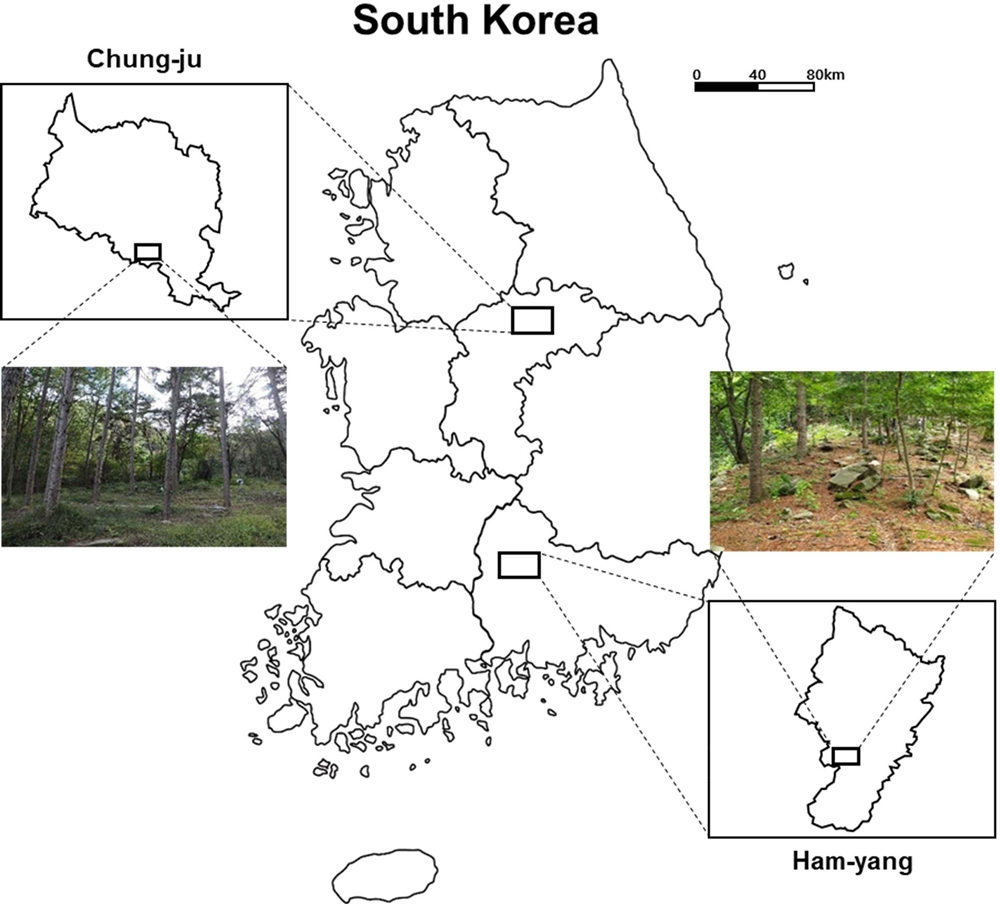
본 연구에서는 침엽수림과 혼효림의 산양삼 (Panax ginseng C. A. Meyer) 시험포지를 조성하기 위해 한국임업진흥원에서 관리·운영하는 종자공급단지 중 침엽수림으로 구성된 충주 종자공급단지와 혼효림으로 구성된 함양 종자공급단지를 선정하였다 (Fig. 1). 산양삼 시험포지는 2019년 3월에 충주와 함양 종자공급단지 내 0.1 ha 씩 조성하였다.
2. 산양삼 시험포지의 토양시료 채취 및 입지환경 조사
본 연구에서 사용한 공시토양은 충주와 함양 종자공급단지 내 조성한 산양삼 시험포지 0.1 ㏊ 내에서 임의의 3 구역을 선정한 후 표토를 제거하고, 20 ㎝ 깊이 이내의 근권 토양을 채취하였다. 채취한 토양시료는 각 지역명 약자에 따라 CJ1, CJ2, CJ3, HY1, HY2, HY3로 표기하였다. 토양시료 중 토양세균 군집 분석에 사용할 토양은 아이스박스에 보관하여 이동 후 -20℃에서 냉동보관 하였고, 이화학성 분석에 사용할 토양은 2 ㎜ 체를 이용하여 거른 후 실온에서 풍건하였다.
풍건한 토양시료는 농촌진흥청 종합검정실 분석 매뉴얼 (RDA, 2013)에 따라 토양 pH, 전기전도도 (electric conductivity, EC), 유기물 (organic matter, OM), 전질소 (total nitrogen, TN), 유효인산 (available phosphate, Avail. P2O5), 치환성 양이온 (exchangeable cation), 양이온치환용량 (cation exchange capacity, CEC)을 측정하였다. 충주와 함양 종자공급단지 내 조성한 각 시험포지에서 10 × 10 m 구획을 선정하여, 임상의 수종, 수고, 흉고직경, 지형, 사면방향, 사면경사, 해발고를 조사하였다.
3. 산양삼 종자 출아율, 종묘 활착률, 생육특성 조사
임상별 시험처리구의 산양삼 종자 출아율, 종묘 활착률, 생육특성 조사를 위해 [산양삼 표준재배지침]을 참고하여 산양삼 종자 직파와 종묘 이식 처리구를 각각 조성하였다 (Jeon et al., 2018).
산양삼 종자의 직파 처리구는 종자를 반복수당 각 90 립씩 파종하였고, 이식 처리구는 2년생 산양삼 종묘를 반복수당 각 100 본 씩 식재하였다. 모든 처리구는 3 반복으로 수행하였다. 파종 및 이식한지 50 일 이후부터 산양삼 종자의 출아개수와 종묘 활착개수를 조사하였고, 이식한 지 120 일 이후에 종묘를 채취하여 국립종자원의 [인삼 작물별 특성조사요령]에 따라 생육특성 (줄기길이, 줄기직경, 지상부 생중량, 지하부 길이, 주근직경, 지하부 생중량, 지하부 건중량)을 조사하였다.
4. 토양세균 군집 분석
산양삼 시험포지의 토양세균 군집을 분석하기 위해 토양 시료의 total DNA는 DNeasy PowerSoil kit (QIAGEN, Hilden, Germany)를 이용하여 추출하였고, 16S rRNA Polymerase chain reaction (PCR) 증폭 및 염기서열 분석은 마크로젠 (Macrogen Inc., Seoul, Korea)의 Illumina MiSeqTM sequencing system (Illumina Inc., San Diego, CA, USA)에 따라 수행하였다.
토양세균 군집의 raw sequence data에 대한 분석은 Mothur pipeline (version 1.43.0, The University of Michigan, USA)을 이용하여 분석하였고, 염기서열에 대한 데이터베이스는 ‘Greengene reference database’를 이용하였으며, 97% 유사수준의 Operational taxonomic units (OTUs)으로 군집화하여 계통수 (phylogenetic tree) 및 상대적 빈도수(relative abundance)를 확인하였다 (Schloss, 2009; Schloss et al., 2009; Kim et al., 2019b).
토양 이화학성, 임상 (평균 흉고직경, 평균 수고, 침엽수 흉고직경, 침엽수 수고, 침엽수 비율, 활엽수 흉고직경, 활엽수 수고, 활엽수 비율), 토양세균 군집 간의 상관관계는 주좌표 분석 (principal coordinate analysis, PCoA)에서 Bray-Curtis similarity를 이용하여 토양세균 군집의 염기서열 간의 유사성을 ordination하여 나타냈고, 상대적 빈도수와 토양이화학성은 각 축에 대한 상관계수를 통해 표시하였다.
5. 통계분석
본 연구에서 도출된 정량적 실험데이터는 평균 ± 표준오차 (means ± standard error, S.E.)으로 나타냈고, 유의성 검정은 Statistical Analysis System (SAS, version 7.1, SAS Institute, Cary, NC, USA) software를 이용하여 토양세균 군집 cluster 간의 상대적 빈도수와 토양이화학성을 T-검정을 통해 유의성을 검정하고, 최소유의차 (Least Significant Difference, LSD)는 p < 0.05 수준에서 통계처리 하였다. 토양세균 군집, 토양이화학성, 수종 비율, 생육특성 간의 상관관계 분석 (Correlation Analysis)은 IBM SPSS Statistics (version 25, IBM Corp., Armonk, New York, USA)를 사용하여 Pearson 상관계수 (Pearson’s correlation coefficient, r)와 유의성 (p < 0.05)을 확인하였다.
결과 및 고찰
1. 임상별 산양삼 시험포지의 입지환경
충주와 함양 종자공급단지 내 조성한 시험포지의 지형과 임상에 대한 결과는 Table 1에 나타냈다.
충주 시험포지는 남동사면에 기울기가 13°, 해발고는 317 m이었고, 함양 시험포지는 북동사면에 기울기가 14°, 해발고는 669 m로 조사되었다. 임상 조사 결과, 충주 시험포지는 일본잎갈나무 (낙엽송, Japanese larch)가 100%로 조성된 침엽 수림이었고, 함양 시험포지는 활엽수인 물푸레나무와 비목나무가 각각 23.3%와 13.3%로 우점종이었으며, 이 외에 개옻나무, 생강나무, 쪽동백나무의 활엽수와 소나무, 잣나무의 침엽수로 조성된 혼효림이었다.
우리나라 산양삼 (Panax ginseng C. A. Meyer) 재배지 임간에 분포하는 수종의 비율은 활엽수가 전체의 67%이었고, 침엽수가 22%로 활엽수가 더 많은 비중을 차지하는 것으로 알려져 있다 (Kwon et al., 2011). 충주 시험포지 침엽수림의 평균 흉고직경은 30.8 ㎝이었고, 평균 수고는 21.5 m이었다. 함양 시험포지에서 활엽수의 평균 흉고직경은 6.9 ㎝, 평균 수고는 7.7 m이었고, 침엽수의 평균 흉고직경은 27.2 ㎝, 평균 수고는 30.2 m이었다. 우리나라에서는 강원도, 경상도, 전라도 등 다양한 지역에서 산양삼을 재배하고 있는데, 사면방향은 북사, 북동, 남동, 남서 등 다양하였고, 사면경사는 5° - 29°, 임상의 평균 수고는 16.7 m, 평균 흉고직경은 21.5 ㎝ 인 것으로 알려져 있다 (KOFPI, 2013; Kim et al., 2019b). 또한 산양삼 재배에는 평지보다 사면지형이 좋고, 방향은 북향 또는 북동향, 경사는 5° - 15°, 주변 임상의 수고는 7 m 이상이 적합하다고 하였다 (Lee, 2010).
임상별 토양특성 분석 결과에서 두 시험포지 모두 토성은 사양토였고, pH 4.89 - 4.97의 산성토양으로 확인되었다 (Table 2). 시험포지 간의 토양 이화학성을 비교 분석한 결과, 토양 유기물 (p ≤ 0.0021), 전질소 (p ≤ 0.0045), 양이온치환용량 (p ≤ 0.0090)이 혼효림인 함양 시험포지에서 침엽수림인 충주 시험포지에 비해 유의적으로 높았다. 우리나라 산양삼 재배지의 토성은 양질사토 (loamy sand), 사양토 (sandy loam), 양토 (loamy soil)로 배수가 양호하고, pH는 4.0 - 6.0으로 산성이나 약산성 토양인 것으로 알려져 있다 (Lee, 2010).
토양의 유기물, 전질소, 양이온치환용량은 식물의 생장과 매우 밀접한 관계가 있다. 토양 유기물은 식물에게 영양원을 공급할 뿐만 아니라 토양 수분함량을 증가시키고 토양 물리성을 개선하며 (Kozlowski and Pallardy, 1999; Son and Cho, 2009), 양이온치환용량은 토양의 비옥도를 나타내는 지표로서 토양 완충능 향상, 양분의 보유 및 공급 등에 관여한다 (Lim, 2005). 활엽수의 낙엽은 산림토양에 유기물을 공급하여 침엽수로 조성된 토양에 비해 유기물 및 전질소의 함량이 높다고 하였다 (Kim et al., 2015; Liu et al., 2016).
이와 같은 선행연구들은 본 연구에서 조성한 산양삼 시험포지의 임상과 토양이 산양삼 재배에 적합하다는 것을 뒷받침하며, 침엽수림인 충주 시험포지에 비해 혼효림인 함양 시험포지가 토양 이화학성 측면에서 산양삼 재배에 보다 적합한 것으로 판단된다.
2. 산양삼 종자 출아율, 종묘 활착률, 생육특성
임상별 시험포지에서 산양삼 종자의 출아율과 종묘 활착률을 조사한 결과, 종자 출아율은 임상별 시험포지 간의 유의적인 차이가 없었으나 종묘 활착률 (p ≤ 0.0080)은 침엽수림인 충주 시험포지에 비해 혼효림인 함양 시험포지에서 유의적으로 높았다 (Table 3). 산양삼 종묘를 이식한지 120 일 이후에 종묘 샘플을 채취하여 생육특성을 조사한 결과, 지상부에서는 줄기직경 (p ≤ 0.0026)과 지상부 생중량 (p ≤ 0.0024)이 혼효림인 함양 시험포지에서 침엽수림인 충주 시험포지보다 유의적으로 높았고, 지하부에서는 주근직경 (p ≤ 0.0170)과 건중량 (p ≤ 0.0331)이 함양 시험포지에서 유의적으로 높았다. 이러한 결과는 침엽수림보다 혼효림이 산양삼 재배에 더 적합하다는 것을 의미한다.
Chamberlain 등 (2013)은 산림의 수종과 산양삼의 생산량은 유의적인 상관관계를 가진다고 하였다. 산양삼은 음지식물로 상층 울폐도의 영향을 많이 받으며, 활엽수의 잎은 상층 울폐도를 높여주는 역할을 하게 되는데 (Woo and Lee, 2002), 본 연구결과와 마찬가지로 침엽수림보다 활엽수림 및 혼효림에서 산양삼의 생육이 증가한다고 보고되었다 (Suh et al., 2011).
3. 토양세균 군집의 상대적 빈도수
침엽수림과 혼효림에 조성한 산양삼 시험포지의 토양세균 군집은 임상별로 뚜렷하게 군집화를 이루는 것을 확인하였다 (Fig. 2). 임상별 시험포지의 토양세균은 두 시험포지 모두 Acidobacteria가 우점종으로 확인되었다.
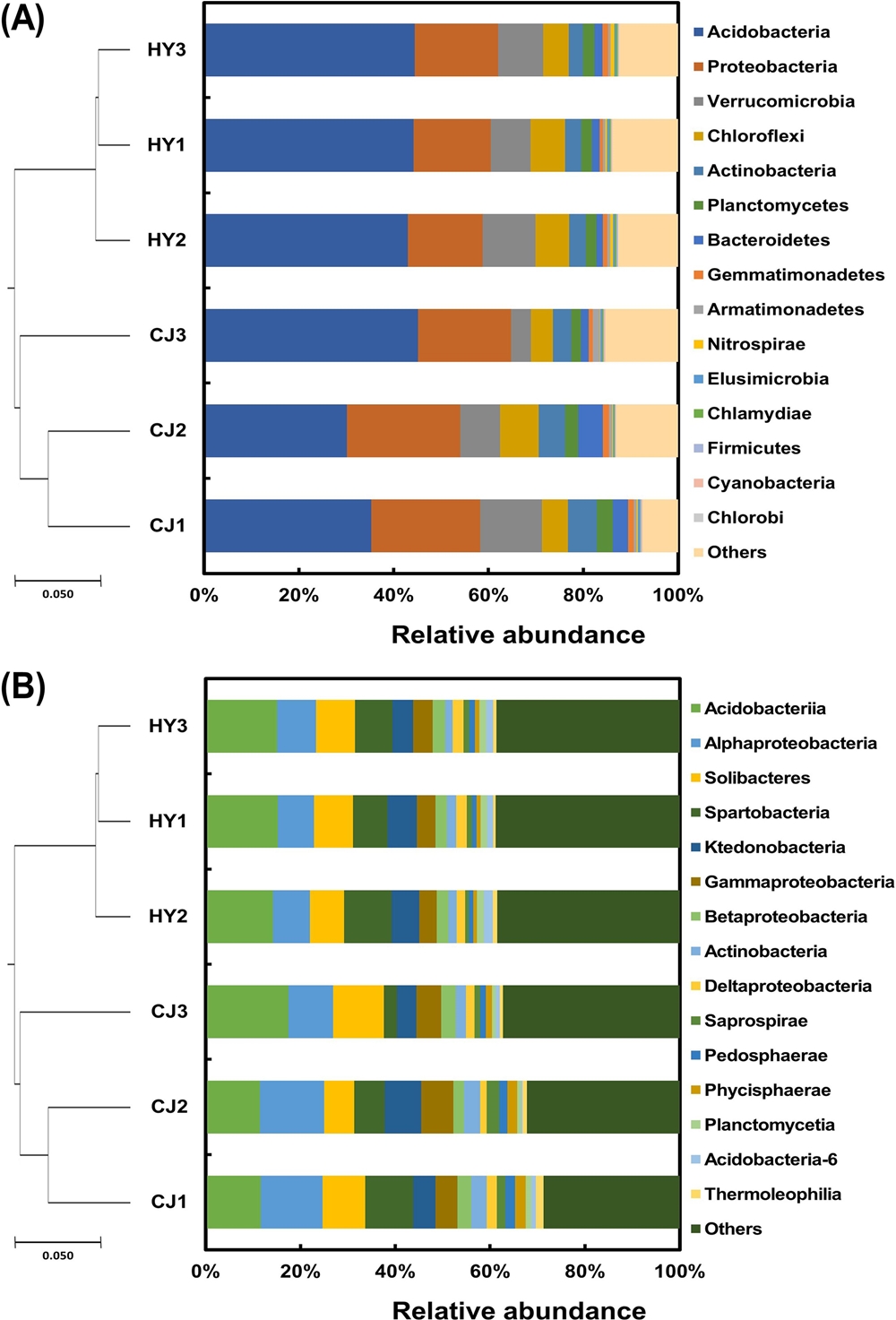
Clustering and relative abundance of bacterial community in two different wild-simulated ginseng experimental fields.Relative abundance of the phylum and top 15 classes in coniferous (CJ) and mixed (HY) forest experimental fields using phylotype-based analysis of taxonomy. Others indicate the sum of the relative abundance of all classes except the 15 classes on the figure. (A); phylum, (B); class. CJ; Chung-ju, HY; Ham-yang.
토양세균 군집의 상대적 빈도수를 임상별로 phylum과 class 수준에서 확인한 결과, 먼저 충주 시험포지의 phylum 수준에서는 Acidobacteria가 전체 토양세균 군집의 36.8%로 상대적 빈도수가 가장 높았고, 그 다음으로 Proteobacteria (22.2%), Verrucomicrobia (8.6%), Chloroflexi (6.1%), Actinobacteria (5.1%) 순으로 높게 나타났다. 함양 시험포지의 phylum 수준도 충주 시험포지와 마찬가지로 Acidobacteria가 43.9%로 가장 높았고, Proteobacteria (16.5%), Verrucomicrobia (9.7%), Chloroflexi (6.6%), Actinobacteria (3.3%) 순으로 높게 나타났다.
Classes 수준에서 확인한 결과, 충주 시험포지는 Acidobacteriia가 13.2%로 상대적 빈도수가 가장 높았고, Alphaproteobacteria (11.9%), Solibacteres (8.7%), Spartobacteria (6.4%), Ktedonobacteria (5.5%) 순으로 상대적 빈도수가 높은 것을 확인할 수 있었다. 함양 시험포지의 경우, Acidobacteriia가 14.8%로 상대적 빈도수가 가장 높았고, Spartobacteria (8.4%), Alphaproteobacteria (7.9%), Solibacteres (7.8%), Ktedonobacteria (3.9%) 순으로 상대적 빈도수가 높은 것을 확인할 수 있었다.
토양 세균 중 Acidobacteria는 주로 pH가 낮은 산성 토양에서 서식하는 호산성 세균 (acidophilic bacteria)이다 (Park et al., 2016). 산양삼은 산성이나 약산성 토양에서 생육이 우수하다고 알려져 있으며 (Kim et al., 2019a), Panax ginseng을 재배하는 토양에서 우점하거나 상대적 빈도수가 높다고 보고되고 있다 (Li et al., 2014b; Sun et al., 2017; Wang et al., 2019). Kim 등 (2019b)의 연구에서도 우리나라 산양삼 재배지에서 Acidobacteria가 토양 pH와 유의적인 부의 상관관계를 보였고, 산양삼의 생육과 유의적인 관계를 가진다고 보고하였다.
4. 임상과 토양세균 군집 간의 상관관계
주좌표 분석을 통해 임상별 산양삼 시험포지에서 우점하는 토양세균 군집을 확인한 결과, 먼저 토양세균 군집은 임상별 시험포지에 따라 군집화를 이루는 것으로 확인되었고, 침엽수림인 충주 시험포지에서는 Proteobacteria, Actinobacteria, Bacteroidetes, Plantomycetes, Ahlpaproteobacteria, Actinobacteria_class, Phycisphaerae, Saprospirae가 우점하였으며, 혼효림인 함양 시험포지에서는 Acidobacteria, Chlamydiae, Chlorobi, Nitrospirae, Acidobacteriia, Deltaproteobacteria, Acidobacteria_6, Plantomycetia가 우점하는 것으로 확인되었다 (Fig. 3). 이 중에서 Proteobacteria, Ahlpaproteobacteria, Actinobacteria class는 유의적으로 침엽수림인 충주 시험포지에서 상대적 빈도수가 높았고, 혼효림인 함양 시험포지에서는 Nitrospirae의 상대적 빈도수가 유의적으로 높은 것으로 확인되었다.
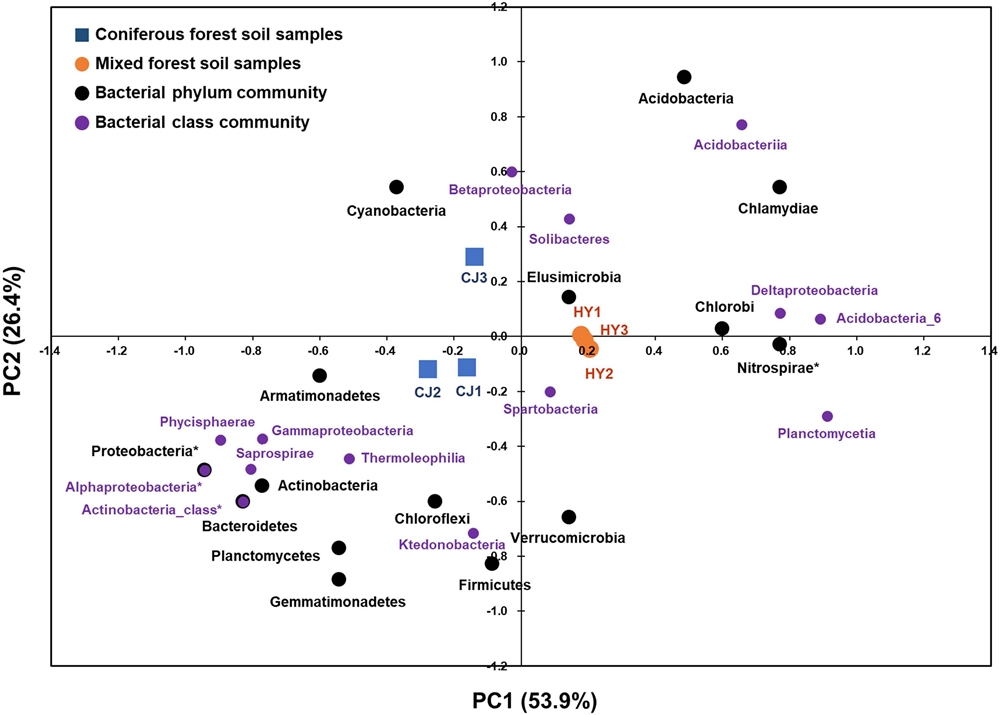
The ordination plot shown the phylum an class community of soil samples according to their bacterial community composition obtained from principal coordinate (PCoA) analysis based on Bray-Curtis dissimilarity matrix generated using Mothur platform.CJ; Chung-ju, HY; Ham-yang.
Pearson 상관관계 분석 결과, 토양세균 군집의 상대적 빈도수는 수종 비율에 따라 상이한 상관관계를 보이는 것으로 확인되었다 (Table 4). 이 중에서 Proteobacteria, Alphaproteobacteria, Actinobacteria_class, Phycisphaerae는 침엽수의 비율과 유의적인 정의 상관관계를 보였고, Nitrospirae, Chlamydiae, Planctomycetia, Acidobacteria_6는 활엽수의 비율과 유의적인 정의 상관관계를 보이는 것으로 나타났다.
Pearson’s correlation analysis between tree species ratio and soil bacterial community in two different experimental fields of wild-simulated ginseng.
산림토양에서 토양세균 군집의 다양성은 침엽수림에 비해 활엽수림에서 높다고 알려져 있다 (Xia et al., 2016). Han 등 (2008)은 침엽수림과 활엽수림의 부식토 내 토양세균 군집구조가 매우 특정적인 계통학적 특성을 가지고 있다고 보고하였고, Li 등 (2014a)은 중국 산림유형에 따른 토양세균 군집을 분석하여 침엽수림에서는 Proteobacteria가 우점하였고, 혼효림에서는 Bacteroidetes와 Nitrospira가 우점종이라고 보고하였다. 이러한 선행연구는 본 연구결과에서 Proteobacteria와 Nitrospira가 각각 침엽수림과 혼효림에서 우점종으로 확인된 결과를 뒷받침 한다. 산림토양에 서식하는 수종과 세균군집 구성은 밀접한 관계가 있고 (Tsai et al., 2009), 식생의 종류에 따라 토양미생물 군집이 형성된다고 알려져 있다 (Hackl et al., 2005). Han 등 (2006)은 특히 수목의 종류 (수종)에 따라 침엽수림과 활엽수림의 토양이화학성과 토양세균 군집이 다양하게 형성된다고 보고하여, 임상별 토양이화학성이 토양세균의 군집에 영향을 주는 것으로 판단된다.
5. 토양이화학성과 토양세균 군집 간의 상관관계
임상별 산양삼 시험포지의 토양이화학성이 토양세균 군집의 군집화에 미치는 영향을 주좌표 분석과 Pearson 상관관계로 분석하였다. 주좌표 분석에서 각 축의 수치는 토양세균 군집의 변화량 (variation)을 의미하며, 각 축의 상관계수를 통해 표시된 토양이화학성은 종 (ordinate) 또는 횡 (abscissa)으로 나누는 토양세균 군집과의 연관성을 가진다 (Kim et al., 2020).
본 연구에서 PC1의 변화량은 53.9%로 PC2의 26.4% 보다 높은 것으로 확인되었다 (Fig. 4). PC1의 변화량이 PC2 보다 높다는 것은 종좌표에 위치한 토양이화학성이 횡좌표에 위치한 토양이화학성 보다 토양세균 군집에 영향을 준다는 것을 의미한다고 할 수 있다. 따라서 토양이화학성 중에서는 전질소, 양이온치환용량, 유기물, 전기전도도, 나트륨 함량이 다른 이화학성에 비해 토양세균 군집에서 영향을 주었다.
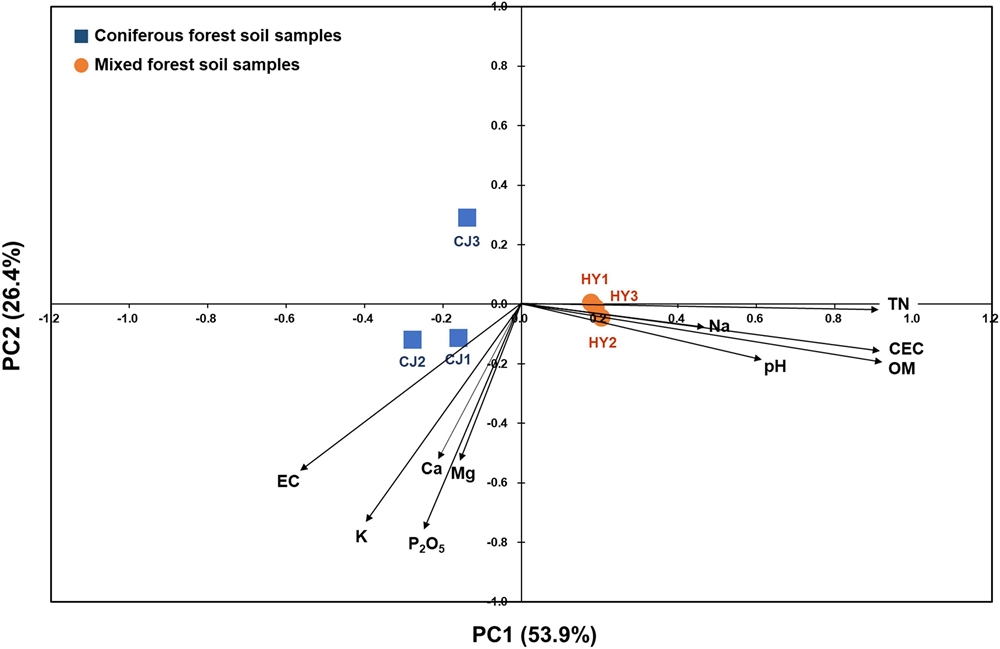
The ordination plot shown the soil samples and soil parameters according to their bacterial community composition obtained from PCoA analysis based on Bray-Curtis dissimilarity matrix.The correlation between soil parameters and two axes are represented by the length and angle of the arrows. CJ; Chung-ju, HY; Ham-yang.
토양이화학성이 토양세균 군집의 미치는 영향을 유의적으로 검정하기 위해 토양세균 군집 중에서 임상별로 우점하는 토양세균 군집의 상대적 빈도수와 토양이화학성 간의 상관관계를 분석하였다. Pearson 상관관계를 통한 분석 결과, 토양 pH, 유기물, 전질소, 양이온치환용량이 토양세균 군집과 유의적인 상관관계를 보였다 (Table 5).
토양세균 중 Nitrospirae (r = 0.848, p = 0.033)와 Planctomycetia (r = 0.893, p = 0.016)는 토양 pH와 유의적인 정의 상관관계를 보였고, Nitrospirae (r = 0.847, p = 0.033), Chlamydiae (r = 0.882, p = 0.020), Planctomycetia (r = 0.908, p = 0.012), Acidobacteria_6 (r = 0.855, p = 0.030)은 토양 유기물과 유의적인 정의 상관관계를 보였다. 전질소 함량의 경우는 Chlamydiae (r = 0.941, p = 0.005), Planctomycetia (r = 0.839, p = 0.037)와 Acidobacteria_6 (r = 0.862, p = 0.027)은 유의적인 정의 상관관계를 보였고, 양이온치환용량은 Nitrospirae (r = 0.823, p = 0.044), Chlamydiae (r = 0.840, p = 0.037), Planctomycetia (r = 0.951, p = 0.004), Acidobacteria_6 (r = 0.909, p = 0.012)와 유의적인 정의 상관관계를 보였다. 토양미생물 군집과 토양 이화학성 간의 상관관계는 이전부터 많은 연구에서 수행되어 왔다 (Mechri et al., 2008; Preem et al., 2012).
본 연구에서는 토양 pH, 유기물, 전질소, 양이온치환용량이 토양세균 군집과 유의적인 상관관계를 가지는 것으로 확인되었다. 토양 pH는 토양미생물 군집에 영향을 주는 대표적인 토양요인으로 Han 등 (2018)과 Xia 등 (2016)은 산림토양에서 토양 pH가 토양세균 군집에 유의적으로 영향을 준다고 보고하였고, Kim 등 (2019b)의 연구에서도 본 연구와 마찬가지로 토양 pH와 Acidobacteria속 토양세균 군집이 유의적인 상관관계를 가진다고 보고하였다.
토양에 서식하는 토양미생물은 유기물 분해, 양분순환 및 토양의 입단화에 중요한 역할을 한다 (An et al., 2015; Han, 2015). 식물의 생육은 토양 유기물, 전질소, 양이온치환용량과 밀접한 관계가 있는 만큼 본 연구결과와 같이 유기물, 전질소, 양이온치환용량과 유의적인 상관관계를 가지는 토양세균 군집은 산양삼의 생육특성과도 유의적인 상관관계를 가질 것으로 판단된다.
6. 토양세균 군집과 산양삼 생육특성 간의 상관관계
임상별 산양삼 시험포지의 토양세균 군집과 산양삼 생육특성 간의 상관관계를 분석한 결과, Nitrospirae, Chlorobi, Planctomycetia, Acidobacteria_6가 산양삼의 생육과 유의적인 정의 상관관계를 보였다 (Table 6).

Pearson’s correlation analysis between soil bacterial community and growth characteristics of wild-simulated ginseng.
Nitrospirae는 종자 출아율 (r = 0.872, p = 0.023), 뿌리길이 (r = 0.847, p = 0.033), 주근직경 (r = 0.867, p = 0.025), 지하부 건중량 (r = 0.850, p = 0.032)과 유의적인 정의 상관관계를 보였고, Chlorobi는 종묘 활착률 (r = 0.920, p = 0.009), 줄기길이 (r = 0.823, p = 0.044), 줄기직경 (r = 0.910, p = 0.012), 지상부 생중량 (r = 0.837, p = 0.038)과 유의적인 정의 상관관계를 보였다. Planctomycetia는 줄기직경 (r = 0.900, p = 0.014), 뿌리길이 (r = 0.927, p = 0.008), 주근직경 (r = 0.909, p = 0.012)과 유의적인 정의 상관관계를 보였고, Acidobacteria_6은 줄기직경 (r = 0.856, p = 0.030), 뿌리길이 (r = 0.817, p = 0.047)와 유의적인 정의 상관관계를 보였다.
인삼 재배지의 토양미생물 군집 구조에 대한 연구는 이전부터 활발히 수행되고 있다. Suleiman 등 (2013)은 Acidobacteriaceae, Nitrospiraceae, Planctomycetaceae가 산림토양에서 우점하는 미생물 군집 중 하나라고 하였고, Nguyen 등 (2016)은 인삼 재배 토양의 토양미생물 군집을 확인한 결과, Nitrospirae와 Chlorobi을 포함하는 토양세균 군집이 확인하였다고 보고하였다. 최근에는 Kim 등 (2019b)이 산양삼의 생육특성과 토양세균 군집 간의 상관관계에 대한 연구를 수행하여 Acidobacteria속 계열인 Acidobacteriia와 Koribacteraceae가 산양삼의 뿌리길이, 주근직경, 지하부의 단면적과 표면적, 지하부 부피와 유의적인 정의 상관관계를 보인다고 보고하였다. 이와 같은 선행연구는 Nitrospirae, Chlorobi, Planctomycetia, Acidobacteria_6 계통의 토양세균이 시험포지에서 산양삼 생육특성과 유의적인 상관관계를 보인다는 연구 결과를 뒷받침 한다.
본 연구결과를 바탕으로 다양한 산림환경에서 토양특성, 토양세균 군집, 산양삼 생육특성 간의 상관관계를 명확하게 구명할 수 있다면 향후 산양삼의 최적 재배지를 선정하는데 있어 도움을 줄 수 있을 것으로 사료된다.
Acknowledgments
본 연구는 국립산림과학원 일반연구사업(과제번호: FP0802-2017-01)의 지원에 의해 이루어진 결과로 이에 감사드립니다.
REFERENCES
-
An NH, Ok JH, Cho JL, Shin JH, Nam HS and Kim SC. (2015). Effects of organic matter application on soil microbial community in a newly reclaimed soil. Korean Journal of Organic Agriculture. 23:767-779.
[https://doi.org/10.11625/KJOA.2015.23.4.767]

-
Chamberlain JL, Prisley S and Mcguffin M. (2013). Understanding the relationship between american ginseng harvest and hardwood forests inventory and timber harvest to improve co-management of the forest of Eastern United States. Journal of Sustainable Forest. 32:605-624.
[https://doi.org/10.1080/10549811.2013.798828]

- Chung JM and Moon HS. (2011). Soil characteristics by the site types around Nari Basin in Ulleung island. Journal of Agricultural and Life Science. 44:45-50.
-
Dong L, Xu J, Li Y, Fang H, Niu W, Li X, Zhang Y, Ding W and Chen S. (2018). Manipulation of microbial community in the rhizosphere alleviates the replanting issues in Panax ginseng. Soil Biology and Biochemistry. 125:64-74.
[https://doi.org/10.1016/j.soilbio.2018.06.028]

-
Hackl E, Pfeffer M, Donat C, Bachmann G and Zechmeister Boltenstern S. (2005). Composition of the microbial communities in the mineral soil under different types of natural forest. Soil Biology and Biochemistry. 37:661-671.
[https://doi.org/10.1016/j.soilbio.2004.08.023]

-
Han D, Wang N, Sun X, Hu Y and Feng F. (2018). Biogeographical distribution of bacterial communities in Changbai Mountain, Northeast China. Microbiology Open. 7:e529. (cited by 2020 March 31).
[https://doi.org/10.1002/mbo3.529]

- Han SI, Cho MH and Whang KS. (2008). Comparison of phylogenetic characteristics of bacterial populations in a quercus and pine humus forest soil. Korean Journal of Microbiology. 44:237-243.
- Han SI, Kim YJ and Whang KS. (2006). Comparison of phylogenetic characteristics of viable but non-culturable(VBNC) bacterial populations in the pine and quercus forest soil by 16S rDNA-ARDRA. Korean Journal of Microbiology. 42:116-124.
-
Han SI. (2015). Phylogenetic characterization of bacterial populations in different layers of oak forest soil. Korean Journal of Microbiology. 51: 133-140.
[https://doi.org/10.7845/kjm.2015.5017]

- Jeon KS, Um YR, Jung CR, Park HW and Kim MJ. (2018). Standard cultivation manual of wild-simulated ginseng. National Institute of Forest Science, Seoul, Korea. p.15-20.
-
Jeon SW, Kim JU and Jung HC. (2013). A Study on the Forest classification for ecosystem services valuation. Journal of the Korean Society of Environmental Restoration Technology. 16:31-39.
[https://doi.org/10.13087/kosert.2013.16.3.031]

-
Kim C, Choo GC, Cho HS and Lim JT. (2015). Soil properties of cultivation sites for mountain-cultivated ginseng at local level. Journal of Ginseng Research. 39:76-80.
[https://doi.org/10.1016/j.jgr.2014.06.004]

-
Kim KY, Han KM, Kim HJ, Jeon KS, Kim CW and Jung CR. (2020). The study of soil chemical properties and soil bacterial communities on the cultivation systems of Cnidium officinale Makino. Korean Journal of Environmental Agriculture. 39:1-9.
[https://doi.org/10.5338/KJEA.2020.39.1.1]

- Kim KY, Um YR, Jeong DH, Kim HJ, Kim MJ and Jeon KS. (2019a). The correlation between growth characteristics and location environment of wild-simulated ginseng(Panax ginseng C. A. Meyer). Korean Journal of Plant Resources. 32:463-470.
-
Kim KY, Um YR, Jeong DH, Kim HJ, Kim MJ and Jeon KS. (2019b). Study on the correlation between the soil bacterial community and growth characteristics of wild-simulated ginseng (Panax ginseng C. A. Meyer). Korean Journal of Environmental Biology. 37:380-388.
[https://doi.org/10.11626/KJEB.2019.37.3.380]

- Korea Forest Service(KFS). (2016). 2016 Statistical yearbook of forest. Korea Forest Service, Daejeon, Korea. p.414.
- Korea Forestry Promotion Institute(KOFPI). (2013). The cultivation of wild-simulated ginseng: In wild-simulated ginseng and cultural environments. Korea Forestry Promotion Institute. Seoul, Korea. p. 14-34.
- Kozlowski TT and Pallardy SG. (1999). Physiology of Woody Plants(2nd Eds.) Academic Press. London, England. p.411.
- Kwon SD, Kang JH, Yoon JH and Moon HS. (2011). An analysis on site, soil and cultivation characteristics of korean mountain cultivated ginseng(Panax ginseng) field. Journal of Agricultural and Life Science. 45:81-88.
- Lee DS. (2010). Weather characteristic and growth of a forest ginseng cultivation site. Journal of Korean Forest Society. 99: 863-870.
- Lee HS and Shim JK. (1994). Studies on the microbial population and the amylase activity of the forest soil. Korean Journal of Ecology. 17:171-183.
-
Lee YM, Ahn JH, Choi YM, Weon HY, Yoon JH and Song JK. (2015). Bacterial core community in soybean rhizosphere. Korean Journal of Microbiology. 51:347-354.
[https://doi.org/10.7845/kjm.2015.5052]

-
Li H, Ye D, Wang X, Settles ML, Wang J, Hao Z, Zhou L, Dong P, Jiang Y and Ma Z. (2014a). Soil bacterial communities of different natural forest types in Northeast China. Plant and Soil. 383:203-216.
[https://doi.org/10.1007/s11104-014-2165-y]

-
Li Y, Ying YX and Ding WL. (2014b). Dynamics of Panax ginseng rhizospheric soil microbial community and their metabolic function. Evidence-Based Complementary and Alternative Medicine. 160373. (cited by 2020 March 31).
[https://doi.org/10.1155/2014/160373]

- Lim SU. (2005). Plant growth and nutrients: In fertilizer. Ilsin. Seoul, Korea. p.38-45.
-
Liu WW, Liu MC, Li WH, Zeng FS and Qu Y. (2016). Influence of ginseng cultivation under larch plantations on plant diversity and soil properties in Liaoning Province, Northeast China. Journal of Mountain Science. 13:1598-1608.
[https://doi.org/10.1007/s11629-015-3753-x]

-
Mechri B, Mariem FB, Baham M, Elhadj SB and Hammami M. (2008). Change in soil properties and the soil microbial community following land spreading of olive mill wastewater affects olive trees key physiological parameters and the abundance of arbuscular mycorrhizal fungi. Soil Biology and Biochemistry. 40:152-161.
[https://doi.org/10.1016/j.soilbio.2007.07.020]

-
Nacke H, Thürmer A, Wollherr A, Will C, Hodac L, Herold N, Schöning I, Schrumpf M and Daniel R. (2011). Pyrosequencing-based assessment of bacterial community structure along different management types in german forest and grassland soils. PLoS ONE. 6:e17000. (cited by 2020 Feb 21).
[https://doi.org/10.1371/journal.pone.0017000]

-
Nguyen NL, Kim YJ, Hoang VA, Subramaniyam S, Kang JP, Kang CH and Yang DC. (2016). Bacterial diversity and community structure in Korean ginseng field soil are shifted by cultivation time. PLoS ONE 11:e0155055. (cited by 2020 Jan 8).
[https://doi.org/10.1371/journal.pone.0155055]

-
Park YD, Kwon TH and Eo SH. (2016). Analysis of soil bacterial community in Ihwaryeong and Yuksimnyeong restoration project sites linking the Ridgeline of Baekdudaegan. Journal of Agriculture and Life Science. 50:117-124.
[https://doi.org/10.14397/jals.2016.50.1.117]

- Pettersson M. (2004). Factors affecting rates of change in soil bacterial communities. University of Lund. Lund, Sweden. p.17-27.
-
Philippot L, Raaijmakers JM, Lemanceau P and van der Putten WH. (2013). Going back to the roots: The microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11:789-799.
[https://doi.org/10.1038/nrmicro3109]

-
Prasad R, Kumar M and Varma A. (2015). Role of PGPR in soil fertility and plant health. In Egamberdieva et al., (eds.)., Plant growth promoting rhizobacteria(PGPR) and medicinal plant. Springer Nature Switzerland AG. Basel, Switzerland. p.247-260.
[https://doi.org/10.1007/978-3-319-13401-7_12]

-
Preem JK, Truu J, Truu M, Mander Ü, Oopkaup K, Lõhmus K, Helmisaari HS, Uri V and Zobel M. (2012). Bacterial community structure and its relationship to soil physico-chemical characteristics in alder stands with different management histories. Ecological Engineering. 49:10-17.
[https://doi.org/10.1016/j.ecoleng.2012.08.034]

- Rural Development Administration(RDA). (2013). Analysis manual of comprehensive examination laboratory(soil, plant, water and liquid manure). Rural Development Administration. Suwon, Korea. p.31-53.
-
Russell AE, Raich JW, Valverde-Barrantes OJ and Fisher RF. (2007). Tree species effects on soil properties in experimental plantation in tropical moist forest. Soil Science Society of America Journal. 71:1389-1397.
[https://doi.org/10.2136/sssaj2006.0069]

-
Schloss PD, Westcott SL, Ryabin T, Hall JR, Hartmann M, Hollister EB, Lesniewski RA, Oakley BB, Parks DH, Robinson CJ, Sahl JW, Stres B, Thallinger GG, van Horn DJV and Weber CF. (2009). Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology. 75:7537-7541.
[https://doi.org/10.1128/AEM.01541-09]

-
Schloss PD. (2009). A high-throughput DNA sequence aligner for microbial ecology studies. PLoS ONE. 4:e8230. (cited by 2020 March 31).
[https://doi.org/10.1371/journal.pone.0008230]

-
Shade A and Handelsman J. (2012). Beyond the venn diagram: The hunt for a core microbiome. Environmental Microbiology. 14:4-12.
[https://doi.org/10.1111/j.1462-2920.2011.02585.x]

- Son JG and Cho JY. (2009). Effect of organic material treatments on soil aggregate formation in reclaimed tidelands. Korean Journal of Soil Science and Fertilizer. 42:201-206.
-
Song X, Tao B, Guo J, Li J and Chen G. (2018). Changes in the Microbial community structure and soil chemical properties of vertisols under different cropping systems in Northern China. Frontiers in Environmental Science. 6:132. (cited by 2020 Jan 10).
[https://doi.org/10.3389/fenvs.2018.00132]

- Suh HM, Seo SM, Woo SY and Lee DS. (2011). Forest cultivated ginseng in Korea: All cure medicinal plants. Journal of Medicinal Plants Research. 5:5331-5336.
-
Suleiman AKA, Manoeli L, Boldo JT, Pereira MG and Roesch LFW. (2013). Shifts in soil bacterial community after eight years of land-use change. Systematic and Applied Microbiology. 36:137-144.
[https://doi.org/10.1016/j.syapm.2012.10.007]

-
Sun H, Wang Q, Liu N, Li L, Zhang C, Liu Z and Zhang Y. (2017). Effects of different leaf litters on the physicochemical properties and bacterial communities in Panax ginseng-growing soil. Applied Soil Ecology. 111:17-24.
[https://doi.org/10.1016/j.apsoil.2016.11.008]

- Tsai SH, Selvam A, Chang YP and Yang SS. (2009). Soil bacterial community composition across different topographic sites characterized by 16S rRNA gene clones in the fushan forest of Taiwan. Botanical Studies. 50:57-68.
-
Wang Q, Sun H, Xu C, Ma L, Li M, Shao C, Guan Y, Liu N, Liu Z, Zhang S, Zhang L and Zhang Y. (2019). Analysis of rhizosphere bacterial and fungal communities associated with rusty root disease of Panax ginseng. Applied Soil Ecology. 138:245-252.
[https://doi.org/10.1016/j.apsoil.2019.03.012]

- Woo SY and Lee DS. (2002). A study on the growth and environment of Panax ginseng in the different forest strands(I). Korean Journal of Agricultural and Forest Meteorology. 4:65-71.
-
Wu Z, Liu Q, Li Z, Cheng W, Sun J, Guo Z, Li Y, Zhou J, Meng D, Li H, Lei P and Yin H. (2018). Environmental factors shaping the diversity of bacterial communities that promote rice production. BMC Microbiology. 18:51. (cited by 2020 Feb 11).
[https://doi.org/10.1186/s12866-018-1174-z]

-
Xia Z, Bai E, Wang Q, Gao D, Zhou J, Jiang P and Wu J. (2016). Biogeographic distribution patterns of bacteria in typical chinese forest soils. Frontiers in Microbiology. 7:1106. https://ncbi.nlm.nih.gov/pmc/articles/PMC4942481, (cited by 2020 Feb 8).
[https://doi.org/10.3389/fmicb.2016.01106]

- Yun CW and Moon HS. (2009). Classification of forest vegetation type and environmental properties in Limestone area of Korea. Journal of Agricultural and Life Science. 43:1-8.