황해쑥 품종 구별을 위한 유전체 염기서열 기반 genomic SSR marker 개발
This is an open access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Artemisia argyi is an economically important medicinal plant with in the genus of Artemisia. However, the lack of the molecular markers hinders the understanding of genetic diversity and cultivar discrimination. Therefore, the main objectives of this study were to characterize the SSRs of Artemisia argyi in the reference genome, and to developed polymorphic SSRs for cultivar discrimination and population genetic studies.
Novel 100 simple sequence repeat (SSR) markers were designed based on the whole-genome sequence data of Artemisia argyi, and selected 78 polymorphic markers were selected. Upon the assessment of the five accessions with the full set of 78 SSR, the major allele frequency (MAF) ranged from 0.20 to 1, with an average of 0.36. In addition, the number of alleles per locus ranged from 2 to 9 alleles with a mean value of 5.2 alleles per locus. The polymorphism information content (PIC) values ranged from 0.27 to 0.87 with a mean PIC value of 0.69.
The Artemisia argyi alleles were detected in 5 accessions, among them six primers (argyi-gSSR003, argyi-gSSR014, argyi-gSSR035, argyi-gSSR036, argyi-gSSR093, argyig-SSR094) produced five cultivar-specific loci, therefore very promising for cultivar identification, genetic diversity and breeding studies in Artemisia argyi.
Keywords:
Artemisia argyi, Cultivar Discrimination, Microsatellite, Molecular Marker, Simple Sequence Repeats서 언
쑥은 국화과 (Asteraceae) 쑥 속 (Artemisia L.) 500여 종에 속하는 다년생 초본 식물로 한국, 중국, 일본 등 동아시아에 분포하며 식용, 약용, 향신료 등으로 사용된다 (Torrell et al., 2003; Lee et al., 2009). 아시아에서 황해쑥 (A. argyi), 사철쑥 (A. capillaris), 더위지기 (A. iwayomogi) 등이 전통의학에 많이 활용되어 왔으며, 그 중 황해쑥은 한의학에서 온경 (溫經), 지혈 (止血), 및 안태 (安胎) 등에 효능이 있는 것으로 알려져 있으며 냉증에 의한 복부통증, 설사 및 소화관내 출혈 억제를 위한 약재로 이용되어왔다 (Zhao et al., 1993; Sung et al., 2008).
농생물자원의 주권적 권리 확보의 중요성이 부각됨에 따라 식물품종보호제도 (plant variety protection system, PVPS)의 시행이 의무화되고 있다. 따라서 육종가의 권리 및 품종의 지적 재산권을 보호하기 위하여 분자표지 기반의 품종판별 기술개발이 활발하게 이루어지고 있다. 그동안 품종판별은 형태적인 특성이나 생화학적 특성을 기반으로 이루어져 왔으나, 이러한 방법은 재배환경과 재료의 보관상태에 따라 결과에 영향을 받을 수 있다 (Chen and Nelson, 2004; Kwon et al., 2005).
최근 국제식물신품종보호동맹 (international union for the protection of new varieties of plants, UPOV)는 이러한 문제점을 보완하기 위하여 SSR (simple sequence repeat) 또는 SNP (single nucleotide polymorphism) 분자표지를 활용하여 품종의 신규성을 보완하는 것을 제안하고 있다 (UPOV, 2010). 또한 유전자원이 활용되어 생기는 이익 및 공유에 대한 가치가 높아지면서 나고야 의정서가 채택되었으며, 이에 국가 자원의 주권을 확보하기 위한 품종판별 연구가 진행되고 있다 (Bang et al., 2011; Buck and Hamilton, 2011).
그동안 종 동정, 품종판별 및 유전적 다양성 분석에 restriction fragment length polymorphism (RFLP), random amplified polymorphic DNA (RAPD), amplified fragment length polymorphism (AFLP), single nucleotide polymorphisms (SNP), simple sequence repeat (SSR) 등의 분자 marker가 활용되어왔다 (Fukunaga et al., 2002; Gobert et al., 2002; Hasan et al., 2006; Mylonas et al., 2014; Stanley et al., 2020).
최근 유전체와 전사체 분석을 기반으로 황해쑥의 전체 게놈의 크기 (3.89 Gb), 염색체 구성 (2n = 34) 및 테르페노이드생합성 경로에 관여하는 유전자가 밝혀졌다. 그러나 유전체 상에 분포하는 SSR 발굴에 대한 연구는 일부 쑥 속 식물의 엽록체 유전체를 대상으로 제한적으로 수행되어왔다 (Liu et al., 2013; Meng et al., 2019; Shahzadi et al., 2020; Chen et al., 2022).
SSR은 1 - 6 bp의 짧은 DNA 서열이 병렬적으로 반복되어 핵, 미토콘드리아 및 엽록체 등에 존재하며 (Khera et al., 2015; Djedid et al., 2021), 높은 수준의 다형성과 공우성, 재현성 등의 장점이 있다 (Zane et al., 2002). 최근 차세대염기서열분석 (next generation sequencing) 기술이 발전하면서 다양한 작물에서 SSR marker 개발이 활발하게 진행되어 품종판별, 유전적 다양성 분석, 집단구조 분석 연구 분야에 활용되고 있다 (Van Inghelandt et al., 2010; Singh et al., 2020; Kumar et al., 2022).
현재까지 SSR marker를 활용한 다양성 분석 및 품종판별은 구기자 (Chung et al., 2009), 콩 (Kim et al., 2006), 국화 (Park and Hong, 2013), 잔대 (Jeong et al., 2022) 등의 다양한 작물에서 활용되었다. 쑥 속 (A. maritima, A. absinthium, A. selengensis, A. frigida) 식물의 경우 엽록체에서 유래된 SSR marker의 개발과 이를 활용한 자원의 분류와 다양성 분석이 일부 진행되었다 (Shahzadi et al., 2020; Chen et al., 2022). 그러나 황해쑥 (A. argyi) genome에 광범위하게 존재하는 SSR marker 개발은 현재까지 보고된 바가 없다.
본 연구에서는 황해쑥 유전체 염기서열을 바탕으로 SSR marker를 개발하였으며 보유하고 있는 자원을 바탕으로 다형성을 검정하고 품종을 식별할 수 있는 최소한의 marker set 선발을 통하여 효율적인 품종 구분법을 제시하고자 하였다.
재료 및 방법
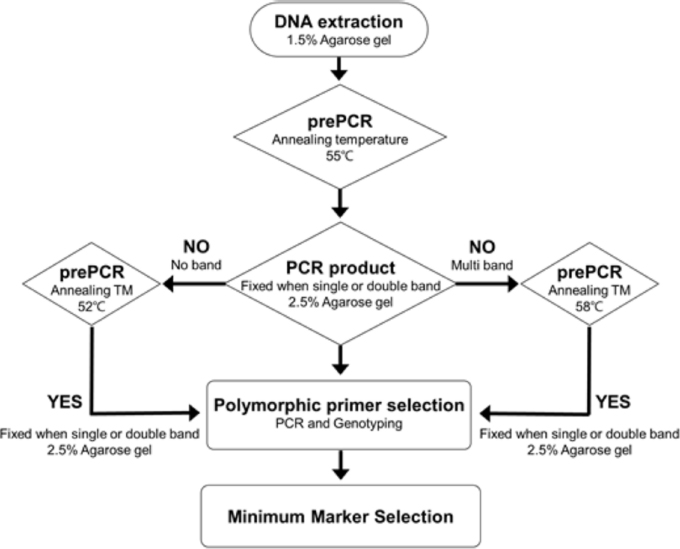
1. 실험 재료 및 DNA 추출
본 실험에서는 농촌진흥청 인삼특작부 약용작물과에서 보유하고 있는 황해쑥 5 자원을 사용하였다 (Table 1). 황해쑥 자원의 잎을 액체질소를 사용하여 분쇄 후 GeneAll Plant SV mini kit (GeneAll, Seoul, Korea)를 사용하여 제공된 protocol에 따라 추출하였다. 추출된 DNA는 1.5% agarose gel에 전기영동 하여 확인 후, Epoch Microplate Spectrophotometer (BioTek, Winooski, VT, USA)를 사용하여 20 ng/㎕로 정량 후 사용하였다.
2. PCR 및 유전자형 분석
SSR 마커 개발을 위해 ‘평안애’ 품종을 대상으로 Illumina MiSeq sequencing을 수행한 후 SPAdes를 이용하여 (Bankevich et al., 2012) 약 778 Mb의 조립된 염기서열을 확보하였다. 조립된 contig들을 대상으로 MISA microsatellite finder를 이용하여 (Thiel et al., 2003) SSR 시퀀스 정보를 확인하였다. 이후 SSR motifs에 따라 di-nucleotide는 6 반복, tri-nucleotide는 5 반복, tetra-nucleotide 4 반복, penta-nucleotide 이상은 3 반복을 기준으로 설정하여 primer를 제작하였으며, 그 중에서 repeat type 별 반복 단위 수가 많은 primer를 선발하였다.
PCR 반응액 조성은 Excel TB 2× TaqPre-Mix (Inclone, Yongin, Korea) 15 ㎕와 10 p/mol primer (forward / revers) 2 ㎕, DW 19 ㎕, DNA 2 ㎕를 혼합하여 40 ㎕의 mixture를 사용하였다.
PCR 반응은 95℃에서 3 분 동안 pre-denaturation 한 뒤 95℃에서 30 초간 denaturation, 각 primer에 확립된 온도 조건에서 45 초간 annealing, 72℃에서 1 분간 extension을 34 회 반복하였고 최종 extension으로 72℃에서 15 분간 반응시켰다.
증폭된 PCR product는 2.5% agarose gel에서 확인 후, Fragment Analyzer (Advanced Analytical Technologies Inc., Heidelberg, Germany)를 사용하여 증폭된 단편의 크기를 분석하였다.
3. 데이터분석
증폭된 DNA 단편의 크기에 따라 allele 타입을 기록한 후 PowerMarker ver 3.25 (Liu and Muse, 2005)를 사용하여 number of allele (NA), major allele frequency (MAF), heterozygosity (Ho), polymorphic information content (PIC)를 분석하였다 (Liu and Muse 2005).
PowerMarker ver 3.25에 포함되어있는 ShareAllele distance를 기반으로 unweighted pair pop method with arithmetic mean (UPGMA) 방법을 사용하여 Phylogenetic tree를 제작하고 MEGA ver 5.2에서 확인하였다 (Tamura et al., 2011).
품종구분을 위한 최소 marker 수를 확인하기 위해 R studio program 의 poppr package (R Studio Team, 2015)를 사용하여 genotype accumulation curve를 확인하였다 (Kamvar et al., 2014) (Fig. 1).
결과 및 고찰
1. 황해쑥 SSR 특성
Illumina MiSeq sequencing을 이용하여 염기서열을 분석한 결과 총 778 Mb의 시퀀싱 데이터를 확보하였고, 총 contig의 갯수는 806,823으로 확인되었다. 평균 contig와 N50의 길이는 각각 965 bp와 1,466 bp로 확인되었다 (Table 2).
황해쑥 genome 시퀀스에서 총 455,776 개의 SSR이 탐색 되었다. 탐색 된 SSR 중 79,555 개의 primer가 디자인되었으며 그 중 임의로 100 세트의 primer (di-nucleotide 28, tri-nucleotide 32, tetra-nucleotide 40)를 선발하였다. 황해쑥 genome 내 SSR은 tetra-nucleotide 142,852 개 (31.34%), di-nucleotide 84,673 개 (18.58%), tri-nucleotide 80,201 개 (17.60%), hexanucleotide 60,723 개 (13.32%), penta-nucleotide 29,962 개 (6.57%), octa-nucleotide 22,350 개 (4.90%), deca-nucleotide 16,101 개 (3.53%), nona-nucleotide 14,782 개 (3.24%) 및 hepta-nucleotide 4,132 개 (0.91%) 순으로 분포하였다 (Table 3).
Frequency of different type of motif in Di-, Tri-, Tetra-, Penta-, Hexa-, Octa-, Nona- and Deca-nucleotide SSRs from Artemisia argyi.
di-nucleotide는 motif의 종류가 12 개로 확인되었으며 TA가 5,441 개(29.27%)로 가장 많았으며 그 다음은 AT (5,108 개, 27.48%), TG (1,833 개, 9.86%), AC (1,532 개, 8.24%) 순으로 분포하였다 (Table 4). tri-nucleotide는 60 개의 motif 중 TTA가 (2,802 개, 13.26%)로 가장 많았으며, AAT (2,732 개, 12.93%), ATT (1,115 개, 5.28%), TTG (972 개, 4.60%) 순으로 분포하였다. tetra-nucleotide는 236 개의 motif 중 AAAT의 빈도가 (3,863 개, 14.36%) 가장 많았으며 그 다음은 TTTA (3,646 개, 13.55%), TTAT (1,550 개, 5.76%), AATA (1,236 개, 4.59%) 순으로 확인되었다. penta-nucleotide는 589 개의 motif 중 AAAAT가 (863 개, 11.42%) 가장 많은 것으로 확인되었으며 그 다음은 TTTTA (699 개, 9.25%), TTTAT (318 개, 4.21%), AAATA (303 개, 4.01%) 순으로 분포하였다. hexa-nucleotide는 1,229 개의 motif 중 AAAAAT가 (231 개, 5.71%) 가장 많은 분포로 나타났으며, 그 다음은 TTTTTA (193 개, 4.77%), ATTTTT (114 개, 2.82%) 순으로 분포하였다. hepta-nucleotide는 444 개의 motif 중 AAAAAAT가 (102 개, 9.96%), octa-nucleotide는 123 개의 motif 중 AAAAAAAT가 (20 개, 8.97%), nona-nucleotide는 53 개의 motif 중 TTATTATTA가 (5 개, 7.81%), deca-nucleotide는 19 개의 motif 중 ATATATATAT가 (2 개, 10%)로 가장 많은 분포로 나타났다 (Table 4).
이전 연구에서, 황기 유전체에서 분석된 SSR은 147,766 개이며, di-nucleotide가 77.02%, tri-nucleotide가 19.04%, tetra-nucleotide가 2.67% 분포하는 것으로 보고되었다 (Hur et al., 2021). 반복되는 염기서열의 유형은 di-nucleotide에서 AG(AG/GA/TC/CT)가 48.5%로 가장 높은 비율을 차지하였고 tri-nucleotide에서는 AAT(AAT/ATA/TAA/ATT/TTA/TAT)가 34.90%의 비율을 차지하였다 (Hur et al., 2021).
잔대의 경우 84,106 개의 SSR이 전체 게놈상에 존재하였으며 di-nucleotide가 84.58%, tri-nucleotide가 11.99%, tetra—nucleotide가 1.52% 분포하였다 (Park et al., 2017). 동일한 과에 속하는 국화 유전체에서 분석된 SSR은 26,780 개이며, di-nucleotide가 61.5%, tri-nucleotide가 24.8%, tetra-nucleotide가 6.3% 순으로 보고하였으며, 반복 유형은 di-nucleotide 중 AC/CA가 93.5%로 대부분이었고, di-nucleotide에서는 AAC가 50.5%로 나타났다 (Thu et al., 2011).
본 연구결과 di-nucleotide, tri-nucleotide, tetra-nucleotide가 전체 유전체에서 60% 이상의 높은 비율이 관찰되어 기존 연구결과와 유사한 경향을 보였다. 또한 AT motif가 GC motif 보다 높은 비율로 존재하는 결과는 쌍자엽 식물의 특징과 일치하였다 (Cserhati, 2015).
2. SSR marker 개발 및 다형성 분석
제작된 100 세트 primer 중 80 개가 증폭되었고 그 중 78 세트에서 다형성 (polymorphism)이 확인되었다. 다형성이 확인된 SSR 마커의 타입은 di-nucleotide가 20 세트 (25%), tirnucleotide가 23 세트 (29%), tetra-nucleotide가 37 세트 (46%)로 확인되었다 (Table 5).

Genetic diversity parameters and sequence information of the 78 selected SSR markers for Artemisia argyi accessions used in this study.
78 세트의 SSR 마커의 MAF는 0.2 (argyi-gSSR003 등 16개)에서 1 (argyi-gSSR051, argyi-gSSR073) 사이로 확인되었으며 평균 MAF는 0.36 이었다. NA는 2 (argyi-gSSR033 등 3 개)에서 9 (argyi-gSSR016) 사이였으며 평균 NA는 5.2 개로 나타났다. HO는 0 (argyi-gSSR008 등 13 개)에서 1 (argyig-SSR016 등 9개) 이었고 평균 HO는 0.56으로 나타났다. PIC는 argyi-gSSR087이 0.27로 가장 낮았으며 argyi-gSSR016이 0.87로 가장 높게 나타났고, 전체 PIC 평균값은 0.69로 나타났다 (Table 5).
Di-nucleotide motif가 반복되는 20 개 marker의 MAF 평균값은 0.315로 나타났으며, NA, HO의 각 평균값은 5.5, 0.577로 나타났다. 평균 PIC은 0.728로 나타났다. tri-nucleotide motif가 반복되는 22 개 marker는 MAF 평균값이 0.358, NA, HO의 평균값은 각 5.4, 0.618로 나타났으며, 평균 PIC 값은 0.694로 나타났다. tetra-nucleotide motif가 반복되는 36 개의 marker는 MAF 평균값이 0.380, NA, HO의 평균값은 5.0, 0.511로 나타났으며 평균 PIC 값은 0.675로 나타났다. di-, tri-, tetra-nucleotide 순으로 다형성이 높게 나타나는 것을 확인하였다.
이전에 Cho 등 (2011)이 선발한 14 개의 SSR marker를 24 개의 감자 품종에 적용하여 평균 PIC이 0.76으로 보고한 결과와 Hur 등 (2021)이 10 개의 SSR marker를 개발하여 황기 81 자원에 적용하여 평균 PIC를 0.66으로 보고한 결과와 비교하였을 때, 본 연구에 개발한 SSR marker와 유사한 다형성의 범위를 갖는 것으로 확인되었다.
3. 황해쑥 품종구분을 위한 최소한의 marker set 선발
개발된 78 개의 SSR marker 중 품종 구분을 위한 효율적인 최소 marker set을 선발하기 위하여 R studio program에 포함되어있는 poppr package를 사용하여 accumulation curve를 확인한 결과, ‘평안애’를 포함한 5 자원을 1 개의 marker로 구분이 가능하였다 (Fig. 2).

The number of markers that can be used as the minimum markers was determined through the accumulation curve calculated using R studio's poppr package.
‘평안애’ 품종 구별을 위하여 ‘평안애’를 포함한 4 개의 황해쑥 계통을 토대로 유전형 (genotyping)을 분석한 결과, 6 세트의 SSR 마커 (argyi-gSSR003, argyi-gSSR014, argyi-gSSR035, argyi-gSSR036, argyi-gSSR093, argyi-gSSR094) 중에서 한세트만으로도 5 자원이 모두 구분되었다 (Fig. 3).
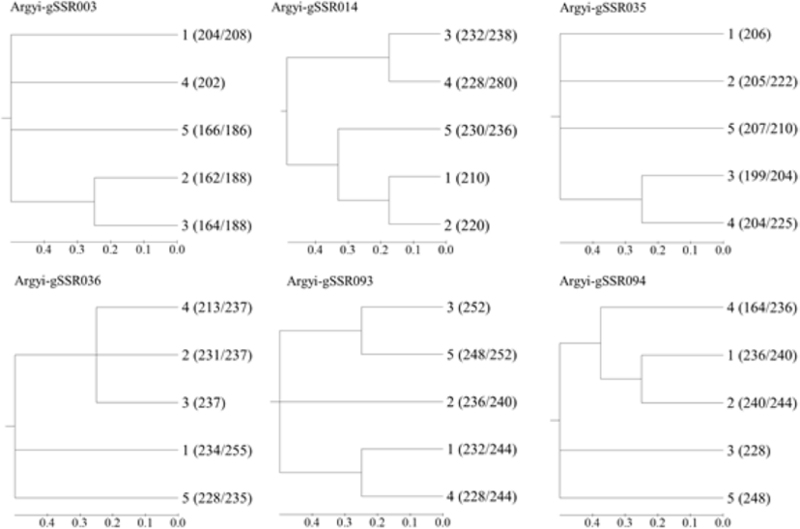
Phylogenetic tree applied to each minimum marker that discriminates A. argyi five accessions.The numbers in parentheses correspond to the sizes (base pair) of the DNA fragments.
‘평안애’는 argyi-gSSR003에서 164/188 bp, argyi-gSSR014에서 232/238 bp, argyi-gSSR035에서 199/204 bp, argyig-SSR036에서 237 bp, argyi-gSSR093에서 252 bp, argyigSSR094에서 228 bp의 특이적 유전자형을 나타냈다.
기존의 연구 결과, 국화 85 품종의 판별을 4 세트의 SSR marker를 사용하여 수행하였고 (Hong et al., 2013), 벼, 21 품종을 4 세트의 SSR marker로 판별이 가능한 것으로 보고하였다 (Kwon et al., 2006). 26 개의 콩 품종을 다형성이 높은 SSR marker 5 세트 중 2 세트를 조합하여 판별하였으며 (Jang et al., 2009), 14 개의 난 품종을 6 세트의 SSR marker 중 최소 2 세트를 이용하여 판별하였다 (Park et al., 2013). 기존의 연구 결과들을 비교하여 볼 때 본 연구에서 개발한 SSR marker는 황해쑥의 품종구분 및 순도 검정 등에 활용할 수 있을 것이다.
본 연구에서 ‘평안애’와 4 점의 황해쑥 계통을 대상으로 분석하였기 때문에 marker의 적용성이 제한적일 수 있으나, 분석에 사용된 자원이 유전적으로 가까움에도 불구하고 개체 간 식별이 가능한 것으로 보아 개발된 SSR marker는 품종개발 및 순도검정에 효율적으로 사용될 수 있을 것이다. 또한 향후 황해쑥 신품종의 유전적 구별성, 국내 및 국외 황해쑥 유전자원의 다양성 분석 등에 활용될 수 있을 것이다. 후속연구를 통해 국내·외 황해쑥 유전자원을 확보하여 78 세트의 SSR 마커를 토대로 다양성 분석 및 집단구조 분석 등의 연구가 수행되어야 할 것이다.
Acknowledgments
본 논문은 농촌진흥청 연구사업(과제번호: PJ017066)의 지원에 의해 이루어진 결과로 이에 감사드립니다.
References
-
Bang KH, Chung JW, Kim YC, Lee JW, Jo IH, Seo AY, Kim OT, Hyun DY, Kim DH and Cha SW. (2011). Development of SSR markers for identification of Korean ginseng(Panax ginseng C. A. Meyer) cultivars. Korean Journal of Medicinal Crop Science. 19:185-190.
[https://doi.org/10.7783/KJMCS.2011.19.3.185]

-
Bankevich A, Nurk S, Antipov D, Gurevich A, Dvorkin M, Kulikov AS, Lesin VM, Nikolenko SI, Pham S, Prjibelski AD, Pyshkin AV, Sirotkin AV, Vyahhi N, Tesler G, Alekseyev MA and Pevzner PA. (2012). SPAdes: A new genome assembler and its applications to single cell sequencing. Journal of Computational Biology. 19:455-477.
[https://doi.org/10.1089/cmb.2012.0021]

-
Buck M and Hamilton C. (2011). The Nagoya Protocol on access to genetic resources and the fair and equitable sharing of benefits arising from their utilization to the Convention on Biological Diversity. Review of European Community and International Environmental Law. 20:47-61.
[https://doi.org/10.1111/j.1467-9388.2011.00703.x]

-
Chen C, Miao Y, Luo D, Li J, Wang Z, Luo M, Zhao T and Liu D. (2022). Sequence characteristics and phylogenetic analysis of the Artemisia argyi chloroplast genome. Frontiers in Plant Science. 13: 906725. https://www.frontiersin.org/articles/10.3389/fpls.2022.906725/full, (cited by 2023 Feb 1).
[https://doi.org/10.3389/fpls.2022.906725]

-
Chen Y and Nelson RL. (2004). Genetic variation and relationships among cultivated, wild, and semiwild soybean. Crop Science. 44:316-325.
[https://doi.org/10.2135/cropsci2004.3160]

- Cho KS, Won HS, Jeong HJ, Cho JH, Park YE and Hong SY. (2011). Development of multiplex microsatellite marker set for identification of Korean potato cultivars. Horticultural Science and Technology. 29:366-373.
- Chung JW, Lee GA, Lee SS, Bang KH, Park CB and Park YJ. (2009). Cultivar discrimination of Korean and Chinese Boxthorn(Lycium chinense Mill. and Lycium barbarum L.) using SSR markers. Korean Journal of Medicinal Crop Science. 17:445-451.
-
Cserhati M. (2015). Motif content comparison between monocot and dicot species. Genomics Data. 3:128-136.
[https://doi.org/10.1016/j.gdata.2014.12.006]

-
Djedid IK, Terzaghi M, Brundu G, Cicatelli A, Laouar M, Guarino F and Castiglione S. (2021). Genetic diversity and differentiation of eleven medicago species from campania region revealed by nuclear and chloroplast microsatellites markers. Genes. 13:97. https://www.mdpi.com/2073-4425/13/1/97, (cited by 2023 Feb 1).
[https://doi.org/10.3390/genes13010097]

-
Fukunaga K, Wang Z, Kato K and Kawase M. (2002). Geographical variation of nuclear genome RFLPs and genetic differentiation in foxtail millet, Setaria italica (L.) P. Beauv. Genetic Resources and Crop Evolution. 49:95-101.
[https://doi.org/10.1023/A:1013852007770]

-
Gobert V, Moja S, Colson M and Taberlet P. (2002). Hybridization in the section Mentha(Lamiaceae) inferred from AFLP markers. American Journal of Botany. 89:2017-2023.
[https://doi.org/10.3732/ajb.89.12.2017]

-
Hasan M, Seyis F, Badani AG, Pons-Kühnemann J, Friedt W, Lühs W and Snowdon RJ. (2006). Analysis of genetic diversity in the Brassica napus L. gene pool using SSR markers. Genetic Resources and Crop Evolution. 53:793-802.
[https://doi.org/10.1007/s10722-004-5541-2]

-
Hong WJ, Khaing AA and Park YJ. (2013). Cultivar identification of Chrysanthemum(Dendranthema grandiflorum Pamat.) using SSR markers. Journal of the Korean Society of International Agriculture. 25:385-394.
[https://doi.org/10.12719/KSIA.2013.25.4.385]

-
Hur M, Um Y, Lee Y, Lee YJ, Koo SC, Park WT, Kim JH, Huh YC and Moon YH. (2021). Development of whole genome sequence based novel SSR markers in Astragalus membranaceus(Fisch). Korean Journal of Medicinal Crop Science. 29:418-424.
[https://doi.org/10.7783/KJMCS.2021.29.6.418]

- International Union for the Protection New Varieties of Plant(UPOV). (2010). The working group on biochemical and molecular techniques and DNA-profiling in particular(BMT/12/09): Application of SSR and SNP in maize variety identification and database construction. International Union for the Protection New Varieties of Plant. Ottawa, Canada. p.1-46.
- Jang SJ, Park SJ, Park KH, Song HL, Cho YG, Jong SK, Kang JH and Kim HS. (2009). Genetic diversity and identification of Korean elite soybean cultivars including certified cultivars based on SSR markers. Korean Journal of Crop Science. 54:231-240.
-
Jeong DH, Um Y, Yun YB, Huh JH, Kim J and Park HW. (2022). Investigation of morphological characteristics and genetic diversity of Adenophora triphylla(Thunb.) A. DC. using SSR markers. Korean Journal of Medicinal Crop Science. 30:411-418.
[https://doi.org/10.7783/KJMCS.2022.30.6.411]

-
Kamvar ZN, Tabima JF and Grünwald NJ. (2014). Poppr: An R package for genetic analysis of populations with clonal, partially clonal, and/or sexual reproduction. PeerJ. 2:e281. https://peerj.com/articles/281/, (cited by 2023 Feb 2).
[https://doi.org/10.7717/peerj.281]

-
Khera P, Saxena R, Sameerkumar CV, Saxena K and Varshney RK. (2015). Mitochondrial SSRs and their utility in distinguishing wild species, CMS lines and maintainer lines in pigeonpea(Cajanus cajan L.). Euphytica. 206:737-746.
[https://doi.org/10.1007/s10681-015-1504-2]

- Kim SH, Chung JW, Moon JK, Woo SH, Cho YG, Jong SK and Kim HS. (2006). Discrimination of Korean soybean cultivars by SSR markers. Korean Journal of Crop Science. 51:658-668.
-
Kumar SPJ, Susmita C, Sripathy KV, Agarwal DK, Pal G, Singh AN, Kumar S, Rai AK and Simal-Gandara J. (2022). Molecular characterization and genetic diversity studies of Indian soybean(Glycine max (L.) Merr.) cultivars using SSR markers. Molecular Biology Reports. 49:2129-2140.
[https://doi.org/10.1007/s11033-021-07030-4]

- Kwon YS, Lee JM, Yi GB, Yi SI, Kim KM, Soh EH, Bae KM, Park EK, Song IH and Kim BD. (2005). Use of SSR markers to complement tests of distinctiveness, uniformity, and stability(DUS) of pepper(Capsicum annuum L.) varieties. Molecules and Cells. 19:428-435.
-
Kwon YS, Park EK, Park CU, Bae KM, Yi SI and Cho IH. (2006). Identification of rice variety using simple sequence repeat(SSR) marker. Journal of Life Science. 16:1001-1005.
[https://doi.org/10.5352/JLS.2006.16.6.1001]

-
Lee JH, Lee JW, Sung JS, Bang KH and Moon SG. (2009). Molecular authentication of 21 Korean Artemisia species (compositae) by polymerase chain reaction-restriction fragment length polymorphism based on trnL–F region of chloroplast DNA. Biological and Pharmaceutical Bulletin. 32:1912-1916.
[https://doi.org/10.1248/bpb.32.1912]

-
Liu K and Muse SV. (2005). PowerMarker: An integrated analysis environment for genetic marker analysis. Bioinformatics. 21:2128-2129.
[https://doi.org/10.1093/bioinformatics/bti282]

-
Liu Y, Huo N, Dong L, Wang Y, Zhang S, Young HA, Feng X and Gu YQ. (2013). Complete chloroplast genome sequences of Mongolia medicine Artemisia frigida and phylogenetic relationships with other plants. PloS one. 8:e57533. https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0057533, (cited by 2023 Feb 2).
[https://doi.org/10.1371/journal.pone.0057533]

-
Meng D, Xiaomei Z, Wenzhen K and Xu Z. (2019). Detecting useful genetic markers and reconstructing the phylogeny of an important medicinal resource plant, Artemisia selengensis, based on chloroplast genomics. PLoS One. 14:e0211340. https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0211340, (cited by 2023 Feb 2).
[https://doi.org/10.1371/journal.pone.0211340]

- Mylonas IG, Georgiadis A, Apostolidis AP, Bladenopoulos K and Koutsika-Sotiriou M. (2014). Barley cultivar discrimination and hybrid purity control using RAPD markers. Romanian Biotechnological Letters. 19:9421-9428.
-
Park KC, Kim YG, Hwang BK, Gil JS, Chung H, Park SG, Hong CP and Lee Y. (2017). Development of simple sequence repeat markers from Adenophora triphylla var. japonica(Regel) H. Hara using next generation sequencing. Korean Journal of Medicinal Crop Science. 25:411-417.
[https://doi.org/10.7783/KJMCS.2017.25.6.411]

-
Park PH, Park YJ, Kim MS, Lee YR, Park PM, Lee DS and Yae BW. (2013). Analysis of genetic diversity and identification of domestic bred Phalaenopsis varieties using SRAP and SSR markers. Horticultural Science and Technology. 31:337-343.
[https://doi.org/10.7235/hort.2013.12204]

- R Studio Team. (2015). RStudio: Integrated development for R. RStudio, Inc., Boston. MA, USA. http://www.rstudio.com, . (cited by 2023 Feb 2).
-
Shahzadi I, Mehmood F, Ali Z, Ahmed I and Mirza B. (2020). Chloroplast genome sequences of Artemisia maritima and Artemisia absinthium: Comparative analyses, mutational hotspots in genus Artemisia and phylogeny in family Asteraceae. Genomics. 112:1454-1463.
[https://doi.org/10.1016/j.ygeno.2019.08.016]

-
Singh RB, Mahenderakar MD, Jugran AK, Singh RK and Srivastava RK. (2020). Assessing genetic diversity and population structure of sugarcane cultivars, progenitor species and genera using microsatellite(SSR) markers. Gene. 753:144800. https://www.sciencedirect.com/science/article/pii/S0378111920304698, (cited by 2023 Feb 2).
[https://doi.org/10.1016/j.gene.2020.144800]

-
Stanley A, Menkir A, Paterne A, Ifie B, Tongoona P, Unachukwu N, Meseka S, Mengesha W and Gedil M. (2020). Genetic diversity and population structure of maize inbred lines with varying levels of resistance to Striga hermonthica using agronomic trait-based and SNP markers. Plants. 9:1223. https://www.mdpi.com/2223-7747/9/9/1223, (cited by 2023 Feb 2).
[https://doi.org/10.3390/plants9091223]

- Sung JS, Lee JH, Lee JW, Bang KH, Yeo JH, Park CG, Park HK, Seong NS and Moon SG. (2008). Phylogenetic analysis of Artemisia spp. by morphological characteristics of reproductive organs in Korea. Korean Journal of Medicinal Crop Science. 16:218-224.
-
Tamura K, Peterson D, Peterson N, Stecher G, Nei M and Kumar S. (2011). MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution. 28:2731-2739.
[https://doi.org/10.1093/molbev/msr121]

- Thu MK, Ra SB, Lee GA, Lee MC, Park HS, Kim DC, Lee CH, Choi HG, Jeon NB and Choi BJ. (2011). Distribution and frequency of SSR motifs in the Chrysanthemum SS-Renrich library through 454 pyrosequencing technology. Journal of the Korean Society of International Agriculture. 23:546-551.
-
Torrell M, Cerbah M, Siljak-Yakovlev S, and Vallès J. (2003). Molecular cytogenetics of the genus Artemisia(Asteraceae, Anthemideae): Fluorochrome banding and fluorescence in situ hybridization. I. Subgenus Seriphidium and related taxa. Plant Systematics and Evolution. 239:141-153.
[https://doi.org/10.1007/s00606-002-0259-0]

-
Van Inghelandt D, Melchinger AE, Lebreton C and Stich B. (2010). Population structure and genetic diversity in a commercial maize breeding program assessed with SSR and SNP markers. Theoretical and Applied Genetics. 120:1289-1299.
[https://doi.org/10.1007/s00122-009-1256-2]

-
Zane L, Bargelloni L and Patarnello T. (2002). Strategies for microsatellite isolation: A review. Molecular Ecology. 11:1-16.
[https://doi.org/10.1046/j.0962-1083.2001.01418.x]

-
Zhao QC, Kiyohara H and Yamada H. (1993). Anti-complementary neutral polysaccharides from leaves of Artemisia princeps. Phytochemistry. 35:73-77.
[https://doi.org/10.1016/S0031-9422(00)90512-0]