초기 염류 스트레스 반응 인삼 잎 단백질체 분석
© The Korean Society of Medicinal Crop Science
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License ( http://creativecommons.org/licenses/by-nc/3.0 ) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Salt stress is one of the major abiotic stresses affecting the yield of ginseng (Panax ginseng C. A. Meyer). The objective of this study was to identify bio-marker, which is early responsive in salt stress in ginseng, using proteomics approach. Ginseng plants were exposed to 5 ds/m salt concentration and samples were harvested at 0, 6, 12 and 18 hours after exposure. Total proteins were extracted from ginseng leaves treated with salt stress using Mg/NP-40 buffer and were separated on high resolution 2-DE. Approximately 1003 ± 240 (0 h), 992 ± 166 (6 h), 1051 ± 51 (12 h) and 990 ± 160 (18 h) spots were detected in colloidal CBB stained 2D maps. Among these, 8 spots were differentially expressed and were identified by using MALDI-TOF/TOF MS or/and LC-MS/MS. Ethylene response sensor-1 (spot GL 1), nucleotide binding protein (spot GL 2), carbonic anhydrase-1 (spot GL 3), thylakoid lumenal 17.9 kDa protein (spot GL 4) and Chlorophyll a/b binding protein (spot GL 5, GL 6) were up-regulated at the 12 and 18 hour, while RuBisCO activase B (spot GL 7) and DNA helicase (spot GL 8) were down-regulated. Thus, we suggest that these proteins might participate in the early response to salt stress in ginseng leaves.
Keywords:
Ginseng, Protein Biomarker, Proteomics, Salt Stress, 2-DE서 언
인삼은 두릅나무과 (Araliaceae)에 속하는 다년생 약초로써 (Attele et al., 1999), 일반적으로 강장제로 사용되어왔으며 임 상적, 약학적으로 항암, 순환계 쇼크 방지, 조혈작용 촉진, 면 역 기능 촉진 효과가 있다고 보고되어 있다 (Kim et al., 2003). 특히, 인삼의 ginsenoside라는 화합물은 의학적으로 콜 레스테롤 수치 감소, 에너지 증가 등에 효과가 있는 것으로 알려져 있다 (Jung et al., 2003). 한국을 대표하는 약용작물인 인삼은 단일 품목으로 수출액 1 위를 차지하고 있는 우리나라 대표 농산물이며, 국민 소득 수준 향상, 건강에 대한 관심 고 조, 웰빙 등의 영향으로 매년 생산량과 소비가 꾸준히 증가하 고 있는 추세이다.
인삼은 주로 한국, 중국의 동아시아와 캐나다, 미국의 북아 메리카를 중심으로 재배, 생산되고 있으며, 인삼을 대상으로 수행된 단백질체 연구는 고려인삼 (Panax ginseng C. A. Mey.), 미국삼 (Panax quinquefolius L.), 죽절삼 (P. japonicus (Nees) C. A. Mey.)의 세 종을 중심으로 이루어지고 있다 (Nam et al., 2005; Kim et al., 2011). 이 중 Panax ginseng (고려인삼)을 대상으로 수행된 단백질체 연구는 인삼 의 잎, 줄기 (Sun et al., 2011), 뿌리 (Kim et al., 2003, 2004a) 등을 대상으로 하는 부위별 단백질 분석 (Nam et al., 2005; We et al., 2007), 생육 단계별 인삼 뿌리 단백질 분석 (Ma et al., 2013), 인삼 품종 간의 단백질 비교 분석 (Nagappan et al., 2012) 등이 활발하게 진행되고 있다.
최근 급격한 기후 변화의 영향으로 고온 장해 및 염류 장해 가 많이 발생하여 인삼의 안정적 생산이 어려운 상황이다 (Bang et al., 2012; Kim et al., 2013). 특히, 염류 장해로 인해 인삼 생산량이 매년 약 20% 정도 감소되고 있어 문제가 되고 있다. 인삼은 적정 토양 염류 농도가 0.5 ds/m로 매우 낮아 염류에 약한 작물에 속하며 염류 농도가 높을 경우 생육 이 위축되고 심하면 고사한다. 따라서 생산의 안정성 증대를 위하여 염류 스트레스에 저항성을 가진 인삼 품종 육성 및 개 발과 더불어 인삼 내재해성 품종 육성 시 내염성, 내고온성 등의 목적 형질을 실험실에서 신속하고 정확하게 탐색하기 위 한 바이오마커의 개발이 필요하다. Kim 등 (2008)은 염류 스 트레스에 반응하는 인삼 단백질에 대한 분석을 통하여 단백질 바이오마커 개발 연구를 수행하였는데, 7일 동안 염류 처리를 한 후 수확한 잎으로 단백질 분석을 하였다.
단백질체학은 단백질 수준에서 유전자 발현의 특성을 파악 하여, 단백질 발현에 대한 정보 획득 및 그 발현 단백질의 기 능을 구명하는데 있어 유용한 기술이라고 할 수 있다 (Pandey and Mann, 2000). 이러한 단백질체학적 접근법은 이차원 전기 영동 (2-DE) 분석으로 특정 조건에 따라 다르게 발현되는 단 백질을 등전점 (pI)과 분자량에 따라 분리한 후 각각의 단백질 을 질량 분석기를 이용하여 아미노산 서열을 결정하고, 이를 바탕으로 단백질이나 유전체 데이터베이스를 생물정보학적 분 석을 수행하여 단백질을 동정하는 과정이다 (Rabilloud, 2002). 단백질체학적 접근법을 이용하면 염류 스트레스 처리 시간대 별 인삼 잎 단백질들의 비교가 가능하며, 그 단백질들의 비교 를 통하여 염류 스트레스 처리 시간대별로 나타나는 특정 단 백질을 동정하여 바이오마커로서도 활용할 수 있다.
본 연구는 염류 스트레스를 처리한 인삼 잎에서 단백질들을 2-DE로 분리한 후, MALDI-TOF/TOF MS, LC-MS/MS 등 단백질체 분석을 통하여 초기 염류 스트레스 반응에 관여하는 단백질들을 동정하고, 염류 스트레스 처리 시간 별로 차이를 보 이는 단백질을 확보하여 바이오마커로서 활용하고자 수행하였다.
재료 및 방법
1. 실험재료
본 실험에서 사용된 인삼은 농촌진흥청 국립원예특작과학원 인삼특작부의 인삼 재배 포장에서 육성 중인 품종 중 ‘천량’ 4년생이다. 염류 처리는 Kim 등 (2008)의 방법에 의해서 수행 되었다. 인삼의 지상부를 염류 농도 5 ds/m의 혼합용액 [21 mM KNO3, 6.1 mM KH2PO4, 6.2 mM MgSO4 • 7H2O] 에 처리하여, 각각 0시간, 6시간, 12시간 및 18시간의 시간 별로 인삼 식물체에서 잎을 수확하여 단백질 등의 분석에 활 용하였다.
2. 단백질 추출
1 g의 인삼 잎에 액체 질소를 부어 완전히 마쇄한 후, 마쇄 된 가루에 2% 2-mercaptoethanol을 첨가한 Mg/NP-40 단백 질 추출용액 [0.5 M Tris-HCI, 2% Nonidet P (NP)-40, 20 mM MgCI2] 10mℓ을 첨가시켜 균질화 시키고 12,000 rpm 으로 4°C에서 10분간 원심분리를 수행하여 상등액만 분리하 였다. 추출한 단백질 부피의 4배 양으로 12.5% trichloroacetic acid (TCA)/acetone을 첨가하여 균질화 후 1시간 동안 −20°C 에 침전시켰다. 침전 후 4°C에서 12,000 rpm으로 5분간 원심 분리 하여 얻은 침전물에 80% acetone을 첨가하여 현탁 후 침 전물을 얻는 방법과 같은 조건으로 다시 원심분리하는 세척 과 정을 두 번 반복하였다. 마지막으로 추출된 단백질에 80% acetone 1.2mℓ을 첨가하여 micro centrifuge tube에 옮기고 −20°C에서 보관하였다.
3. 단백질 정량
추출된 단백질은 2-DE Quant Kit (GE Healthcare, Piscataway, NJ, USA)를 사용하여 정량 분석하였다. 80% acetone에 침전 되어 있는 단백질을 필요한 양만큼 micro centrifuge tube에 옮기고 원심분리를 수행하여 acetone을 완전 히 제거한 후, rehydration 용액 [7 M Urea, 2 M Thiourea, 4% (v/v) CHAPS, 2 M dithiothreitol (DTT), 0.5% (v/v) IPG buffer pH 4-7 (GE Health, Waukesha, WI, USA)]에 녹였다. Pellet pestle을 이용하여 완전히 녹인 후, 2분간 원심 분리 하여 buffer에 녹지 않는 찌꺼기를 가라앉힌 후, 5μℓ씩 1.5mℓ micro centrifuge tube에 담았다. 500μℓ precipitant (GE Healthcare, Piscataway, NJ, USA)를 첨가하여 섞어 준 뒤, 상온에 2 ~ 3분간 놓아 둔 후 500μℓ co-precipitant (GE Healthcare, Piscataway, NJ, USA)를 첨가하여 가볍게 현탁 시킨 후 10,000 rpm 이상으로 5분간 원심분리 하여 상등액을 완전히 제거하였다. 100μℓ copper solution (GE Healthcare, Piscataway, NJ, USA)과 400μℓ 증류수를 첨가 후, 침전물이 완전히 용해될 때까지 혼합하였다. 각 튜브당 1mℓ working color reagent [color reagent A : B = 100 : 1 (GE Healthcare, Piscataway, NJ, USA)]를 첨가한 즉시 부드럽게 섞어 주었다. 상온에서 15 ~ 20분간 놓아 둔 후, spectrophotometer (Mecasys, Daejeon, South Korea)를 이용하여 480nm 파장에서 단백질 농 도를 측정하였다.
4. 이차원전기영동 (2-DE) 분석
2-DE 분석은 Kim 등 (2012)의 방법으로 수행하였다. 각각 의 단백질 시료 (600μg)는 24cm IPG strip (GE Healthcare, Piscataway, NJ, USA)을 이용하여 등전점 전기영동을 실시하 였다. Rehydration 용액에 6시간 이상 rehydration 시킨 strip 을 이용하여 전기영동을 수행하였다. 전기영동은 50 V 4시간, 100 V 1시간, 500 V 1시간, 1000 V 1시간, 2000 V 1시간, 4000 V 2시간, 8000 V 5시간, 8000 V 9시간, 50 V 6시간 으로 수행하였으며 IPGphore II platform (GE Healthcare, Waukesha, WI, USA)을 사용하였다. 전기영동 후 strip은 equilibration 용액 [30% (v/v) glycerol, 2% (v/v) sodium dodecyl sulfate (SDS), 50 mM Tris-HCI (pH 6.8), 100 mM DTT, 6 M Urea, 0.1mg/mℓ bromophenol blue]에 20분간 equilibration 과정을 거친 뒤 55 mM iodoacetamide (Amresco, Solon, OH, USA)를 첨가한 후 암실에서 20분간 한번 더 equilibration을 수행하였다. Strip은 12% SDS-PAGE gel 위에 수평으로 올려놓고 전기영동을 수행하였다. 전기영동은 gel 당 2 W로, 500 V, 300 mA 에서 30분을 걸어 준 뒤, gel 당 16 W에서 700 V, 300mA로 loading dye가 gel 아래 0.5cm 로 내려갈 때까지 걸어 주었다. Gel은 colloidal Coomassie Briliant Blue (CBB) 염색 용액 [34% (v/v) methanol, 17% (w/v) ammonium sulfate, 3% (v/v) phosphoric acid, 0.1% (w/v) CBB G-250]으로 염색하였다.
5. 이미지 분석
2-DE gel은 transmissive scanner (PowerLook 1120, UMAX) 를 사용하여 스캔 후 Image Master 2D Platinum software 6.0 (GE Healthcare, Waukesha, WI, USA)을 이용하여 분석 되었다. 분석 과정에는 spot 판별 과정과 부피 측정이 포함되 어 있다. 첫 번째로 염류 스트레스 처리 시간마다 (0, 6, 12, 18시간) 양적 변화를 보이는 spot을 판별 한 후 각 spot의 volume 값을 측정하였다.
6. 단백질 동정
Kim 등 (2004b)에서 보고된 바와 같이 2-DE gel에서 In-gel tryptic digestion을 수행한 뒤 MALDI-TOF/TOF MS 분석을 수 행하였다. Gel에서 colloidal CBB로 염색된 protein spot을 잘 라내어 50% (v/v) acetonitrile (ACN)이 포함된 0.1 M NH4 HCO3에 세척하여 SpeedVac Concentrator (Thermoscientific, Bremen, Germany)으로 건조시켰다. 건조된 gel 조각에 10mM DTT이 포함된 0.1 M NH4HCO3을 처리한 후 55°C에서 45분 간 환원시킨 후 원심분리로 상등액을 제거하고 55 mM iodoacetamide이 포함된 0.1 M NH4HCO3을 처리하여 암실에 서 30분간 알킬화 반응을 시켰다. Gel 조각을 50% ACN이 포함된 0.1 M NH4HCO3로 세척 작업을 수행한 뒤 digesting solution [25 mM NH4HCO3, 0.1% N-octylglucoside(w/v), and 12.5 ng/μℓ trypsin]을 처리하여 37°C에서 over night하여 digestion 시킨 후 건조시켰다. 단백질 동정은 ABI 4800 Plus TOF-TOF Mass spectrometer (Applied Biosystems, Framingham, MA, USA)를 이용하여 355nm에서 200 Hz ND : YAG laser를 가동시켰다. 모든 데이터는 4700 Proteomics analyzer를 위한 Mass Standard Kit (Applied Biosystems, Framingham, MA, USA)를 이용하여 분석되었다. MS/MS spectra는 database 검색 프로그램인 MASCOT에서 NCBI database를 바탕으로 검색하였으며 검색 척도에 대해서 experimental peptide mass value에 대한 오차를 ± 50 ppm으 로 정하고 methionine oxidation과 carboxyamidomethylation 을 variable modification으로 지정하였으며 miscleavage는 1로 지정하여 검색하였다.
LC-MS/MS는 LTQ-Orbitrap system (Thermo Fisher Scientific, Waltham, MA, USA)을 이용하여 Takahashi 등 (2014)의 방법에 따라서 분석하였다. LC-MS/MS 분석에서 얻 은 Raw data post-MS는 Proteome Discoverer 1.3 software (Thermo Fisher Scientific)을 이용하여 재가공하여 NCBI database를 바탕으로 검색하고 또한 인삼 RNAseq 분석에서 얻은 단백질정보를 바탕으로 검색을 실시하였다.
결과 및 고찰
1. 염류 저항성 ‘천량’ 품종
본 실험에서 사용된 인삼 품종은 농촌진흥청 국립원예특작 과학원 인삼과에서 육성한 염류 저항성 품종인 ‘천량’이다 (Kim et al., 2013). 초기 인삼 염류 처리 시간 별로 발현 정 도에 차이가 나는 단백질을 비교하기 위하여 ‘천량’ 품종을 0 시간, 6시간, 12시간, 18시간 동안 염류 농도 5 ds/m, 염류 조 성은 21 mM KNO3, 6.1 mM KH2PO4, 6.2 mM Mg SO4 • 7H2O의 혼합 용액에 인삼 잎이 붙어 있는 잎자루를 꽂 아 염류 처리 실험을 수행하였다. 또한 염류 처리에 따른 식물 체의 반응을 엽록체 형광 측정을 통하여 분석하였고, 형광 반 응이 보이지 않는 인삼 잎을 단백질체 분석에 사용하였다. 결 과적으로 ‘천량’에서는 염류 과다에 의해서 발생되는 지상부 의 황증 현상과 형광반응 증상이 거의 나타나지 않았다.
2. 초기 염류 스트레스 반응 단백질 2-DE 분석
초기 시간 별 인삼에서 수확한 잎에서 TCA/Acetone 추출 법을 이용하여 단백질을 추출하였다. 추출한 단백질로 이차원 전기영동을 실시한 후 colloidal CBB 염색법으로 각 gel을 염 색하였고 총 4번의 반복 실험을 수행하였다. 그 중 초기 시간 대 별로 발현의 차이를 보이는 단백질 spot들을 관찰할 수 있 었다. 염류 처리 시간 별로 총 1003 ± 240 (0시간), 992 ± 166 (6시간), 1051 ± 51 (12시간), 990 ± 160 (18시간)개의 spot들이 관찰 되었고, 각 spot의 발현 차이를 one-way ANOVA 검정 을 통하여 95% 이상의 유의 수준을 보이는 단백질을 찾았다 (Fig. 1). 총 4번 반복 실험된 이차원 전기영동 gel을 Image Master software를 이용하여 시간 별로 비교.
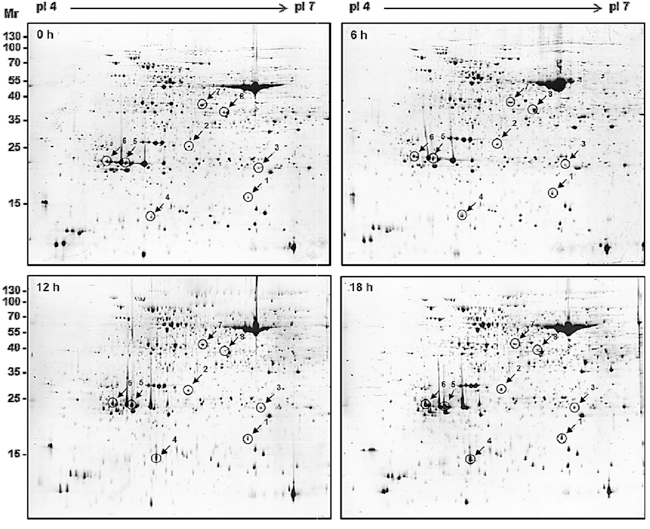
Representative 2-DE gel profiles of ginseng leaves proteins isolated from exposed to salt stress. For 2-DE, 600 μg of proteins were resolved on IPG strips (4-7, 24 cm). Second dimension separation was carried out on 12.5% SDS-PAGE. Gels were stained with colloidal CBB G250.
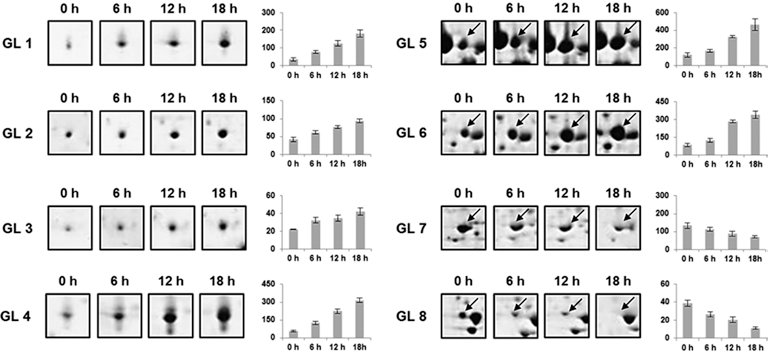
한 결과 8개의 단백질이 발현에 차이를 보였다. 단백질 spot GL 1, GL 2, GL 3, GL 4, GL 5, GL 6은 염류 처리 시간 이 증가할수록 점점 발현량이 증가하는 단백질이고, spot GL 7, GL 8은 시간이 증가할수록 발현량이 감소하는 단백질이다 (Fig. 2). 이들 단백질들은 인삼 초기 염류 스트레스 반응에 관여하는 지표 단백질일 것으로 예상이 된다.
3. 초기 염류 스트레스 반응 단백질 동정
시간대 별로 발현량의 변화를 보이는 단백질 spot들을 gel에 서 잘라내어 In-gel tryptic digestion 과정을 거쳐 MALDITOF/ TOF MS로 분석을 실시하고 NCBI database를 기반으로 한 MASCOT 검색 engine을 이용하여 단백질을 동정하였다 (Table 1). 동정한 결과, ethylene response sensor-1 (spot GL 1), carbonic anhydrase-1 (spot GL 3), chlorophyll a/b binding protein (spot GL 5, GL 6), RuBisCO activase B (spot GL 7), DNA helicase (spot GL 8)들을 동정하였다. 단백질 spot GL 2와 GL 4은 MALDI-TOF/TOF MS로 동 정되지 않아 LC-MS/MS로 다시 동정하였다. 동정한 결과, nucleotide binding protein (spot GL 2)과 thylakoid lumenal 17.9 kDa protein (spot GL 4)를 동정하였다 (Table 2).
4. 초기 염류 장애 관련 단백질
‘천량’은 염류 생리장해 저항성 품종 육성을 목적으로 2003 년부터 2011 년까지 특성검정, 생산력검정시험, 지역적응시험 을 실시한 결과, 다수성이면서 염류 저항성이 강한 품종으로 2011 년에 직무육성 신품종 선정위원회에서 신규 등재 품종으 로 인정받았다 (Kim et al., 2013). 그러나 ‘천량’ 품종에 대 한 염류 스트레스 및 저항성에 대한 단백질 바이오마커 개발 을 위하여 단백질체 분석을 시행한 결과는 없다.
본 연구는 염류 저항성 인삼 품종인 ‘천량’을 대상으로 초 기 염류 반응에 관여하는 단백질 발굴을 위하여 6시간부터 최 대 18시간 동안 염류 처리를 하였다는 점에서 이전의 연구 (Kim et al., 2008)와 다르다고 할 수 있다. 결론적으로, 초기 염류 저항성 관련 지표 단백질을 MALDI-TOF/TOF MS와 LC-MS/MS를 이용하여 동정하였다 (Table 1, Table 2). 그 결과 광합성에 관련된 단백질인 thylakoid lumenal 17.9 kDa protein (spot GL 4), chlorophyll a/b binding protein (spot GL 5, GL 6), RuBisCo activase B (spot GL 7) 등이 동정 되었다. 이들 광합성 관련 단백질들은 일반적으로 인삼뿐만이 아니라 여러 식물이 환경적 스트레스에 노출되었을 때에 반응 하는 단백질들로 잘 알려져 있다 (Aghaei and Komatsu, 2013; Kim et al., 2014). 염류 생리장해 관련 단백질체 연구 는 인삼 염류 감수성 계통 STG3134와 저항성 계통 STG3159 에 염류 처리하여 광합성에 관련된 단백질 (Oxygen-evolving enhancer ptotein 1과 2, RuBisCo, RuBisCo activase) 등이 염류 스트레스에 관련된 단백질들로 동정되었다 (Kim et al., 2008). 따라서 광합성 관련 단백질들의 염류 장해에 대한 직 접적인 연관이 있을 것으로 추정된다.
Spot GL 1은 인삼 ethylene response sensor-1 (ERS1) 단 백질로 동정이 되었다. 식물에서 ethylene 신호 전달은 식물의 성장, 발생 그리고 생리, 과일의 성숙, 줄기와 뿌리의 성장 억 제, 발아와 개화 촉진, 잎의 노화 등 분화 발달 관련성뿐만이 아니라 염류 스트레스와도 밀접한 관련이 있다는 보고는 오래 전부터 잘 알려진 사실이다 (Guo and Ecker, 2004; Cao et al., 2007). ERS1 유전자는 ethylene 수용체인 ETR1에 대한 유사성에 근거하여 확인되었으며, 이 유전자는 dominant ethylene insensitivity를 보여 주는 것으로 아마 ERS1은 ethylene 반응의 양성 조절자로 추정이 된다. 그러나 아직까지 인삼에서 ethylene 관련 단백질이 염류 스트레스와 관련된 보 고는 없다.
Spot GL 3은 carbonic anhydrase로 동정되었다. 이 효소는 CO2와 물을 bicarbonate로 전환시켜 주는 역할을 하는 것으로 잘 알려져 있다. 흥미롭게도 이 효소는 고농도의 NaCl 용액에 서 저항성을 보이는 녹색 조류의 일종인 Dunaliella salina의 막 단백질인 carbonic anhydrase의 증가에 의해서 고농도의 염 용액에서 살 수 있는 것으로 보고되었다 (Fisher et al., 1996). 또한 고추 병저항성과 삼투 스트레스 내성 관련 유전자 의 기능을 규명하는 연구 결과에서 식물에서 과민성 세포 사 멸 (hypersensitive cell death)의 포지티브 조절인자로서 고추 과민성 유도 반응 단백질인 capsicum annuum hypersensitive induced reaction protein-1 (CaHIR1)이 과발현된 형질전환체에 서 삼투 스트레스에 의해서 carbonic anhydrase의 발현이 상 향 조절되는 결과를 보고하였다 (Jung et al., 2008). 따라서 carbonic anhydrase는 염류 저항성에 밀접한 연관관계가 있는 것으로 추정되며 인삼에서 염류 저항성에 중요한 역할을 할 것으로 예상이 된다.
Spot GL 8은 DNA helicase로 이 단백질은 이중나선 구조 로 꼬여 있는 DNA를 풀어 주는 역할을 하는 것으로 잘 알 려져 있지만 이 단백질은 식물의 환경적 스트레스에 민감하게 반응하는 것으로도 알려져 있다. 최근에 완두의 DNA helicase 45 유전자가 형질전환된 벼의 다수확 품종인 IR64가 염류에 대한 저항성을 보이는 연구 결과를 발표하였다 (Sahoo et al., 2012). 따라서 GL 8도 인삼 염류 저항성과 밀접한 관련이 있 을 것으로 추정된다.
감사의 글
본 연구는 농촌진흥청 FTA 대응 경쟁력향상 기술개발사 업(PJ01010401)의 지원을 받아 수행된 결과로 이에 감사드립 니다.
REFERENCES
-
Aghaei, K, Komatsu, S, Crop and medicinal plants proteomics in response to salt stress, Frontiers in Plant Science, (2013), 4, p8, doi: 10.3389/fpls.2013.00008.
[https://doi.org/10.3389/fpls.2013.00008]

- Attele, AS, Wu, JA, Yuan, CS, Ginseng pharmacology-Multiple constituents and multiple actions, Biochemical Pharmacology, (1999), 58, p1685-1693.
-
Bang, KH, Seo, AY, Chung, JW, Kim, YC, Jo, IH, Kim, JU, Kim, DH, Cha, SW, Cho, YG, Kim, HS, Analysis of genetic polymorphism and relationship of korean ginseng cultivars and breeding lines using EST-SSR marker, Korean Journal of Medicinal Crop Science, (2012), 20, p277-285.
[https://doi.org/10.7783/kjmcs.2012.20.4.277]

- Cao, WH, Liu, J, He, XJ, Mu, RL, Zhou, HL, Chen, SY, Zhang, JS, Modulation of ethylene responses affects plant salt-stress responses, Plant Physiology, (2007), 143, p707-719.
-
Fisher, M, Gokhman, I, Pick, U, Zamir, A, A salt-resistant plasma membrane carbonic anhydrase is induced by salt inDunaliella salina, The Journal of Biological Chemistry, (1996), 271, p17718-17723.
[https://doi.org/10.1074/jbc.271.30.17718]

-
Guo, H, Ecker, JT, The ethylene signaling pathway: new insights, Current Opinion in Plant Biology, (2004), 7, p40-49.
[https://doi.org/10.1016/j.pbi.2003.11.011]

- Jung, HW, Lim, CW, Lee, SC, Choi, HW, Hwang, CH, Hwang, BK, Distinct roles of the popper hypersensitive induced reaction protein gene CaHIR1 in disease and osmotic stress, as determined by comparative transcriptome and proteome analyses, Planta, (2008), 227, p409-425.
-
Jung, JD, Park, HW, Hahn, Y, Hur, CG, in, DS, Chung, HJ, Liu, JR, Choi, DW, Discovery of genes for ginsenoside biosynthesis by analysis of ginseng expressed sequence tag, Plant Cell Reports, (2003), 22, p224-230.
[https://doi.org/10.1007/s00299-003-0678-6]

-
Kim, GH, Seong, BJ, Kim, SI, Han, SH, Kim, HH, Lee, KS, Yield and quality characteristics of ginseng's first byproducts, Korean Journal of Medicinal Crop Science, (2011), 19, p313-318.
[https://doi.org/10.7783/kjmcs.2011.19.5.313]

-
Kim, SI, Kim, JY, Kim, EA, Kwon, KH, Kim, KW, Cho, K, Lee, JH, Nam, MH, Yang, DC, Yoo, JS, Park, YM, Proteome analysis of hairy root fromPanax ginsengC. A. Meyer using peptide fingerprinting, internal sequencing and expressed sequence tag data, Proteomics, (2003), 3, p2379-2392.
[https://doi.org/10.1002/pmic.200300619]

- Kim, SI, Kweon, SM, Kim, EA, Kim, JY, Kim, S, Yoo, JS, Park, YM, Characterization of RNase-like major storage protein from the ginseng root by proteomic approach, Journal of Plant Physiology, (2004a), 161, p837-845.
-
Kim, ST, Bae, DW, Lee, KH, Hwang, JE, Bang, KH, Kim, YC, Kim, OT, Yoo, NH, Kang, KY, Hyun, DY, Lim, CO, Proteomic analysis of Korean ginseng(Panax ginsengC. A. Meyer) following exposure to salt stress, Journal of Plant Biotechnology, (2008), 35, p185-193.
[https://doi.org/10.5010/jpb.2008.35.3.185]

-
Kim, ST, Kim, SG, Agrawal, GK, Kikuchi, S, Rakwal, R, Rice proteomics: A model system for crop improvement and food security, Proteomics, (2014), 14, p4-5.
[https://doi.org/10.1002/pmic.201300388]

- Kim, ST, Kim, SG, Hwang, DH, Kang, SY, Kim, HJ, Lee, BH, Lee, JJ, Kang, KY, Proteomic analysis of pathogenresponsive proteins from rice leaves induced by rice blast fungus,Magnaporthe grisea, Proteomics, (2004b), 4, p3569-3578.
-
Kim, UG, Jung, HJ, Lee, SJ, Kim, ST, Crop proteomics: Practical method for high resolution of two-dimensional electrophoresis, Journal of Plant Biotechnology, (2012), 39, p81-92.
[https://doi.org/10.5010/jpb.2012.39.1.081]

-
Kim, YC, Kim, DH, Bang, KH, Kim, JU, Hyun, DY, Lee, SW, Kang, SW, Cha, SW, Kim, KH, Choi, JK, Han, SH, An, YN, Jeong, HN, A high yielding and salt resistance ginseng variety ‘Cheonryang’, The Korean Journal of Breeding Science, (2013), 45, p434-439.
[https://doi.org/10.9787/kjbs.2013.45.4.434]

-
Ma, R, Sun, LW, Chen, XN, Jiang, R, Sun, H, Zhao, DQ, Proteomic changes in different growth periods of ginseng roots, Plant Physiology and Biochemistry, (2013), 67, p20-32.
[https://doi.org/10.1016/j.plaphy.2013.02.023]

- Nagappan, A, Karunanithi, N, Sentrayaperumal, S, Park, KI, Park, HS, Lee, DH, Kang, SR, Kim, JA, Senthil, K, Natesan, S, Muthurajam, R, Kim, GS, Comparative root protein profiles of Korean ginseng(Panax ginseng) and indian ginseng(Withania sonifera), The American Journal of Chinese Medicine, (2012), 40, p203-218.
- Nam, MH, Kim, SI, Liu, Jr, Yang, DC, Lim, YP, Kwon, KH, Yoo, JS, Park, YM, Proteomic analysis of Korean ginseng(Panax ginsengC. A. Meyer), Journal of Chromatography. B, Analytical Technologies in the Biomedical and Life Science, (2005), 815, p147-155.
-
Pandey, A, Mann, M, Proteomics to study genes and genomes, Nature, (2000), 405, p837-846.
[https://doi.org/10.1038/35015709]

-
Rabilloud, T, Two-dimensional gel electrophoresis on proteomics: old, old fashioned, but it still climbs up the mountains, Proteomics, (2002), 2, p3-10.
[https://doi.org/10.1002/1615-9861(200201)2:1<3::aid-prot3>3.0.co;2-r]

-
Sahoo, RK, Gill, SS, Tuteja, N, Pea DNA helicase 45 promotes salinity stress tolerance in IR64 rice with improved yield, Plant Signaling & Behavior, (2012), 7, p1042-1046.
[https://doi.org/10.4161/psb.20915]

- Sun, LW, Ma, PT, Li, DN, Lei, XJ, Ma, R, Qi, C, Protein extraction from the stem ofPanax ginsengC. A. Meyer A tissue of lower protein extraction efficiency for proteomic analysis, African Journal of Biotechnology, (2011), 10, p4328-4333.
- Takahashi, D, Li, B, Nakayama, T, Kawamura, Y, Uemura, M, Shotgun proteomics of plant plasma membrane and microdomain proteins using nano-LC-MS/MS, Methods in Molecular Biology, (2014), 1072, p481-498.
-
We, JS, Park, HS, Kwon, KR, Proteome analysis of various types ofPanax ginsengusing 2-dimensional electrophoresis, Journal of Pharmacopuncture, (2007), 10, p5-18.
[https://doi.org/10.3831/kpi.2007.10.2.005]