
고려인삼 신품종 ‘천량’ 특이적 DNA 판별 마커 개발
†Corresponding Author : Corresponding author: (Phone) +82-43-871-5534 bang31@korea.kr
© The Korean Society of Medicinal Crop Science. All rights reserved
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License ( http://creativecommons.org/licenses/by-nc/3.0 ) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
This study describes the efficient method for the discrimination of ‘Cheonryang’ in Panax ginseng Meyer using a STS primer. A total of 208 STS primers were applied to polymerase chain reaction (PCR) amplification for discriminating Korean ginseng cultivars. Co-dominant polymorphic band patterns were generated with two primers, MFGp 0019, MFGp 0248, and successful identification of ‘Cheonryang’ was achieved from out of 11 Korean ginseng cultivars. Two different sizes of DNA band patterns were detected with MFGp 0019 primer. Ten Korean ginseng cultivars shared the same size of amplified DNAs (389 bp), but ‘Cheonryang’ showed a different size. Thus ‘Cheonryang’ can be efficiently distinguished from the other ten ginseng cultivars by using the MFGp 0019 primer. In the case of MFGp 0248, two different sizes of DNA band patterns were detected in the eleven ginseng cultivars. Same sized amplified DNA bands (307 bp) were shown in five cultivars (Chunpoong, Gopoong, Kumpoong, Cheongsun, Sunhyang) and 254 bp sized DNA bands were identified in the other 6 cultivars (Yunpoong, Sunpoong, Sunun, Sunone, Cheonryang, K-1). In conclusion, the two STS primers, MFGp 0019, and MFGp 0248, provide a rapid and reliable method for the specific identification of ‘Cheonryang’ cultivar from a large number of samples.
Keywords:
Panax ginseng Cultivar, Insertion and Deletion, Sequence Tagged Site, Genomic DNA Library서 언
최근 지구 온난화 등의 기후변화와 신규 병해충 발생으로 인한 환경 피해가 심각한 문제로 제기되고 있어, 이에 따른 능동적인 대처 방안 마련이 농업분야의 커다란 화두로 부각되 고 있다. 국내에서도 인삼을 재배하는데 있어 이상 기후에 따 른 피해가 많이 발생하고 있는데, 특히 인삼은 고온과 염류 집적으로 인한 피해가 심각한 실정이다. 따라서 이러한 고온 과 염류 장해 등의 문제를 해결하기 위한 방안의 하나로 인삼 육종분야에서 환경 저항성 인삼 신품종 개발이 이루어지고 있 다 (Kim et al., 2013).
최근 국내 연구진에 의해 염류에 강하고, 다수성의 특성을 을 지닌 인삼 신품종 ‘천량’이 개발되었다. 천량 품종개발을 위해 1999년도 여주 지방의 농가포장에서 인삼자원을 수집하 였으며, 2002년 3년생 개체 선발 후, 2003년부터 2006년까지 특성검정 및 증식을 수행 하였다. 또한 천량 품종은 2007년부 터 2009년도까지 생산력검정시험을 통하여 음성1호로 계통명 을 부여한 후 2010년부터 2011년까지 지역적응시험을 거쳐 개발되었다. 천량은 기존 품종인 천풍에 비해 수량이 10% 정 도 많고, 염류에 대한 저항성이 강하며, 고온에도 잘 견디는 등 품질이 우수하여 기후변화에도 적응력이 높은 것으로 보고 되었다. 또한 기억력과 학습기능 증진에 관여하는 것으로 알 려진 Rg1과 Rg2 등의 사포닌 함량이 높은 특성이 있다 (Kim et al., 2013). 줄기는 연한 자색이고 열매는 적색이며, 잎이 아래로 뒤집혀 볼록한 형태를 나타내고 있어 다른 품종과 구 분되는 특징이 있다.
한편, 신품종이 개발되면 이를 법적 • 제도적으로 보호받을 수 있어야 육종가의 지적소유권이 보장되고, 궁극적으로 우량 품종 종자의 보급을 촉진시켜 농가의 생산성과 소득을 증대시 킬 수 있다. 최근 종자산업법에서는 육종가의 권리를 보호하 기 위하여 신품종 개발 시 기존 품종과의 차별성을 제시하도 록 하고 있으며, 국제식물보호품종연맹 (UPOV)에서도 신품종 의 구별성 검정에 DNA 마커 활용을 적극 검토하고 있다 (Kim et al., 2011).
최근 유럽을 중심으로 벼 (Komori and Nitta, 2004; Sun et al., 2009), 오이 (Bernet et al., 2003), 배추 (Kwon et al., 2003), 밀과 토마토 (Cooke et al., 2003; Noil et al., 2008), 고추 (Lefebvre et al., 2001) 등을 대상으로 다양한 DNA 마커를 적용하여 이들 작물로부터 유래된 품종의 구별 성 검정과 이들 연구결과의 품종 보호 적용 가능성에 대한 논 의가 이루어져왔다. 한편, 개발된 신품종이 대내외적으로 품종 등록과 보호를 받기 위해서는 구별성 (Distinctness), 균일성 (Uniformity), 안정성 (Stability)의 3가지 기술적 요건과 신규성 (Novelty), 품종명칭 (Denomination)의 2가지 방식적 요건을 갖추어야 한다.
국내에서 인삼을 포함한 작물의 DUS (Distinctness, Uniformity, Stability) 검정 시 일반적으로 육종포장에서 대조 품종과의 생육 특성을 비교하는데, 분석 시 많은 시간이 소요 되는 단점이 있다. 따라서 이들 특성조사를 보완할 수 있는 방법으로 DNA 마커를 이용하여 빠른 시간 내에 품종의 구별 성과 균일성을 검정하는 방법의 개발이 필요성이 제기되었다 (Bang et al., 2011). PCR 기반의 DNA 분석은 비교적 적은 비용으로 다양한 유전자 좌위를 포함하는 다수의 마커를 이용 할 수 있는 장점이 있기 때문에 작물의 종 간 및 품종 간 유 전적 변이를 검정하는데 널리 이용되어 왔다. 따라서 품종 특 이적인 염기서열을 확보하여 DNA 마커로 전환시켜 신품종 보호와 종자보증을 위한 과학적인 지표로의 활용성을 높이는 것이 필요하리라 판단된다.
DNA 마커는 유전현상의 본질인 DNA의 염기서열 차이를 대상으로 하기 때문에 개체 간 변이를 찾는 것이 쉽고, 식물 의 생장과 상관없이 모든 조직에서 안정적으로 탐색할 수 있 는 장점이 있다. 또한 환경에 영향을 받지 않고, 유전자간의 상위 작용이나 다면 발현에 의한 영향을 받지 않기 때문에 식 물체 간 형태적인 특징의 차이를 찾는 전통적인 구별 방법에 비해 DNA 수준에서의 정확한 식별이 가능한 장점이 있다 (Jo et al., 2012b).
Olson 등 (1989)은 DNA 라이브러리로부터 클론의 염기서 열을 확보하고 그 정보를 바탕으로 특이적인 프라이머들을 합 성하여 사용하는 기법인 STS-PCR (Sequence tagged sitespolymerase chain reaction) 방법을 개발하였다. STS 마커는 아가로스겔에서 쉽게 결과를 확인 할 수 있고 분석방법이 간 단하여 대량의 유전자원 평가와 품종판별 및 유연관계 분석에 효율적으로 적용 가능하다. 따라서 이들 STS 마커는 인삼 (Bang et al., 2010; Jo et al., 2013a), 콩 (Hwang et al., 2012), 보리 (Talbert et al., 1994; Blake et al., 1996; Erpelding et al., 1996), 무궁화 (Lee et al., 2002), 델피늄 (Li et al., 2002) 등의 품종 판별과 유전적 다양성 분석에 널 리 활용되었다. 한편 Bang 등 (2010)은 고려인삼 6품종을 대 상으로 208쌍의 STS primer를 적용하여 이들을 구분할 수 있 는 8종의 STS marker를 개발하여 보고한바 있다.
본 연구는 향후 중국, 미국, 캐나다 등 인삼 산업 경쟁국과 의 FTA 체결로 인한 시장개방과 최근 문제가 되고 있는 인삼 신품종의 해외 불법 유출에 대비하기 위해 수행되었다. 또한 국내 국가연구기관인 농촌진흥청에서 최초로 개발된 인삼 신 품종 천량의 유전적 구별성과 균일성을 확보하고, 더 나아가 신품종의 대내외 지적재산권을 행사하기 위한 수단으로 이용 하고자 천량 품종에 특이적인 DNA 마커를 개발하였다.
재료 및 방법
1. 시험재료
본 연구에서 사용한 재료는 충청북도 음성군 소재 농촌진흥 청 국립원예특작과학원 인삼특작부 시험연구 포장에서 재배중 인 천풍, 연풍, 고풍, 금풍, 선풍, 선운, 선원, 청선, 선향, K- 1, 천량 등 11품종을 대상으로 하였으며, 인삼의 연근 (年根) 은 4년생을 기준으로 하였다 (Table 1). 인삼 재배, 병해충 방 제 및 기타 관리는 인삼 GAP 표준재배지침서에 준하였다 (RDA, 2009). DNA 추출은 샘플을 액체질소로 급랭시켜 막자 사발을 이용하여 분말상태가 되도록 마쇄한 후, DNeasy Plant Mini Kit (Qiagen, Hilden, Germany)을 이용하여 제작사가 제공한 protocol에 따라 추출하였다.
2. DNA 추출 및 정량
인삼 식물체로부터 genomic DNA를 확보하기 위하여 4년 생 잎을 채취하여 세척한 후 암실냉장 조건에서 하루 동안 보 관하였다. 이후 액체질소로 급랭시켜 막자사발과 유봉을 이용 하여 고운 분말 상태가 되도록 마쇄한 후, 분말 상태의 시료 를 2㎖ 튜브에 10㎎ 넣고, 68℃로 미리 가열된 extraction buffer type-I 용액 300㎕ (50 mM Tris, 25 mM EDTA 및 0.35 M sorbitol), 5% sarcosyl 용액 50㎕, 5M NaCl 용액 60㎕ 및 CTAB (8.6%) 35㎕를 넣어, 68℃ 조건의 항온 수 조에 30분 동안 처리하면서 시료가 뭉치지 않도록 5분 간격 으로 흔들어 주었다.1
다음 과정은 PIC (phenol 25 : chloroform 24 : isoamylalchohol 1) 용액을 600㎕ 첨가하여 시료가 잘 섞이도록 1분 이상 흔들 어준 후, 10℃에서 12,000 rpm으로 15분간 원심분리를 하였 고, PIC를 통해 분리된 상층액 450 ~ 500㎕를 새 튜브에 옮 긴 후, 3M NaOAC (pH 5.2) 50㎕, Isopropyl alcohol 600㎕를 각각 넣어 하얀 펠릿이 생성 될 때까지 흔들어준 뒤 −70℃의 초저온 냉장고에 20분간 방치하여 펠릿을 응축시켰다.
응축된 펠릿을 확보하기 위하여 10℃에서 12,000 rpm으로 5분 동안 원심분리 하여 펠릿을 제외한 나머지 용액을 버 린 후, 에탄올 (70%) 600㎕를 넣어 1분 이상 흔들어 주었 으며, 10℃에서 12,000 rpm으로 2분간 원심분리 하여 에탄올 을 버린 후 실온에서 펠릿을 건조하였다. 완전히 건조된 펠릿 은 Tris-EDTA 버퍼를 100㎕ 넣어 실온에서 잘 녹여주었다. 이 러한 과정을 거쳐 추출된 DNA는 Nano Drop 2000 (Thermo Fisher Scientific, Wilmington, Delaware, USA) 기기를 이용하 여 농도를 측정하였으며, 농도 측정이 완료된 각각의 DNA 샘 플들은 멸균된 증류수를 이용하여 최종 DNA 농도를 5 ng/㎕ 로 각각 정량하였다.
3. STS-PCR (Polymerase Chain Reaction)
선행연구에서 methylation filtering (MF) 기법을 활용하여 인삼의 genomic DNA library를 제작하였으며 이들을 해독한 염기서열 정보로부터 216 쌍의 STS 프라이머들을 디자인 하 였다 (Bang et al., 2010). 기존에 개발된 8종의 STS 마커를 제외한 208쌍의 STS 프라이머는 국내인삼 11품종을 대상으로 분석에 사용하였다. PCR 조성은 genomic DNA 10 ng, 20 pmole forward와 reverse primer, 2.5 mM dNTP, 10 × PCR buffer, 1 unit Taq DNA polymerase (Inclone, Korea)를 혼합 하여 총 반응액을 20 μL로 조정하였다. PCR 반응은 BioRad 사의 thermal cycler C-1000 (BIO-RAD, Hercules, CA, USA)를 이용하여 94℃에서 5분간 초기변성 후, 95℃에서 30 초 변성, 55 ~ 65℃에서 40초 결합, 72℃에서 1분간 신장 조 건으로 총 35회 반복한 후 마지막으로 72℃에서 10분 간 신 장반응을 수행하였다. 증폭된 DNA 산물은 1.5% agarose gel 에서 150 Volt로 전기영동한 후, Safe Gel Stain (Inclone, Korea) 으로 염색하여 UV-illuminator (BioRad, Richmond, CA, USA)에서 유전양상을 확인하였다.
4. 인삼 품종 특이적인 프라이머 선발 및 염기서열분석
고려인삼 11품종을 대상으로 208쌍의 STS 프라이머를 적용 하여 품종 간 차이를 나타내는 프라이머를 선발 하였다. 품종 간 다형성을 나타내는 PCR 산물들은 Mega Quick-spin Kit (Intron Bio, Korea)를 사용하여 정제한 후 Genetic Analyzer 3130XL (Applied Biosystems, Foster City, CA, USA)를 이 용하여 염기서열을 분석하였다. 분석된 염기서열은 Bioedit 프 로그램을 이용하여 편집하였고 no gap으로 저장한 후 Cluster X (ver. 1.83)로 염기서열을 정렬하였다.
결과 및 고찰
본 연구는 인삼의 genomic DNA 라이브러리로부터 제작된 208쌍의 STS 프라이머들을 이용하여 천량 품종 특이적인 마 커를 찾고자 수행하였다. 총 208쌍의 프라이머들을 고려인삼 11품종에 적용한 결과, 품종 간 차이를 나타내는 2종의 프라 이머 (MFGp 0019, MFGp 0248)를 선발하였다 (Table 2).
MFGp 0019 프라이머를 이용하여 PCR한 결과, 약 250 ~ 400 bp 사이에서 두 개의 DNA 단편이 증폭되었는데 (Fig. 1A), 이들의 차이는 염기서열의 삽입 또는 결실 (Insertion or Deletion)에 의한 것으로 추측되었다. 이러한 결과를 정확하 게 확인하기 위하여 두 개의 PCR 증폭산물을 대상으로 유전 자클로닝을 수행한 후 이들의 염기서열을 분석한 결과, 천풍 을 포함한 10품종 (천풍, 연풍, 고풍, 금풍, 선풍, 선운, 선원, 청선, 선향, K-1)은 389 bp 크기의 염기서열이 확인되었고, 반 면 천량은 264 bp 크기의 특이적인 염기서열을 확인할 수 있 었는데, 114 ~ 238 bp 위치에서 125 bp의 염기서열 결실이 확 인 되어 천량품종이 다른 고려인삼 10품종과 뚜렷한 차이를 나타내었다 (Fig. 3). 한편 천량 품종 특이적인 MFGp 0019 프라이머의 재현성을 검정하기 위하여 천량 10개체를 대상으 로 PCR을 수행한 결과, 10개체 모두 동일한 크기의 DNA 단편이 확인 되었다 (Fig. 2).
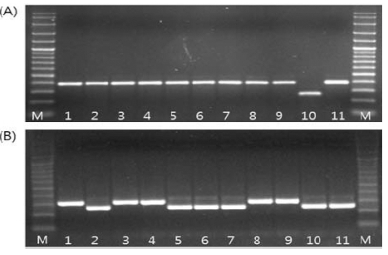
Agarose gel electrophoresis of the PCR products amplified with MFGp 0019(A) and MFGp 0248(B) primers.Lane 1; Chunpoong, Lane 2; Yunpoong, Lane 3; Gopoong, Lane 4; Gumpoong, Lane 5; Sunpoong, Lane 6; Sunun, Lane 7; Sunone, Lane 8; Cheongsun, Lane 9; Sunhyang, Lane 10; Cheonryang, Lane 11; K-1, M; 100 bp DNA ladder (Inclone, Korea).
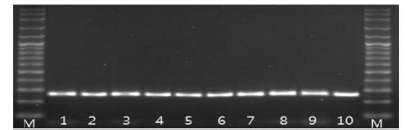
Reproducibility testing using MFGp 0019 primer in Cheonryang cultivar.Lane 1 ~ 10; Cheonryang, M; 100 bp DNA ladder (Inclone, Korea).
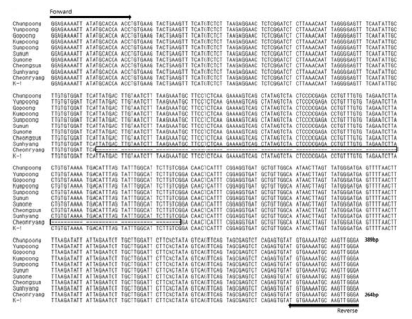
Nucleotide sequence alignment of polymorphic fragments amplified from eleven Korean ginseng cultivars by MFGp 0019 primer.The areas enclosed by the boxes indicates In-Del variations. Primer binding regions and directions were indicated with arrows.
MFGp 0248 프라이머를 이용하여 PCR한 결과, MFGp 0019 프라이머를 이용한 결과와 동일하게 염기서열의 삽입 또 는 결실에 의한 차이를 보였는데, 고려인삼 11품종이 두 개의 유전자형으로 분리 되었다 (Fig. 1B). 증폭된 DNA 단편의 크 기는 약 250 ~ 300 bp 이었으며, 염기서열분석을 수행한 결과 천풍을 포함한 5품종 (천풍, 고풍, 금풍, 청선, 선향)의 염기서 열 길이는 307 bp로 확인 되었다. 반면에 연풍을 포함한 6품 종 (연풍, 선풍, 선운, 선원, 천량, K-1)은 염기서열의 길이가 254 bp로써 861 ~ 38 bp 위치에서 53 bp의 염기 결실이 관찰 되었다 (Fig. 4).

Nucleotide sequence alignment of polymorphic fragments amplified from eleven Korean ginseng cultivars by MFGp 0248 primer.The areas enclosed by the boxes indicates In-Del variations. Primer binding regions and directions were indicated with arrows.
최근 인삼의 신품종 보호와 종자보증을 위한 품종 특이적인 마커 개발에 관련한 연구가 많이 진행되고 있는데, Wang 등 (2010)은 고려인삼 9품종 (천풍, 연풍, 고풍, 선풍, 금풍, 선운, 선원, 청선, 선향)의 미토콘드리아 cytochrome oxidase subunit 2 (cox2) intron II 지역에서 SNP (Single nucleotide polymorphisms)를 바탕으로 천풍 품종 특이적인 마커를 발굴하 였다. Hua 등 (2011)은 고려인삼 9품종 (천풍, 연풍, 고풍, 선 풍, 금풍, 선운, 선원, 청선, 선향)을 대상으로 glyceraldehyde 3-phosphate dehydrogenase (GAPDH) 유전자 부위의 SNP를 타겟으로 연풍 품종 특이적인 마커를 개발하였으며, Wang 등 (2011)은 핵 리보솜 26S DNA의 SNP 변이를 바탕으로 금풍 과 청선 품종 특이적인 마커를 개발하였다. 이러한 결과는 국 내 인삼 품종 간의 유전적 다형성이 아주 낮은 것을 보여주는 사례로, 품종 간 차이를 나타내는 것이 삽입, 결실, 전좌 등의 커다란 수준에서의 변이가 아니라 염기치환 수준에서 나타나 는 다형성이 대부분이라는 것을 나타내는 결과를 의미한다 (Lee et al., 2012).
앞서 서술한 바와 같이 현재까지 고려인삼의 품종 특이적인 마커는 주로 SNP 변이를 바탕으로 개발되어왔다. 그러나 SNP 를 타겟으로 하는 DNA 마커는 multiplex PCR과 같은 복잡 한 과정을 거쳐야 되고 SNP 타겟의 allele specific 프라이머 를 제작해야 한다. SNP allele specific PCR (AS-PCR)은 증 폭 밴드의 유무의 따라 유전자형을 판단하는 우성 마커 (dominant)로써 증폭반응 시 annealing 온도에 따라 비특이적 인 반응이 일어날 확률이 있어 정확한 식별에 어려움도 있다. 반면에 삽입 또는 결실을 기반으로 하는 DNA 마커는 공우성 (co-dominant)을 나타내는 것이 특징으로, 아가로스겔에서 쉽 게 확인할 수 있는 장점이 있어 대량의 유전자원 분석과 품종 판별 등의 목적에 효율적으로 사용이 가능하다. Jo 등 (2013) 은 고려인삼 11품종, 미국삼 및 중국삼을 대상으로 미토콘드 리아 nad1/2-3 지역의 삽입 또는 결실을 기반으로 종 판별용 DNA 마커를 개발한 바 있다. 삽입 및 결실 부위를 타겟으로 DNA 마커를 개발 할 경우 증폭구간의 길이를 조절하여 PCR 반응시간을 최소화 할 수 있으며 종 간 염기서열 길이의 차이 가 발생하기 때문에 샘플의 혼입여부를 확인 할 수 있는 장점 이 있다.
STS 마커를 개발하기 위해서는 많은 경비와 시간이 소요되 는 단점이 있지만, 일단 DNA 마커가 개발되면 이들 마커를 바탕으로 새롭게 육성되는 품종, 외국 도입종, 근연종 및 원연 종 모두에 광범위하게 적용되어질 수 있어서 반영구적으로 사 용할 수 있다는 장점이 있다.
결론적으로 본 연구를 통하여 선발된 2종의 STS 마커 (MFGp 0019, MFGp 0248)는 모두 삽입 또는 결실을 기반으 로 하는 공우성 마커로써, 누구나 손쉽게 이용할 수 있으며 또한 기존에 개발된 8종의 STS 마커와 조합하여 사용할 수 있는 장점이 있다. 특히 MFGp 0019 마커의 경우 염류에 강 하고 수량이 높은 천량 품종의 유전적 구별성과 균일성 확보, 과학적인 종자관리 및 해외 종자 불법 유출과 재래종의 신품 종 둔갑 등 각종 종자사고에 대응할 수 있는 원천 기술로 유 용하게 활용될 수 있을 것으로 판단된다.
감사의 글
본 연구는 농촌진흥청 공동연구사업(과제번호: PJ008395)과 2014년도 박사후 연수과정지원사업에 의하여 이루어진 결과로 이에 감사드립니다.
REFERENCES
- Bang, KH, Lee, JW, Kim, YC, Kim, DH, Lee, EH, Jeung, JU, Construction of genomic DNA library of Korean ginseng(Panax ginsengC A Meyer) and development of sequence-tagged sites, Biological and Pharmaceutical Bulletin, (2010), 33, p1579-1588.
- Bang, KH, Chung, JW, Kim, YC, Lee, JW, Jo, IH, Seo, AY, Kim, OT, Hyun, DY, Kim, DH, Cha, SW, Development of SSR markers for identification of Korean ginseng(Panax ginsengC A Meyer) cultivars, Korean Journal of Medicinal Crop Science, (2011), 19, p185-190.
- Bernet, GP, Bramardi, S, Calvache, D, Carbonell, EA, Asins, MJ, Applicability of molecular markers in the context of the protection of new varieties of cucumber, Plant Breeding, (2003), 122, p146-152.
- Blake, TK, Kadyrzhanova, D, Shepard, KW, Islam, AKMR, Langridge, PL, McDonald, CL, Erpelding, J, Larson, S, Blake, NK, Talbert, LE, STS-PCR markers appropriate for wheat-barley introgression, Theoretical and Applied Genetics, (1996), 93, p826-832.
- Cooke, RJ, Bredemeijer, GMM, Ganal, MW, Peeters, R, Isaac, P, Rendell, S, Jackson, J, Rder, MS, Korzun, V, Wendehake, K, Areshchenkova, T, Dijcks, M, Laborie, D, Bertrand, L, Vosman, B, Assessment of the uniformity of wheat and tomato varieties at DNA microsatellite loci, Euphytica, (2003), 132, p331-341.
-
Erpelding, JE, Blake, NK, Blake, TK, Talbert, LE, Transfer of sequence tagged site PCR markers between wheat and barley, Genome, (1996), 39, p802-810.
[https://doi.org/10.1139/g96-101]

- Hua, S, Wang, H, Kwon, WS, Kim, YJ, In, JG, Yang, DC, A simple and rapid technique for the authentication of the ginseng cultivar, Yunpoong, using an SNP marker in a large sample of ginseng leaves, Gene, (2011), 487, p75-79.
- Hwang, TY, Jung, SM, Lee, SK, Park, HM, Jeong, KH, Lee, YY, Kim, SL, Yun, HT, Lee, JE, Kim, DW, Jung, GH, Kwon, YU, Kim, HS, Kim, YH, Discrimination of 110 Korean soybean cultivars by sequence tagged sites(STS)-CAPS markers, Korean Journal of Breeding Science, (2012), 44, p258-272.
- Jo, IH, Shin, MR, Kim, YC, Lee, SH, Kim, JU, Moon, JY, Noh, BS, Kang, ST, Lee, DJ, Hyun, DY, Kim, DH, Kim, KH, Bang, KH, Discrimination of Korean ginseng cultivars by sequence tagged sites(STS) markers, Korean Journal of Medicinal Crop Science, (2013a), 21, p353-360.
- Jo, IH, Bang, KH, Kim, YC, Kim, JU, Shin, MR, Moon, JY, Noh, BS, Hyun, DY, Kim, DH, Cha, SW, Kim, HS, Analysis of mitochondrial DNA sequence and molecular marker development for identification ofPanaxspecies, Korean Journal of Medicinal Crop Science, (2013b), 21, p91-96.
- Kim, GJ, Song, YH, Gi, GY, Kim, ST, Lee, JH, Han, TH, Application of UPOV data for the analysis of genetic variation in rose cultivars, Korean Journal of Horticultural Science and Technology, (2011), 29, p240-246.
-
Kim, YC, Kim, DH, Bang, KH, Kim, UK, Hyun, DY, Lee, SW, Kang, SW, Cha, SW, Kim, KH, Choi, JK, Han, SH, An, YN, Jeong, HN, A high yielding and salt resistance ginseng variety ‘Cheonryang’, Korean Journal of Breeding Science, (2013), 45, p434-439.
[https://doi.org/10.9787/kjbs.2013.45.4.434]

-
Komori, T, Nitta, N, A simple method to control the seed purity of japonica hybrid rice varieties using PCR-based markers, Plant Breeding, (2004), 123, p549-553.
[https://doi.org/10.1111/j.1439-0523.2004.01029.x]

- Kwon, YS, Moon, JY, Kwon, YS, Park, DY, Yoon, WM, Song, IS, Yi, SI, AFLP analysis for cultivar discrimination in radish and chinese cabbage, Korean Journal of Breeding Science, (2003), 35, p319-328.
- Lee, OR, Kim, MK, Yang, DC, Authentication of medicinal plants by SNP-based multiplex PCR, Methods in Molecular Biology, (2012), 862, p135-147.
- Lee, SJ, Jeung, JU, Cho, SK, Um, BY, Chung, WI, Bae, JM, Shin, JS, Diversity and varietal classification ofHibiscus syriacusL with the heterogeneity within retrotransposon-like elements, Molecules and Cells, (2002), 13, p362-368.
-
Lefebvre, V, Goffinet, B, Chauvet, JC, Caromel, B, Signoret, P, Brand, R, Palloix, A, Evaluation of genetic distances between pepper inbred lines for cultivar protection purposes Comparison of AFLP, RAPD and phenotypic data, Theoretical and Applied Genetics, (2001), 102, p741-750.
[https://doi.org/10.1007/s001220051705]

-
Li, X, Gardner, DR, Ralphs, MH, Wang, RRC, Development of STS and CAPS markers for identification of three tall larkspurs(Delphinium spp), Genome, (2002), 45, p229-235.
[https://doi.org/10.1139/g01-149]

- Noil, E, Teriaca, MS, Sanguineti, MC, Conti, S, Utilization of SSR and AFLP markers for the assessment of distinctness in durum wheat, Molecular Breeding, (2008), 22, p301-313.
-
Olson, M, Hood, L, Cantor, C, Botstein, D, A common language for physical mapping of the human genome, Science, (1989), 245, p1434-1435.
[https://doi.org/10.1126/science.2781285]

- Rural Development Administration(RDA, Standard cultivation guidebook for good agricultural practice, (2009), Suwon, Korea, Rural Development Administration, p47-117.
- Sun, MM, Choi, KJ, Kim, HS, Song, BH, Woo, SH, Lee, CW, Jong, SK, Cho, YG, Genetic diversity and discrimination of recently distributed Korean cultivars by SSR markers, Korean Journal of Breeding Science, (2009), 41, p115-125.
-
Talbert, LE, Blake, NK, Chee, PW, Blake, TK, Magyar, GM, Evaluation of “sequence-tagged-site” PCR products as molecular markers in wheat, Theoretical and Applied Genetics, (1994), 87, p789-794.
[https://doi.org/10.1007/bf00221130]

-
Wang, H, Hua, S, Kwon, WS, Jin, H, Yang, DC, A PCR-based SNP marker for specific authentication of Korean ginseng(Panax ginseng) cultivar “Chunpoong”, Molecular Biology Reports, (2010), 37, p1053-1057.
[https://doi.org/10.1007/s11033-009-9827-5]

-
Wang, H, Kim, MK, Kwon, WS, Jin, H, Liang, Z, Yang, DC, Molecular authentication ofPanax ginsengand ginseng products using robust SNP markers in ribosomal external transcribed spacer region, Journal of Pharmaceutical and Biomedical Analysis, (2011), 55, p972-976.
[https://doi.org/10.1016/j.jpba.2011.03.037]

