한국산 둥굴레속 식물의 형태적 특성 및 엽록체 DNA 염기서열을 이용한 유연관계 분석
© The Korean Society of Medicinal Crop Science. All rights reserved
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License ( http://creativecommons.org/licenses/by-nc/3.0 ) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Polygonatum is a genus placed in the family Liliaceae, distributed throughout the Northern Hemisphere and 16 of the species are grown naturally in Korea. In oriental medicine, the rhizomes of Polygonatum have been used as two different medicines, Okjuk (Polygonati odorati Rhizoma) and Hwangjeong (Polygonati Rhizoma). However, it is difficult to identify the morphological and chemical differences between the medicinal groups and thus easy to confuse the one with the other. Therefore, a clear classification standard needs to be established so as to be able to discriminate between them. In the study, the morphological characteristics of the plants, Polygonatum spp., were examined. Then, the differences in SNPs among the DNA sequences of 7 of the Polygonatum spp. and 1 of the Disporum spp. were analyzed by DNA barcoding with rpoC1, rpoB2, matK, and psbA-trnH of the cpDNA region. In the results, three regions, rpoC1, rpoB2, and matK were useful for discriminating the species, P. stenophyllum and P. sibiricum.Furthermore, it was possible to discriminate the individual germplasm within the species by using the combination of the results obtained from rpoB2, rpoC1, and matK.
Keywords:
DNA Barcoding, Polygonatum, SNPs, rpoC1, rpoB2, matK, psbA-trnH서 언
둥굴레속 (Polygonatum)은 백합과 (Liliaceae)에 속하는 다 년생 초본으로 북미, 유럽 등 전 세계적으로 40여종이 분포하 며, 국내에는 16종이 자생하는 것으로 알려져 있다 (Jang and Kim, 1999). 둥굴레는 동양의 전통 한의학에서 옥죽 (玉竹) 또는 위유 ( )와 황정(黃精)으로 명명되어 이용되어 왔다 (Ahn, 1985). 옥죽과 황정은 둥굴레속 식물의 뿌리줄기로부터 기원된 한약재로서, 옥죽은 혈압강하, 혈관수축 (An et al., 2006), 지질강하 (Seo et al., 2011) 등에, 황정은 항균, 항진 균, 혈압강하 등에 효과가 있다고 알려져 있다 (Kim et al., 1998). 옥죽과 황정의 기원종에 대한 기준은 각 나라마다 다 르며, 그 기준 자체도 명확하지 않다. 대한민국약전에 따르면 국내에서는 둥굴레 (Polygonatum odoratum)와 동속 근연종의 근경을 ‘옥죽 (위유), 층층갈고리둥굴레 (P. sibiricum)와 진황 정 (P. falcatum)의 근경을 ‘황정’으로 기재하고 있다. 옥죽과 황정의 적절한 이용을 위해서는 기원 식물 종을 명확히 확립 할 필요가 있으나, 이처럼 분류 기준이 불분명하고, 찌거나 볶 는 등의 가공과정을 거쳐 다양한 형태로 유통되기 때문에 한 약재의 기원식물을 명확하게 판별하는 데에는 한계가 있다.
따라서 둥굴레속 식물의 종판별을 위해 현재까지 형태학적 특성, 이화학적 성분, 분자생물학적 방법 등 많은 연구가 진행 되어왔다. 형태학적 연구에 있어서 Jang (2002)은 엽서, 포, 줄기의 능각, 지하경, 염색체 수 등 형태적 특성을 기준으로 국내산 둥굴레속 식물을 14종과 2변종, 총 16분류군으로 정리 한 바 있다. 그러나 형태학적 특성은 동일종 내에서도 지역에 따라 다른 경우가 많으며 (Jang and Kim, 1999), 한약재 유 통 시 절단 가공된 상태이므로 식물 원종의 명확한 판별에는 한계가 있다. 또한 Jang 등 (2002)의 둥굴레종 뿌리줄기 성분 분석 연구에서 각 종의 성분이 유사한 결과를 보였으며, 타 약용식물의 성분 또한 재배환경의 영향을 받을 수 있어 이화 학적 판별 역시 한계가 있다 (An and Chung, 2014).
분자생물학적 방법은 형태적 및 이화학적 판별에서의 한계 를 보완하고 종판별 결과의 신뢰도를 향상시켜 종 분류 및 계 통분석에 관한 연구에 흔히 이용된다 (Williams et al., 1990; Moon et al., 2013). 둥굴레속 판별에는 현재까지 RAPD (Lee et al., 2004; Jang et al., 2004), RAMP (Ahn et al., 2006), rDNA를 이용한 염기서열 분석 (Yun et al., 2006) 등 이 적용되었다. 식물 종판별을 위한 유전적 다양성분석을 위 해 최근 DNA barcoding 기술이 제안되어 그 유용성이 인정 되었다 (Hebert et al., 2003; CBOL Plant Working Group, 2009). DNA barcoding은 각각 다른 생물 종 유전체에서 유래된 동일 유전자 부위의 SNPs (Single Nucleotide Polymorphism) 다형성 분석으로 생물 종을 식별하는 표준화된 방법이다. 동물 종판별에는 미토콘드리아 DNA의 CO1 (cytochrome c oxydase subunit 1) 분석이 유용하지만, 식물 종판별의 경우 표준화된 유전자 부위가 없어 엽록체 DNA의 coding region인 matK, rbcL, rpoB, rpoC1와 non-coding region인 atpF-atpH, psbK-psbI. psbA-trnH spacer 등의 이용 이 제시되고 있다 (CBOL Plant Working Group, 2009). DNA barcoding 기술은 다량의 생물 종을 표준화된 유전자 부위로 동시에 분석할 수 있다. 또한 식물의 형태적 특성 및 유효성 분 분석에 의한 식물 종판별이 불가능한 경우 이를 보완할 수 있으며, 종간차이는 물론 종내 변이 규명에도 이용이 가능하 다 (Li et al., 2011). 따라서 이 기술은 분류기준이 명확하지 않은 둥굴레속 식물자원의 종판별은 물론 이들의 가공 약재인 옥죽과 황정의 원재료 식물 판별에 유용할 것으로 판단된다.
본 연구는 둥굴레속 식물종간 외부 형태적 특성 비교는 물론, 엽록체 DNA 구간 내 바코드 후보 유전자인 rpoC1, rpoB2, matK, psbA-trnH 각 4개의 구간으로부터 발견된 SNPs 다형성을 분석함으로써 분자생물학적 측면에서의 둥굴 레속 식물의 종 판별법을 모색하고자 수행되었다.
재료 및 방법
1. 공시재료
본 연구를 위해 일산 소재 동국대학교 실험농장에서 보유증 식 중인 둥굴레 (Polygonatum odoratum var. pluriflorum), 무늬둥굴레 (P. odoratum var. pluriflorum f. variegatum)와 포항기청산식물원, 충북농업기술원, 농촌진흥청 국립원예특작 과학원 인삼특작부에서 분양받은 각시둥굴레 (P. humile), 용 둥굴레 (P. involucratum), 층층둥굴레 (P. stenophyllum), 층층 갈고리둥굴레 (P. sibiricum), 진황정 (P. falcatum) 등 총 7종 12점의 식물자원을 공시재료로 사용하였으며, 공시재료의 분 류학적 구분은 농촌진흥청 농업유전자원센터에 의뢰하였다. 또한 둥굴레와 형태적으로 유사한 애기나리속 윤판나물 (Disporum sessile) 1점과 서울 경동시장에서 구입한 가공되지 않은 둥굴레속 한약재 옥죽 1점을 둥굴레 생체 시료들과 비교 하고자 사용하였다 (Table 1).
2. 외부 형태적 분석
둥굴레속 식물체의 외부 형태적 분석을 위해 수집지역별로 3개체씩 선별하여 종간의 형태적 차이를 비교하였다. 이를 위 하여 엽장 및 엽폭, 장폭비를 측정하였으며, 엽연 (葉緣), 엽선 (葉先), 엽맥 (葉脈), 근경 (根莖) 등 각 식물종의 형태적 특성 을 비교하였다.
3. DNA 추출 및 PCR 분석
둥굴레속 식물체의 엽록체 DNA를 추출하기 위해 식물체의 잎으로부터 HiYieldTM Genomic DNA Mini Kit (RBC, Inc., Taipei, Taiwan)를 이용하여 제조사의 매뉴얼에 따라 처리하 였다. 추출된 DNA는 Multiskan GO UV/Vis microplate spectrophotometer (Thermo Scientific, Waltham, MA, USA) 를 이용하여 농도 및 순도를 확인하였으며 각각의 시료를 20 ng/㎕로 정량하여 PCR 반응에 사용하였다. PCR 분석을 위해 엽록체 DNA 구간인 rpoC1, rpoB2, matK, psbA-trnH 의 서열 내에서 primer를 제작하여 각각 사용하였으며, PCR 증폭은 40 ng genomic DNA와 forward 및 reverse primer 10 pmol, 10 mM Tris-HCl (pH 8.0), 50 mM KCl, 2.0 mM MgCl2, 2 unit Taq-DNA polymerase와 함께 총 반응액을 40㎕로 하여 Thermo cycler (PTC-200 DNA Engine, Bio- Rad Co., Hercules, CA, USA)에서 각 primer 조건별로 수행 하였다 (Table 2). PCR 증폭은 결과의 신뢰도를 높이기 위하 여 각 시료 당 2회 씩 반복하였다.
4. 염기서열과 계통학적 유연관계 분석
증폭된 PCR 산물은 정제 후 ㈜제노텍 (Genotech Inc., Daejeon, Korea)에 의뢰하여 ABI Prism 3730xl DNA sequencer (Applied Biosystems, Foster City, CA)로 염기서 열을 분석하였다. 또한 본 연구에 사용된 둥굴레의 종간 염기서열 차이 비교를 위해 NCBI (National Center for Biotechnology Information, Bethesda, MD, USA)에 등록된 동 일 종 또는 실험에 제외된 총 13종을 선정하여 적용하였다. 각 시료의 염기서열은 BioEdit version 7.2.5 프로그램 ( http://bioedit.software.informer.com )의 ClustalW multiple alignment 로 정렬한 후 MEGA6 (molecular evolutionary genetics analysis version 6.0) (Tamura et al., 2004)를 이용하여 Neighbor-Joining (NJ) 방법으로 (Saitou and Nei, 1987) 분 석하여 phylogenetic tree를 작성하였으며, 계통수에 대한 지지 도는 Bootstrap 값을 1000회 반복하여 평가하였다 (Felsenstein, 1985).
결 과
1. 외부 형태적 특성 비교
둥굴레속 식물의 외부 형태적 특성을 비교한 결과는 Fig. 1 및 Table 3와 같다. 엽장은 층층갈고리둥굴레가 다른 종들에 비해 길었으며 장폭비는 층층둥굴레와 함께 약 6 : 1로 선형의 엽형인 것을 확인하였다 (Fig. 1D, 1E). 한편 진황정을 포함한 타 둥굴레 속 식물체들의 엽형은 난형 (Fig. 1C) 또는 장타원 형인 것으로 확인되어 (Fig. 1A, 1B, 1F, 1G), 층층둥굴레 및 층 층갈고리둥굴레의 잎은 타 둥굴레 식물 종들과 뚜렷하게 구별 되었다. 엽연은 둥굴레속 전체가 거치가 없이 밋밋하였으며, 엽맥은 평행맥이었다 (Table 3). 둥굴레 속 식물체의 엽선은 전반적으로 뾰족한 형태이며, 층층갈고리둥굴레의 경우 잎 끝 이 말리는 것이 층층둥굴레를 포함한 둥굴레속 잎들의 뾰쪽한 말단과 구별되었다. 엽서는 층층둥굴레와 층층갈고리둥굴레의 경우, 3개에서 8개의 잎이 윤생하며 이를 제외한 나머지 종은 호생하는 것으로 확인되었다. 뿌리줄기의 직경은 층층갈고리둥 굴레가 가장 굵었으며, 각시둥굴레가 가장 가늘어 타종과 뚜렷 한 차이를 보였다. 그러나 뿌리줄기가 한약재로서 시중에 유통 될 때에는 다양한 형태로 가공되고 있으므로 외부 형태적 특 성 비교에 따른 명확한 판별에는 한계가 있을 것으로 사료된다.

Morphological characteristics of Polygonatum species.A; P. odoratum var. pluriflorum, B; P. humile, C; P. odoratum var. pluriflorum f. variegatum, D; P. stenophyllum, E; P. sibiricum, F; P. falcatum, G; P. involucratum, H; Disporum sessile.
2. PCR 증폭 및 종간 염기서열 분석
7종의 둥굴레속 식물의 엽록체 DNA로부터 rpoC1, rpoB2, matK, psbA-trnH 구간을 PCR 증폭한 결과, rpoC1에서는 약 550 ~ 600 bp, rpoB2에서는 약 550 ~ 600 bp, matK에서는 약 900 ~ 950 bp, psbA-trnH에서는 약 650 ~ 700 bp의 범위에서 PCR 산물이 확인되었다 (Fig. 2). 각 PCR 산물은 정제하여 염기서열분석 및 정렬 후, 각각 432 bp, 463 bp, 811 bp, 563 bp의 염기서열에 대한 SNP 분석을 수행하였다. 또한 phylogenetic tree 내에서 발생한 분기점에 대한 Bootstrap value를 참고하여 종간 차이를 분석하였다.
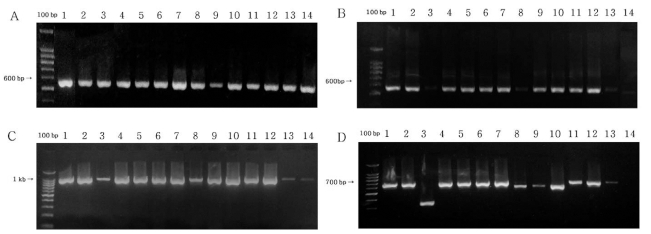
PCR amplification products by using chloroplast DNAs.A; rpoC1, B; rpoB2, C; matK, D; psbAtrnH, 1; Polygonatum odoratum var. pluriflorum (Pohang), 2; P. odoratum var. pluriflorum (Ilsan), 3; Disporum sessile, 4; P. odoratum var. pluriflorum f. variegatum (Pohang), 5; P. odoratum var. pluriflorum f. variegatum (Ilsan), 6; P. odoratum var. pluriflorum f. variegatum (Cheongju), 7; P. stenophyllum (Eumseong), 8; P. stenophyllum (Pohang), 9; P. sibiricum, 10; P. falcatum (Pohang), 11; P. falcatum (Cheongju), 12; P. involucratum, 13; P. humile, 14; Okjuk (dried).
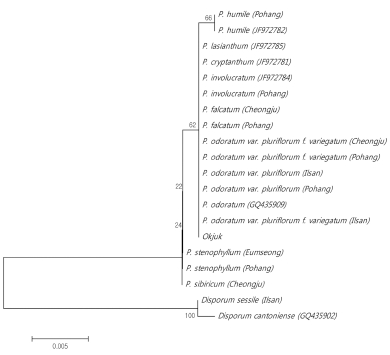
엽록체 DNA의 coding region인 rpoC1 구간에서 확인된 SNP는 총 25개로 각시둥굴레에서 1개, 둥굴레속과 동일한 백 합과에 속하는 애기나리속인 윤판나물에서 23개의 SNP가 발견되었으며, 윤판나물과 대만윤판나물 (D. cantoniense, Genbank accession no. GQ435902) 사이에는 1개의 SNP 차 이가 확인되었다 (data not shown). Bootstrap을 분석한 결과, 윤판나물과 둥굴레속은 100%의 bootstrap value로 자매군 (Sister group)으로서 명확하게 분지되었다 (Fig. 3). 둥굴레속 내에서는 층층둥굴레와 층층갈고리둥굴레가 하나의 집단으로 구분되었으며, 이때의 bootstrap 값은 62%로 나타났다. 나머지 둥굴레속에서는 각시둥굴레가 66%의 bootstrap 지지도로 타종 과 구분되었다. 한편, 한약재 옥죽의 경우 Neighbor-Joining tree 상에서 둥굴레 그룹 내에 속해있어 대한민국약전한약 (생 약)규격집 10개정 (2012)에 기재된 옥죽에 대한 규정과 일치 되는 결과를 보여주었다.
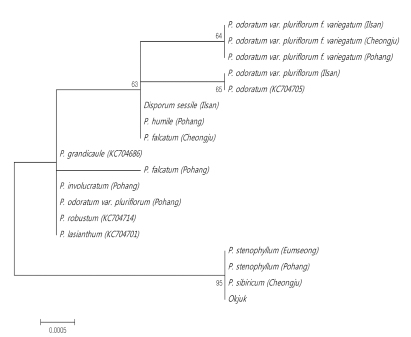
rpoB2 구간에서 뚜렷하게 확인된 SNP는 총 1개였다. 이 부 위에 의하여 층층둥굴레와 층층갈고리둥굴레가 하나의 그룹으 로 묶여 다른 둥굴레속 식물 종들과 구별되었으며, 이때의 bootstrap 지지도는 65%로 나타났다 (Fig. 4). 한편, 한약재 옥죽은 층층둥굴레 및 층층갈고리둥굴레와 같은 집단 내에 있 어, 대한민국약전 기준에 따라 본 실험에 이용된 한약재가 황 정이거나 혼용되었을 가능성이 있는 것으로 생각된다.
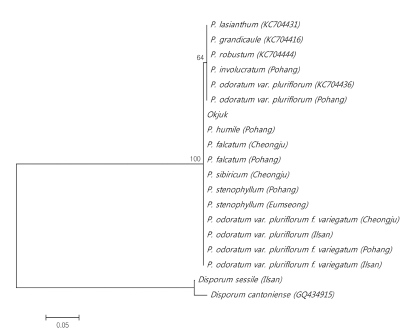
matK 구간 내 염기서열에서는 7개의 SNP가 확인되었으며 (data not shown), 층층둥굴레와 층층갈고리둥굴레의 서열에는 3개의 SNP가 존재하여 동속타종과 구별되었다. 포항기청산식 물원 둥굴레의 matK 구간 내 염기서열은 용둥굴레 (P. involucratum)와 동일하였고, NCBI에 등록된 선둥굴레 (P. grandicaule), 왕둥굴레 (P. robustum), 죽대 (P. lasianthum)의 서열과도 동일하였다. 그러나 동일종인 일산 동국대학교실험 농장 보유 개체의 서열은 NCBI에 등록된 둥굴레 (Genbank accession no. KC704705)와 일치하였으며, 포항과 청주에서 분양받은 두 진황정 간에도 서로 다른 위치에서 SNP가 1개씩 확인되어 종내 SNP 차이를 확인하였다. Bootstrap 분석 결과, 층층둥굴레와 층층갈고리둥굴레가 95%의 높은 bootstrap 지지 도로 동속타종과 구분되어 matK 구간이 층층둥굴레집단을 판 별하는 기준이 될 가능성을 보여주었다. 또한 한약재 옥죽이 Neighbor-Joining tree 상에서 층층둥굴레 및 층층갈고리둥굴레 집단에 속하는 것이 확인됨에 따라 황정과의 혼용가능성을 보 여주고 있다 (Fig. 5). 반면 동일한 분기군으로부터 분지된 다 른 둥굴레속 자매군의 경우, 무늬둥굴레집단이 64%의 bootstrap 수치로 분지된 것을 제외하면 나머지 종들은 분지 경향이 동종 내에서도 변동이 심하였으며, 애기나리 속인 윤 판나물 또한 이들과 구분되지 않았다.
엽록체 DNA의 non-coding region인 psbA-trnH spacer에서 는 둥굴레속의 종간 차이를 나타내는 2개의 SNP가 확인되었 고, 층층둥굴레와 층층갈고리둥굴레 서열에서는 74개의 연속 적 gap과 SNP 1개가 발견되었으며, 윤판나물의 서열에서는 각각 311개와 5개의 연속적 gap이 발견되어 둥굴레속 식물종 과 확연히 구별되었다 (data not shown). 분지양상은 둥굴레속 이 애기나리속인 윤판나물과 자매군으로서 100%의 bootstrap value로 분지되었고, 둥굴레속 내에서는 포항기청산식물원 분 양 둥굴레가 용둥굴레, 왕둥굴레, 선둥굴레, 죽대와 동일 집단 으로 분지되어 일산 동국대실험농장 보유 둥굴레 개체와 다른 집단으로 분류되었다 (Fig. 6). 한편, rpoC1, rpoB2, matK 세 구간에서 비교적 뚜렷하게 구분되었던 층층둥굴레와 층층갈고 리둥굴레가 다른 종으로부터 구분되지 않아 이들을 위한 판별 마커로 이용하기는 어려울 것으로 사료된다.
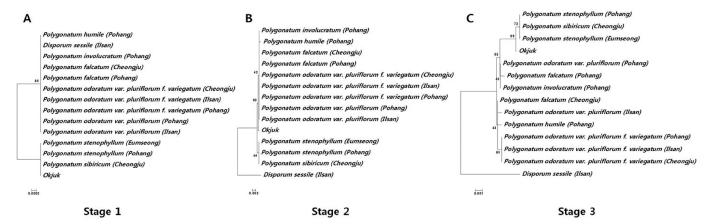
본 연구에서 상대적으로 종간 차이가 적었던 rpoB2 구간을 rpoC1과 조합한 결과, rpoB2에서 구별되지 않았던 각시둥굴 레의 종간 구분이 확인되었으며 (Fig. 7A, 7B), 이에 matK 구 간을 추가 조합한 결과를 바탕으로 작성된 phylogenetic tree 에서는 matK 단독 이용 시 구분되지 않았던 각시둥굴레와 충 북농업기술원 분양 진황정 사이의 종간 차이가 확인되었다 (Fig. 7C).
고 찰
둥굴레속 식물은 종에 따라 한약재 옥죽 또는 황정으로 구 분되나, 형태적으로 종을 구분하기 어려우며, 약재 유통 시 절 단 및 건조 등 가공과정을 거치므로 식물원종의 기원 판별이 어렵다. DNA barcoding 분석법은 표준화된 마커를 사용하여 DNA수준에서 생물 종간 또는 종내 SNP 차이를 비교분석하 는 방법으로 형태적 분류동정법의 한계를 보완할 수 있을 뿐 만 아니라 식물 분류 및 약재 원산지 판별에 용이하다.
본 연구의 공시재료인 둥굴레속 7종 식물자원은 농촌진흥청 농업유전자원센터 성정숙 박사의 도움으로 종간 형태적 비교 를 수행하였다. 한약재 황정으로 사용되는 진황정 및 층층갈 고리둥굴레의 경우 타종들과 육안으로 일부 구별이 가능하였 다 (Fig. 1). 그러나 다양한 형태로 가공유통되는 뿌리줄기의 경우 판별에 어려움이 있어 보다 명확한 구분을 위해 cpDNA 구간의 염기서열분석을 수행하였다. 실험에 이용된 4개의 cpDNA 구간 중 rpoC1, rpoB2, matK 구간에서 공통적으로 층층둥굴레 및 층층갈고리둥굴레와 구별되는 SNP가 확인되었 으며, Neighbor-Joining tree 에서도 이 그룹이 독립적으로 분 지된 것으로 보아 계통적 분류가 선행된 형태학적 연구결과와 일치하는 것으로 확인되었다 (Table 3). 따라서 rpoC1, rpoB2, matK 구간이 둥굴레속으로부터 층층둥굴레 및 층층갈 고리둥굴레를 판별하기 위한 바코드 유전자로 사용 가능할 것 이라 사료된다. 또한 이들 구간에서 층층둥굴레와 층층갈고리 둥굴레가 한 집단에 속함에 따라 층층둥굴레의 근경이 한약재 황정에 포함되는 것이 타당하다는 연구 결과를 뒷받침해준다 (An et al., 2006). 한편, 한약재 옥죽의 경우 rpoC1에서는 둥 굴레와 근연종으로 분류되고, rpoB2, matK에서는 층층둥굴레 및 층층갈고리둥굴레 그룹으로 분류되는 상반된 결과가 나와 옥죽의 유통과정에서 한약재 황정과 혼합되었을 가능성을 보 여준다.
한편, matK 구간은 동일한 종간에도 서열에 차이가 발생하 거나, 둥굴레와 속(屬)이 전혀 다른 윤판나물과 동일한 그룹으 로 분류되는 결과가 나와 둥굴레계열에 대한 판별마커로 이용 되기는 어려울 것으로 판단된다. 반면, 층층둥굴레 및 층층갈 고리둥굴레 그룹의 경우 95%의 높은 bootstrap 지지도로 동속 타종과 명확히 구분되어, 황정 판별에 있어서는 matK가 유용 할 것으로 사료된다 (Fig. 5).
psbA-trnH 결과에서 층층둥굴레 및 층층갈고리둥굴레의 경 우, 서열에서 수십 개의 indel이 발견되었으며, 타 둥굴레 종 들과 구분되는 SNP가 적어 phylogenetic tree 상에서도 분지 양상이 명확하지 않았다 (Fig. 6). Ma 등 (2014)는 둥굴레와 동과 식물인 산자고 (Tulipa edulis) 판별 연구에서 psbA-trnH 가 구간 내 insertion 및 deletion (indel)의 잦은 발생으로 판 별마커로서 적합하지 않다고 하였으며, 본 연구의 결과가 이 와 유사한 경향을 보인 바, 둥굴레속 판별에 psbA-trnH를 판 별 마커로 이용하기에는 부적합한 것으로 사료된다.
한편, psbA-trnH를 제외한 나머지 세 개 구간을 조합한 결 과에서 단일 primer 분석 시 차이를 보이지 않던 각시둥굴레 가 두 구간 이상의 상호조합 시 타종과 구분되었으며, 특히 rpoB2, rpoC1, matK 세 구간을 조합한 경우에는 각시둥굴레 와 충북농업기술원 분양 진황정의 종간 차이가 확인되었다 (Fig. 7).
본 연구결과에서 엽록체 DNA를 대상으로 한 barcoding 분 석은 둥굴레속 식물체 판별에 유용하며, 특히 종판별에서의 primer 단독 이용의 한계점이 여러 primer의 조합으로 상호보 완 됨으로써 비교적 정확한 결과가 도출되는 것을 확인하였다. 또한 본 연구 결과를 둥굴레속 기원 한약재 판별마커 개발뿐 만 아니라 향후 기타 약용식물자원의 표준화와 이들을 원료로 하는 건강기능식품 및 한약재의 품질개선을 위한 기초자료로 이용할 수 있을 것으로 사료된다.
감사의 글
본 연구는 2014년도 농촌진흥청 공동연구사업(과제번호: PJ008567072014) 연구비 지원에 의해 수행된 결과로 이에 감 사드립니다.
REFERENCES
- Ahn, DK, A herbal study on Polygonatum species, Korean Journal of Pharmacognosy, (1985), 16, p105-113.
-
An, HJ, Chung, KM, Analysis of index components in medicinal crops of Andong area cultivated at different altitude, Korean Journal of Medicinal Crop Science, (2014), 22, p91-97.
[https://doi.org/10.7783/kjmcs.2014.22.2.91]

- An, SM, Ryuk, JA, Kim, YH, Chae, BC, Kim, HJ, Kim, KH, Kang, KK, Ko, BS, Lee, MY, Genetic analysis of Polygonati rhizoma and Polygonati odorati rhizoma using random amplified microsatellite polymorphism, Korean Journal of Medicinal Crop Science, (2006), 14, p125-129.
- CBOL Plant Working Group, A DNA barcode for land plants, (2009), Proceedings of the National Academy of Sciences of the United States of America, 106, p12794-12797.
-
Felsenstein, J, Confidence Limits on Phylogenies An approach using the bootstrap, Society for the study of Evolution, (1985), 39, p783-791.
[https://doi.org/10.2307/2408678]

- Ha, WY, Shaw, PC, Liu, J, Forrest, CFY, Wang, J, Authentication ofPanax ginsengandPanax quinquefoliususing amplified fragment length polymorphism(AFLP) and directed amplification of minisatellite region DNA(DAMD), Journal of Agricultural and Food Chemistry, (2002), 27, p1871-1875.
-
Hebert, PDN, Cywinska, A, Ball, SL, deWaard, Jr, Biological identifications through DNA barcodes, The Royal Society, (2003), 270, p313-321.
[https://doi.org/10.1098/rspb.2002.2218]

- Jang, CG, Kim, YS, Taxonomic relationships of the KoreanPolygonatum(Liliaceae) using the RAPDs analysis, Korean Journal of Plant Taxonomy, (1998), 28, p371-384.
-
Jang, CG, A taxonomic review of KoreanPolygonatum(Ruscaceae), Korean Journal of Plant Taxonomy, (2002), 32, p417-447.
[https://doi.org/10.11110/kjpt.2002.32.4.417]

- Jang, KH, Kang, JH, Jeon, BS, Song, GW, Lee, SD, Major chemical components in rhizome of nativePolygonatumspecies in Korea, Korean Journal of Medicinal Crop Science, (2002), 10, p185-193.
- Kim, CM, Shin, MK, Ahn, DK, Lee, KS, Encyclopedia of chinese herbal medicine, (1998), 10, Seoul, Korea, Jungdammedia, p6550-6556.
- Lee, MY, Kim, KH, Kim, YH, Oh, SE, Kang, KK, Ko, BS, A taxonomic examination ofPolygonatumrhizoma andPolygonati odoratirhizoma based on RAPD analysis, Korean Journal of Pharmacognosy, (2004), 35, p265-270.
-
Li, M, Cao, H, But, PPH, Shaw, PC, Identification of herbal medicinal materials using DNA barcodes, Journal of Systematics and Evolution, (2011), 49, p271-283.
[https://doi.org/10.1111/j.1759-6831.2011.00132.x]

-
Ma, HL, Zhu, ZB, Zhang, XM, Miao, YY, Guo, QS, Species identification of the medicinal plantTulipa edulis(Liliaceae) by DNA barcode marker, Biochemical Systematics and Ecology, (2014), 55, p362-368.
[https://doi.org/10.1016/j.bse.2014.03.038]

-
Moon, BC, Kim, WJ, Ji, Y, Lee, YM, Kim, HK, Genetic diversity ofCurcumagenus collected germplasm using analysis of AFLP, Korean Journal of Medicinal Crop Science, (2013), 21, p455-460.
[https://doi.org/10.7783/kjmcs.2013.21.6.455]

- Saitou, N, Nei, M, The neighbor-joining method A new method for reconstructing phylogenetic trees, Molecular Biology and Evolution, (1987), 4, p406-425.
- Seo, YS, Park, WH, Cha, YY, Effect ofPolygonatum odoratumon lowering lipid and antioxidation, Journal of Oriental Rehabilitation Medicine, (2011), 21, p49-62.
-
Tamura, K, Nei, M, Kumar, S, Prospects for inferring very large phylogenies by using the neighbor-joining method, Proceedings of National Academy of Sciences of the United States of America, (2004), 101, p11030-11035.
[https://doi.org/10.1073/pnas.0404206101]

-
Williams, JGK, Kubelik, AR, Livak, KJ, Rafalski, JA, Tingey, SV, DNA polymorphism amplified by arbitrary primers are useful as genetic markers, Nucleic Acids Research, (1990), 18, p6531-6535.
[https://doi.org/10.1093/nar/18.22.6531]

- Yun, JS, Kim, IH, Park, JS, Lee, CH, Hong, EY, Yun, T, Jong, SK, Characterization of 18S rDNA inPolygonatumspp. collections, Korean Journal of Medicinal Crop Science, (2006), 14, p178-182.