nrDNA ITS 및 엽록체 DNA 염기서열 분석에 의한 유통 한약재 오가피 판별
© The Korean Society of Medicinal Crop Science All rights reserved
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License ( http://creativecommons.org/licenses/by-nc/3.0 ) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Plants belonging to 5 species of the genus Eleutherococcus are currently distributed in the Korean peninsula. The traditional medicine ‘Ogapi’, derived from Eleutherococcus sessiliflorus and other related species, and ‘Gasiogapi’, derived from Eleutherococcus senticosus, are frequently mixed up and marketed. Therefore, accurated identification of their origins in urgently required.
Candidate genes from nuclear ribosomal DNA (nrDNA) and chloroplast DNA (cpDNA) of Eleutherococcus plants were analyzed. Whereas the nrDNA-internal transcribed spacer (ITS) regions were useful in elucidating the phylogenetic relationships among the plants, the cpDNA regions were not as effective. Therefore, a combined analysis with nrDNA-ITS was performed. Various combinations of nrDNA and matK were effective for discriminating among the plants. However, the matK and rpoC1 combination was ineffective for discriminating among some species. Based on these results, it was found that OG1, OG4, OG5, OG7, GS1, GS2, and GS3 were derived from E. sessiliflorus. In particular, it was confirmed that GS1, GS2, and GS3 were not derived from E. senticosus. However, more samples need to be analyzed because identification of the origins of OG2, OG3, OG6 and GS4 was not possible.
The ITS2, ITS5a, and matK combination was the most effective in identifying the phylogenetic relationship among Eleutherococcus plants and traditional medicines based on Eleutherococcus.
Keywords:
Eleutherococcus, Chloroplast DNA, DNA Barcoding, Internal Transcribed Spacers, Nuclear Ribosomal DNA서 언
두릅나무과 (Araliaceae) 다년생 낙엽관목인 오갈피속 (Eleutherococcus) 식물은 한국을 비롯한 중국 동북지역, 러시 아 우수리강 유역, 일본 북해도 등 동북아시아에 약 35종이 분포하며, 국내에는 오갈피나무 (Eleutherococcus sessiliflorus), 가시오갈피 (E. senticosus), 털오갈피 (E. divaricatus), 지리산 오갈피 (E. divaricatus var. chiisanensis), 섬오갈피나무 (E. gracilistylus) 총 5종이 자생하는 것으로 알려져 있다.
오갈피속 식물의 뿌리 및 줄기는 강장작용, 피로회복 및 인 체 저항력 증진 등의 약리효과가 있어 예로부터 민간약으로 이용되어 왔다 (Kim, 1997). 이들의 약리효과는 면역조절 (Kim et al., 2004), 항피로, 항우울 및 항스트레스 (Deyama et al., 2001)뿐만 아니라 최근 항산화 (Heo et al., 2011), 혈 당강하 및 항당뇨 (Lim et al., 2010)에 이르기까지 다양하게 보고되어 있다.
국내 약재시장에서 오갈피나무는 ‘오가피 (五加皮)’로, 가시 오갈피는 ‘가시오가피’로 유통되고 있다. 대한민국약전 제 10 개정 (MFDS, 2015)에서 오가피 (五加皮)는 오갈피나무 또는 기타 동속식물의 뿌리껍질 및 줄기껍질로 정의된다. 또한 가 시오가피는 대한민국약전외한약(생약)규격집 (MFDS, 2013)에 서 ‘자오가 (刺五加)’로 기재되어 있으며, 가시오갈피의 뿌리 및 뿌리줄기로 정의된다. 이렇듯 대한약전에 오가피와 가시오 가피가 구분 기재되어 있음에도 혼용 유통되는 사례가 많아 (Kim et al., 1997) 오갈피속 식물종을 포함한 약재 간에 명 확한 기원정립이 요구된다.
현재까지 오가피 기원정립을 위해 형태적 판별 연구 (Kim et al., 1997, 2008), 이화학적 성분 분석 (Kim et al., 2009), 분자유전학적 계통변이 분석 (Huh et al., 2005; Lim et al., 2000; Kim et al., 1998; Hong et al., 2000) 등 많은 연구 가 진행되어 왔다. 오갈피속 식물은 형태적 특성 분석을 통해 감별 가능하나 (Kim et al., 2008), 1년생과 2년생 이상 가지 의 가시밀생도 및 색상차이 등 동일 개체 내 변이가 큰 것으 로 보고되었으며 (Kim et al., 1997), 재배지역 및 시기, 부위 등에 따라 성분차이가 있어 (Kim et al., 1996) 형태 및 이화 학적 분석은 오갈피 종판별에 한계가 있다.
DNA 마커를 이용한 분자유전학적 판별 기법은 재배환경조 건에 영향을 받지 않아 다양한 약용식물 기원정립의 대안으로 제시되고 있다 (Li et al., 2011; Ma et al., 2014; Kim et al., 2014). 오갈피속 식물을 대상으로 한 분자유전학적 판별에 는 RAPD (Huh et al., 2005; Lim et al., 2000; Kim et al., 1998), ISSR 표지자 분석 (Hong et al., 2000) 등의 연 구가 수행되었으나 이들보다 재현성이 높고 특이적 마커 선발 이 가능한 분석법의 확립이 필요하다. Consortium for the Barcode of Life (CBOL)가 제안한 DNA barcoding 기술은 nrDNA (nuclear ribosomal DNA) 및 cpDNA (chloroplast DNA)의 후보유전자 구간으로부터 탐색된 SNP (Single Nucleotide Polymorphism) 차이를 통해 식물 종을 판별하는 방법으로 신속성, 정확성, 보편성 측면에서 식물 종간 변이 분 석에 유용하다 (CBOL Plant Working Group, 2009). 이와 관련하여 Lim 등 (2012)은 감초종 및 약재 판별연구에 DNA barcoding을 적용하였으며, Kim 등 (2014)은 이를 둥굴레속 식물 종간 유연관계에 적용한 바 있다. 오갈피속 식물을 대상 으로 ITS (internal transcribed spacer) 염기서열을 이용한 유 연관계 분석 (Han et al., 2006) 등이 수행된 바 있으나 이외 의 DNA barcoding 기술을 이용한 기원정립 연구는 미흡한 실정이다. 따라서 본 연구는 nrDNA 및 cpDNA 구간 염기서 열 분석을 통하여 오갈피속 식물 종간 유연관계 분석을 수행 하였으며, 이를 시중 유통 오가피 한약재 판별에 적용함으로써 한약재 유통 질서 확립과 품질 향상에 기여하고자 수행되었다.
재료 및 방법
1. 공시재료
본 연구를 위해 국립농업유전자원센터에 등록된 가시오갈피 (Eleutherococcus senticosus, IT239073, IT239084, IT239239, IT239040, IT239279) 5점과 오갈피나무 (Eleutherococcus sessiliflorus), 지리산오갈피 (Eleutherococcus divaricatus var. chiisanensis), 섬오갈피나무 (Eleutherococcus gracilistylus) 각 각 1점씩 총 4종의 자생종을 비롯하여, 중국 원산 재배종으로 알려진 오가나무 (Eleutherococcus sieboldianus) 1점을 강원도 농업기술원 인삼약초연구소로부터 분양받아 공시재료로 이용 하였다. 또한, NCBI (National Center for Biotechnology Information) Genbank에 등록된 12점의 염기서열 정보를 본 연구에 사용한 오갈피속 식물 5종과 비교하였다. 한편, 국내 유 통 오가피 한약재 판별을 위해 서울약령시장, 금산약재시장, 대 구약령시장으로부터 다양한 명칭으로 유통 중인 국내산 오가 피 10점과 중국산 1점, 총 11점을 수집하여 약재 판별에 이용 하였다 (Table 1).
2. 외부 형태적 분석
오갈피속 식물체 5종에 대한 형태적 분석을 위해 원색대한 식물도감 (Lee, 2006)을 참고하여 잎과 줄기의 형태를 비교하 였다. 잎의 경우, 엽형, 엽색을 비롯하여 엽장 및 엽폭, 장폭비 를 측정하였으며, 줄기는 수피의 색상 및 가시의 유무 및 형 태를 바탕으로 특성을 비교하였다.
3. DNA 추출 및 PCR 분석
오갈피속 식물의 nrDNA 및 cpDNA를 추출하기 위해 식물 체의 잎으로부터 HiYield™ Genomic DNA Mini Kit (RBC Bioscience Co., Taipei, Taiwan)를 이용하여 제조사의 매뉴얼 에 따라 처리하였다. 추출된 DNA는 Multiskan GO UV/Vis microplate spectrophotometer (Thermo Fisher Scientific, Waltham, MA, USA)를 이용하여 농도 및 순도를 확인하였으 며, 각각의 시료를 20 ng/μℓ로 정량하여 PCR 반응에 이용하 였다. PCR 분석을 위해 nrDNA 구간인 internal transcribed spacers (ITS2 및 ITS5a), 그리고 cpDNA 구간인 Maturase K (matK), RuBisCO large subunit (rbcL), RNA polymerase subunit (rpoB2 및 rpoC1)의 서열 내에서 primer를 제작하여 사용하였으며, PCR 증폭은 40 ng genomic DNA와 forward 및 reverse primer 10 pmol, 20 mM Tris-HCl (pH 8.0), 100 mM KCl, 2.5mM MgCl2, 1.25 unit Taq-DNA polymerase 와 함께 총 반응액을 50μℓ로 하여 PTC-200 DNA Engine thermo cycler (Bio-Rad Co., Waltham, MA, USA)에서 각 primer 조건별로 수행하였다 (Table 2). PCR 증폭은 결과의 신 뢰도를 높이기 위하여 각 식물시료 당 2회씩 반복하였다.
. 염기서열 및 분자계통학적 유연관계 분석
증폭된 PCR 산물은 정제 후 ㈜제노텍 (Genotech Inc., Daejeon, Korea)에 의뢰하여 ABI Prism 3730xl DNA sequencer (Applied Biosystems, Foster City, CA, USA)로 염 기서열을 분석하였다. 각 시료의 염기서열은 BioEdit version 7.2.5 프로그램 ( http://bioedit.software.informer.com/ )의 ClustalW multiple alignment로 정렬한 후 MEGA6 (molecular evolutionary genetics analysis version 6.0) (Tamura et al., 2004)의 Kimura 2-parameter method로 유전적 거리를 계산하 여 neighbor-joining (Saitou and Nei, 1987) 방법으로 phylogenetic tree를 작성하였다. 계통수에 대한 지지도는 bootstrap 값을 1000회 반복하여 평가하였다 (Felsenstein, 1985).
결과 및 고찰
1. 외부 형태적 특성 비교
오갈피속 식물 잎의 형태적 특성은 모두 5출엽인 장상복엽 으로, 오갈피나무의 소엽은 잎의 장폭비가 약 1 : 2.3인 도란상 타원형, 가시오갈피와 지리산오갈피는 각각 1 : 2.7과 1 : 2.2 비 율의 타원상 도란형, 섬오갈피나무 및 오가나무는 각각 1 : 1.9와 1 : 2.9 비율의 도피침형이었다. 가지의 형태에 있어서 오 갈피나무, 지리산오갈피, 섬오갈피나무는 회갈색, 그리고 가시 오갈피와 오가나무는 회백색을 보였으며, 가지 경침 형태 또 한 다양하였다. 오갈피나무의 가지에는 경침 흔적만이 확인된 반면, 지리산오갈피와 섬오갈피나무의 경우 경침이 짧았고, 가 시오갈피와 오가나무에서는 다소 긴 경침이 관찰되었다. 관찰 된 가시오갈피의 형태는 회갈색으로 기재된 원색대한식물도감 (Lee, 2006)의 내용과 상이하였으며, 엽색의 경우 광택이 있는 섬오갈피나무 및 진녹색의 오가나무와는 쉽게 구분되었으나 오갈피나무 및 지리산오갈피와는 구분이 어려웠다 (Fig. 1). 한 편 Kim 등 (1997)은 가시오갈피가 동일 개체 내에서도 변이 가 크다고 보고하였는데, 섬오갈피나무에서도 생육단계에 따 라 어린 가지와 과년지 (過年枝)에서 경침의 발달에 차이를 보 이므로 형태적 특성에 의한 오갈피속 식물 종판별은 어려울 것으로 생각된다.
2. DNA barcoding에 의한 오갈피속 5종의 계통 분류
오갈피속 식물 5종에 대하여 nrDNA 구간인 ITS2, ITS5a 와 cpDNA 구간인 matK, rbcL, rpoB2, rpoC1을 증폭한 결 과, ITS2는 450 - 500 bp, ITS5a에서는 700 - 800 bp 범위에서 증폭 밴드가 확인되었고, matK와 rbcL에서는 각각 900 bp - 1 kb와 700 - 800 bp, rpoB2와 rpoC1은 500 - 600 bp 범위에서 확인되었다 (Fig. 2). DNA barcoding은 이종 식물 간 동일 유전자 구간의 SNP 차이에 의해 식물 종을 구분하는 방법으 로, 분석대상이 되는 이종 식물의 동일 유전자 구간의 크기는 통상적으로 유사하다 (Hollingsworth et al., 2011). 오갈피속 식물을 대상으로 한 각 유전자 구간의 PCR 결과는 감초 (Lim et al., 2012), 방풍 (Lim et al., 2013), 둥굴레 (Kim et al., 2014) 등 타과 식물 대상 연구로부터 얻어진 밴드의 크기와 유사한 것으로 확인되어 Hollingsworth 등 (2011)의 보고와 일치하였다. 증폭된 PCR 산물은 염기서열분석을 진행 하였으며 ITS2와 ITS5a는 각각 423 bp, 615 bp, matK, rbcL, rpoB2, rpoC1에서는 각각 752 bp, 675 bp, 415 bp, 487 bp 구간을 대상으로 SNP 및 phylogeny 분석을 통해 종 간 유연관계를 확인하였다.
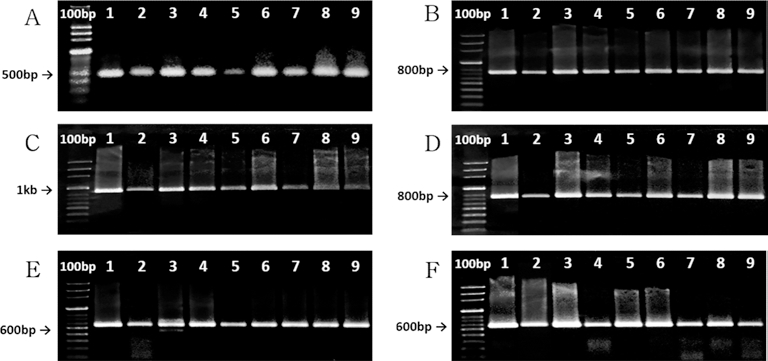
PCR products of Eleutherococcus spp.A; nrDNA ITS2, B; nrDNA ITS5a, C; cpDNA matK, D; cpDNA rbcL, E; cpDNA rpoB2, F; cpDNA rpoC1, 1; E. sessiliflorus, 2; E. divaricatus var. chiisanensis, 3; E. gracilistylus, 4; E. sieboldianus, 5; E. senticosus (Yangpyeong-Yongmunsa), 6; E. senticosus (Bukhaedo), 7; E. senticosus (Cheorwon-Daeseongsan), 8; E. senticosus (Baekjeoksan), 9; E. senticosus (China).
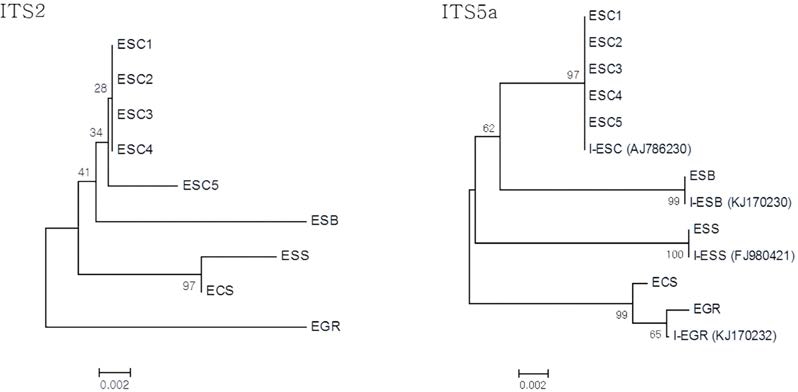
ITS2 구간에서 발견된 SNP는 총 20개로 (Table 3), 오갈피 나무 (E. sessiliflorus : ESS), 지리산오갈피 (E. divaricatus var. chiisanensis: ECS), 섬오갈피나무 (E. gracilistylus: EGR), 오가 나무 (E. sieboldianus: ESB), 가시오갈피 (E. senticosus: ESC) 총 5종의 오갈피속 식물 모두 phylogenetic tree 상에서 각각 다른 집단으로 분지되었다. 가시오갈피 집단의 경우, ESC5를 제외한 나머지 4개의 개체들이 한 집단으로 유집되었 으며, ESC5의 경우, 다른 가시오갈피 종들과 근접한 분지양상 이 나타나 종내 변이의 가능성을 보여주었다. 이는 가시오갈 피 군락 간 유전변이 발생도가 타종보다 높으며 (Hong et al., 2000), 군락이 위치한 지역 환경에 따른 유전변이에 기인 한 것으로 사료된다.
ITS5a 구간에서는 총 29개의 SNP가 확인되었으며 (Table 4), 이를 바탕으로 phylogenetic tree로 나타낸 결과, ITS2와 마 찬가지로 오갈피속 식물 5종이 각각의 집단으로 분지되었다. 한편 5개체의 가시오갈피가 97%의 높은 지지도로 하나의 집 단으로 유집되어, ESC5가 독립적으로 분지되었던 ITS2의 결 과와 차이를 보였다 (Fig. 3). 이러한 차이는 ITS2 구간에서 가시오갈피 종내 개체 간 차이를 보이는 SNP가 발견된 것에 기인하며, ESC5의 ITS2 구간 염기서열 결과는 가시오갈피 종 내 변이 연구에 이용될 수 있을 것으로 사료된다.
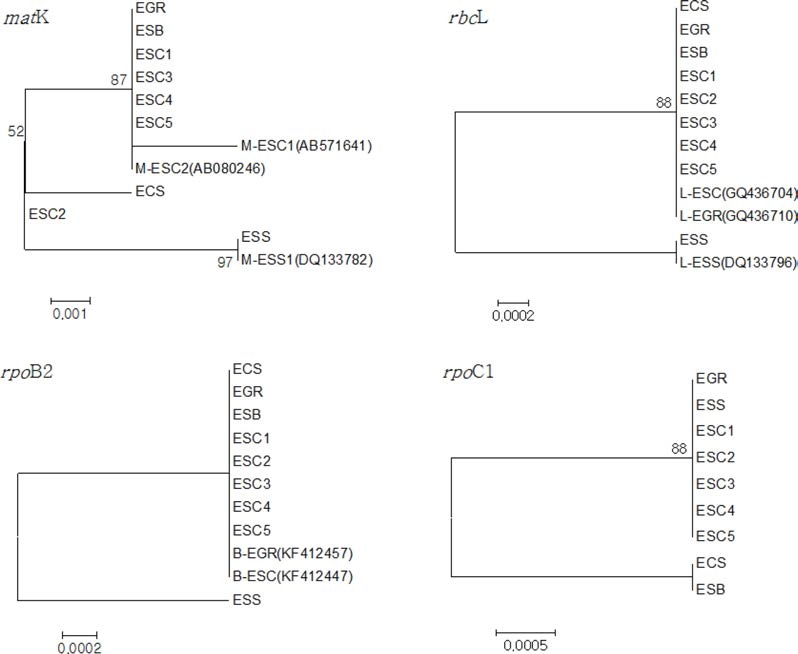
matK 구간에서 확인된 SNP는 총 8개로 (Table 5), 이에 대한 phylogenetic tree 분석 결과 오갈피나무와 지리산오갈피 가 타종들과 구분되었으며, 섬오갈피나무, 오가나무, 가시오갈 피는 하나의 집단으로 유집되어 서로 구분되지 않았다. 한편, ESC2의 경우 나머지 가시오갈피 개체들과 상대적으로 큰 유 전적 차이로 분지되었으며, NCBI에 등록된 가시오갈피 MESC1은 다른 가시오갈피 종들과 독립된 분지양상을 보여, 이 들 개체에서 종내 변이가 발생하였을 것으로 사료된다.
오갈피속 식물 종의 rbcL 구간 내 SNP는 2개, rpoB2 구 간의 경우 1개로 (Table 5), 이에 대한 phylogenetic tree 분 석 결과 오갈피나무만 구분되고 나머지 종은 한 집단으로 유 집되는 양상을 보였다. 한편, rpoC1 구간의 경우 총 2개의 SNP가 발견되었다 (Table 5). Phylogenetic tree 상에서 오갈 피나무와 가시오갈피 및 섬오갈피나무가 하나의 집단으로, 그 리고 지리산오갈피와 오가나무가 동일 집단으로 유집되어 전 반적으로 rpoC1 구간 내에서는 오갈피속 식물 종이 구분되지 않았다 (Fig. 4). 본 연구 결과와 관련하여, rbcL은 거의 모든 식물에 적용가능하나 진화속도가 느려 식별력이 떨어진다는 보고가 있으며 (CBOL Plant Working Group, 2009; Little, 2014), rpoB2 및 rpoC1의 경우, 적은 SNP로 Kim 등 (2014)의 연구 결과와 유사한 경향을 보인 바, 근연 종간 판별 력이 낮은 것으로 사료된다.
본 연구에서 nrDNA ITS2 및 ITS5a 단독 이용만으로 오갈 피속 5종에 대한 구분이 가능하였다. 그러나 cpDNA 구간은 단독 primer 만으로는 종간 구분에 한계를 보여 Kim 등 (2014)의 연구결과와 일치하였으며, cpDNA 구간의 판별능에 대한 한계를 보완하기 위해 nrDNA와 cpDNA 구간의 조합 분석을 수행하였다. cpDNA 구간 중 rbcL과 rpoB2는 오갈피 나무를 제외한 나머지 종에 대한 판별 유용성이 크지 않아 조 합 대상 구간에서 제외하였다. ITS2와 matK 구간을 조합한 결과, ITS2에 의한 분석결과와 달리 ESC2가 추가적으로 분지 되었다 (Fig. 5A). ITS5a와 matK 구간 조합 결과, matK 단 독 이용 시 구분되지 않았던 오가나무와 섬오갈피나무가 분지 되어 구분이 용이하였으며, ITS5a 단독 적용시 가시오갈피 집 단에 속해있던 ESC2가 matK의 영향으로 분지되었다 (Fig. 5B). 이에 ITS2를 추가 조합한 결과, ITS5a 및 matK 조합에 서 가시오갈피 집단에 유집되어 있던 ESC5가 다른 가시오갈 피로부터 구분되었다 (Fig. 5C). 한편 cpDNA matK와 rpoC1 조합 결과, rpoC1에서 구분되지 않았던 가시오갈피, 오가나무, 오갈피나무, 그리고 matK에서 구분되지 않았던 오가나무가 두 유전자 구간의 상호보완으로 용이하게 구분되었다. 한편 섬오 갈피나무의 경우 여전히 가시오갈피 집단과 유집되어, matK 및 rpoC1 두 cpDNA 구간 조합에 의한 구분에 한계를 보였 다 (Fig. 5D).
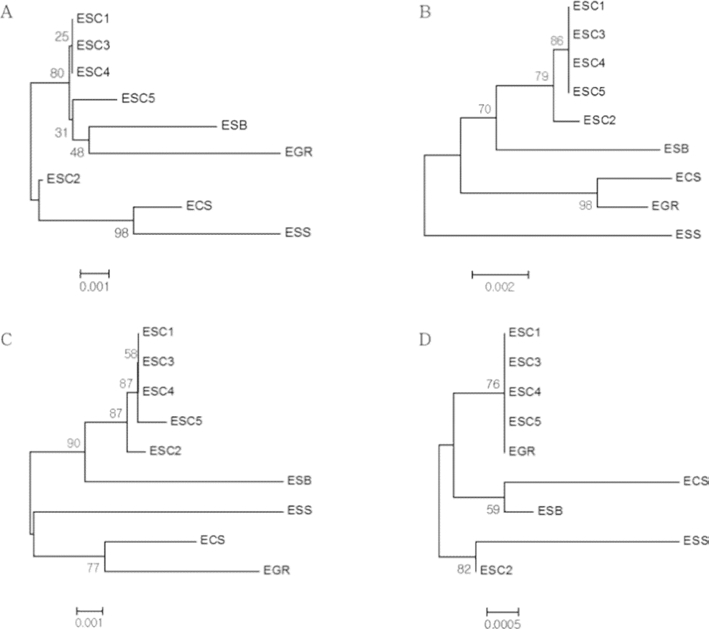
Combinations of neighbor-joining trees of nrDNA and cpDNA of Eleutherococcus spp.A; ITS2 + matK, B; ITS5a + matK, C; ITS2 +ITS5a +matK, D; matK+rpoC1, ESS; E. sessiliflorus, ECS; E. divaricatus var. chiisanensis, EGR; E. gracilistylus, ESB; E. sieboldianus, ESC; E. senticosus.
본 연구에서 수행된 4가지 조합 중 ITS2, ITS5a 및 matK 조합에서는 오갈피속 식물 5종이 구분된 것은 물론, 가시오갈 피 종내 ESC2와 ESC5가 독립적으로 분지됨으로써 본 연구에 서 수행된 조합 중 판별능이 가장 우수하였다. 따라서 ITS2 + ITS5a + matK 세 구간의 조합이 오갈피속 식물 종간 유연관계분석에 있어 최적의 조합인 것으로 사료된다.
시중 유통 오가피는 다양한 형태로 줄기가 종단 또는 횡단 으로 절단되거나 수피가 박피된 형태였고, ‘오가피’, ‘오가목’, ‘오갈피나무’, ‘진오가피’, ‘가시오가피’ 등의 명칭으로 유통되 며, 유통 명칭이 동일하더라도 가공된 형태가 달라 이들 약재 간 육안 구분이 어려웠다 (Fig. 6). 이와 관련하여 Kim 등 (2008)은 오가피가 뿌리 및 줄기껍질이 약재로 이용되어 다양 한 형태로 유통되므로 이들의 정확한 형태적 판별의 중요성을 보고하였으며, Kim 등 (1997)은 동일 개체 내에서 큰 형태적 변이의 문제를 언급하였다. 따라서 본 연구에서는 이러한 문 제점들을 보완할 수 있는 분자생물학적 분석법의 확립을 위해 시중 유통 오가피 약재를 대상으로 원 식물종과의 염기서열 차이를 비교하여 약재 판별을 수행하고자 하였다.
시중 유통 오가피 한약재 판별을 위해 nrDNA ITS 및 cpDNA 구간 분석을 바탕으로 오갈피속 식물의 유연관계를 분 석한 후, 오가피 약재 판별에 적용하였다. 대상 유전자의 단독 구간 분석에 있어서 nrDNA의 ITS2 및 ITS5a 구간이 오갈피 속 식물 판별에 유용하였으나, 가시오갈피, 지리산오갈피, 섬 오갈피나무의 유집형태가 다소 다른 결과를 보였고 (Fig. 3), cpDNA의 matK 구간에 있어서는 섬오갈피나무와 오가나무의 구분이 어려웠다 (Fig. 4). 이에 nrDNA의 ITS2와 ITS5a 및 cpDNA matK 구간의 조합분석을 수행한 결과 구분이 불분명 했던 종간종내 판별력이 향상되어 (Fig. 5C) 이를 한약재 판 별에 적용하였다.
오갈피속 식물체 구분에 최적 조합으로 판단되는 ITS2 + ITS5a + matK 조합을 이용하여 시중 유통 오가피 한약재 11점 의 판별에 적용한 결과, OG1, OG4, OG5, OG7, GS1, GS2, GS3가 오갈피나무와 동일한 집단으로 유집되어 오갈피 나무로부터 유래된 약재인 것으로 확인되었다 (Fig. 7). 특히 GS1, GS2, GS3의 경우 시중에서 가시오가피라는 명칭으로 유통된 한약재로서, 실제로는 가시오갈피 (E. senticosus)로부 터 유래된 약재가 아닌 것으로 사료된다. 이러한 결과는 Kim 등 (1997)이 국내 가시오가피 시중품 대부분은 지리산오갈피 및 오갈피나무의 수피인 것으로 보고한 것과 연관성이 있다. 한편 OG2, OG3, OG6, GS4는 본 연구에 이용된 오갈피속 5종과는 별개의 집단으로 분지되어 이들 한약재의 기원 종판 별이 가능하지 않았다. 따라서 다양한 종의 오갈피속 식물체 수집 및 추가연구가 필요할 것으로 사료된다.
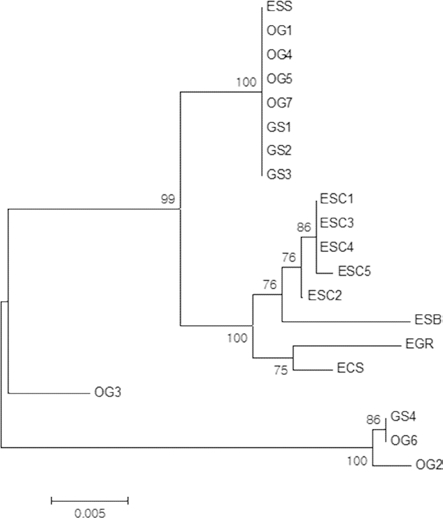
Neighbor-joining tree combining matK, ITS5a, and ITS2 sequences of plants and dried medicinal materials of Eleutherococcus spp.ESS; E. sessiliflorus, ECS; E. divaricatus var. chiisanensis, EGR; E. gracilistylus, ESB; E. sieboldianus, ESC; E. senticosus, OG1; Ogapi, OG2; Ogapi, OG3; Ogapi (China), OG4; Ogamok, OG5; Ogamok, OG6; Ogalpinamu, OG7; Jinogapi, GS1; Gasiogapi, GS2; Gasiogapi, GS3; Gasiogapi, GS4; Gasiogapi.
CBOL Plant Working Group (2009)은 비록 cpDNA의 matK, rbcL, rpoB2, rpoC1 등 다양한 유전자 구간이 식물의 종 구분에 유용하지만, 이들 구간의 단독 이용보다는 유전자 조합에 의한 분석에서 더 높은 신뢰도를 갖는다고 하였으며, Li 등 (2011)은 ITS 구간은 변이가 많아 높은 종 판별력을 가질 뿐만 아니라, 기술적으로 유전자 구간의 증폭이 용이하 여 식물 계통분석은 물론 약재 판별에 자주 이용된다고 하였 다. 현재 국내에서 DNA barcoding을 이용한 시중 유통 한약 재 판별 연구는 감초 (Lim et al., 2012)와 방풍 (Lim et al., 2013) 등 제한적인 식물 종을 대상으로 nrDNA ITS2 또 는 cpDNA의 matK 및 psbA-trnH 구간을 단독으로 이용한 사례에 머물러 있을 뿐 다양한 유전자 조합을 통한 유통 한약 재 판별연구는 미흡한 실정이다.
본 연구에서 시중 유통 오가피 한약재 판별을 위해 nrDNA 의 ITS2 및 ITS5a 각각을 이용한 결과가 ITS2 + ITS5a + matK 조합을 이용한 약재 판별 결과와 동일하였으나 (data not shown), nrDNA 및 cpDNA 조합 시 오갈피속 식물체에 대한 판별력이 향상되었기 때문에 이 조합을 바탕으로 한 오 가피 한약재 판별이 신뢰성이 높을 것으로 판단된다.
향후 추가적인 오갈피속 식물 종 수집 및 약재 판별 연구가 필요하나, nrDNA와 cpDNA 유전자 구간의 조합을 이용한 본 연구결과는 오가피 한약재뿐만 아니라 오• 혼용 가능성이 높은 약용식물 기원정립, 판별기술을 활용한 한약재 유통질서 확립, 원료표준화에 의한 한약재 품질향상에 기여할 수 있을 것으로 사료된다.
감사의 글
본 연구는 농촌진흥청 연구사업(과제번호: PJ008567072015) 의 지원에 의해 이루어진 결과로 이에 감사드립니다.
REFERENCES
- CBOL Plant Working Group, A DNA barcode for land plants, Proceedings of the National Academy of Science of the United States of America, (2009), 106, p12794-12797.
- Deyama, T, Nishibe, S, Nakazawa, Y, Constituents and pharmacological effects of Eucommia and siberian ginseng, Acta Pharmacologica Sinica, (2001), 22, p1057-1070.
-
Felsenstein, J, Confidence limits on phylogenies An approach using the bootstrap, Evolution, (1985), 39, p783-791.
[https://doi.org/10.2307/2408678]

- Han, HS, Kim, DY, Lee, KY, Park, WG, Cho, IK, Jung, J, Comparative analysis of Acanthopanax senticosus Harms from Korea China and Russia based on the ITS sequences of nuclear ribosomal DNA, Korean Journal of Plant Resources, (2006), 19, p54-58.
-
Heo, SJ, Ahn, HY, Kang, MJ, Lee, JH, Cha, JY, Cho, YS, Antioxidative activity and chemical characteristics of leaves Roots. stems and fruits extracts from Acanthopanax senticosus, Journal of Life Science, (2011), 21, p1052-1059.
[https://doi.org/10.5352/jls.2011.21.7.1052]

-
Hollingsworth, PM, Graham, SW, Little, DP, Choosing and using a plant DNA barcode, PLoS ONE, (2011), 6, -e19254.
[https://doi.org/10.1371/journal.pone.0019254]

- Hong, KN, Cho, KJ, Park, YH, Hur, SD, Hong, YP, Kang, BY, Genetic variation of some patches of Eleutherococcus senticosus(Rupr and Maxim.) Maxim. in Korea, Journal of Korean Forestry Society, (2000), 89, p645-654.
- Huh, MK, Choi, YH, Choi, BT, Kim, GC, Discrimination and genetic diversity of Acanthopanax senticosus using RAPD markers, Korean Journal of Oriental Physiology and Pathology, (2005), 19, p1046-1049.
- Kim, CH, Systematics of Eleutherococcus and related genera(Araliaceae), Ph. D. Thesis. Chonbuk National University, (1997), p1-263.
- Kim, HS, Han, HS, Lee, YJ, A study on a morphological identification of Acanthopanacis cortex, Korea Journal of Herbology, (2008), 23, p41-49.
-
Kim, JH, Seo, JW, Byeon, JH, Ahn, YS, Cha, SW, Cho, JH, Morphological characteristics and phylogenetic analysis of Polygonatum species based on chloroplast DNA sequences, Korean Journal of Medicinal Crop Science, (2014), 22, p489-496.
[https://doi.org/10.7783/kjmcs.2014.22.6.489]

- Kim, MS, Sung, CK, Kim, HC, Gal, SW, Lee, SW, Physicochemical composition of the Acanthopanax chilsanensis, Journal of Life Science, (2009), 19, p1815-1820.
- Kim, NS, Kwon, J, Koh, HY, Choi, DS, Oh, CH, Immunoregulatory action of Ogapi, Korean Journal of Oriental Physiology and Pathology, (2004), 18, p1337-1342.
- Kim, S, Kim, KY, Park, MS, Choi, SY, Yun, SJ, Intraspecific relationship of Eleutherococcus senticosus Max by RAPD markers, Korean Journal of Medicinal Crop Science, (1998), 6, p165-169.
- Kim, YJ, Park, MS, Park, HK, Kim, S, Sung, CK, Eleutheroside E content in Eleutherococcus spp, Korean Journal of Medicinal Crop Science, (1996), 4, p333-339.
- Kim, YJ, Park, HK, Park, MS, Kim, S, Choi, KG, Morphological and anatomical characteristics of five Eleutherococcus species, Korean Journal of Breeding Science, (1997), 29, p56-63.
- Lee, CB, Coloured Flora of Korea I, Hyangmunsa. Seoul. Korea, (2006), p811-813.
-
Li, M, Cao, H, But, PPH, Shaw, PC, Identification of herbal medicinal materials using DNA barcodes, Journal of Systematics and Evolution, (2011), 49, p271-283.
[https://doi.org/10.1111/j.1759-6831.2011.00132.x]

- Lim, JD, Seong, ES, Choi, KJ, Kim, SK, Kim, MJ, Yu, CY, Intraspecific relationship analysis of Eleutherococcus senticosus Max by RAPD markers, Korean Journal of Plant Resources, (2000), 13, p104-110.
- Lim, JM, Ahn, YS, Park, CG, Park, CB, Cho, JH, Authentication of traded medicinal herb, Glycyrrhiza spp (Licorice). based on nrDNA-ITS2 sequence analysis, Journal of the Korean Society of International Agriculture, (2012), 24, p435-443.
-
Lim, JM, Kim, MS, Byeon, JH, Park, HS, Ahn, YS, Park, CG, Cho, JH, Classification and discrimination of geographical origin of Bang-Poong[Saposhnikovia divaricata (Turcz) Schischkin] medicinal plant and related pecies by using DNA sequence analysis, Journal of the Korean Society of International Agriculture, (2013), 25, p395-405.
[https://doi.org/10.12719/ksia.2013.25.4.395]

-
Lim, SH, Park, YH, Kwon, CJ, Ham, HJ, Jeong, HN, Kim, KH, Ahn, YS, Anti-diabetic and hypoglycemic effect of Eleutherococcus spp, Journal of the Korean Society of Food Science and Nutrition, (2010), 39, p1761-1768.
[https://doi.org/10.3746/jkfn.2010.39.12.1761]

-
Little, DP, A DNA mini-barcode for land plants, Molecular Ecology Resources, (2014), 14, p437-446.
[https://doi.org/10.1111/1755-0998.12194]

-
Ma, HL, Zhu, ZB, Zhang, XM, Miao, YY, Guo, QS, Species identification of the medicinal plant Tulipa edulis (Liliaceae) by DNA barcode marker, Biochemical Systematics and Ecology, (2014), 55, p362-368.
[https://doi.org/10.1016/j.bse.2014.03.038]

- Ministry of Food and Drug Safety(MFDS), The Korean herbal pharmacopoeia, Ministry of Food and Drug Safety. Seoul. Korea, (2013), 4th ed, p157-158.
- Ministry of Food and Drug Safety(MFDS), The Korean pharmacopoeia, Ministry of Food and Drug Safety. Seoul. Korea, (2015), 10th ed, -1839.
- Saitou, N, Nei, M, The neighbor-joining method A new method for reconstructing phylogenetic trees, Molecular Biology and Evolution, (1987), 4, p406-425.
-
Tamura, K, Nei, M, Kumar, S, Prospects for inferring very large phylogenies by using the neighbor-joining method, Proceedings of the National Academy of Sciences of the USA, (2004), 101, p11030-11035.
[https://doi.org/10.1073/pnas.0404206101]