
차세대염기서열분석법을 이용한 잔대의 SSR 마커 개발
© The Korean Society of Medicinal Crop Science. All rights reserved.
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License ( http://creativecommons.org/licenses/by-nc/3.0 ) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Adenophora triphylla var. japonica (Regel) H. Hara shows vegetative growth with radical leaves during the first year and shows reproductive growth with cauline leaves and bolting during the second year. In addition, the shape of the plant varies within the same species. For this reason, there are limitations to classifying the species by visual examination. However, there is not sufficient genetic information or molecular tools to analyze the genetic diversity of the plant.
Approximately 34.59 Gbp of raw data containing 342,487,502 reads was obtained from next generation sequencing (NGS) and these reads were assembled into 357,211 scaffolds. A total of 84,106 simple sequence repeat (SSR) regions were identified and 14,133 primer sets were designed. From the designed primer sets, 95 were randomly selected and were applied to the genomic DNA which was extracted from five plants and pooled. Thirty-nine primer sets showing more than two bands were finally selected as SSR markers, and were used for the genetic relationship analysis.
The 39 novel SSR markers developed in this study could be used for the genetic diversity analysis, variety identification, new variety development and molecular breeding of A. triphylla.
Keywords:
Adenophora triphylla, Genetic Diversity, Molecular Breeding, Next Generation Sequencing, SSR Marker서 언
잔대 [Adenophora triphylla var. japonica (Regel) H. Hara]는 초롱꽃과에 속하는 타식성 식물이며 전 세계적으로 약 60 여 종이 분포하지만 주로 아시아권에서 자생하며 그 중 약 30 여 종이 우리나라에 자생하고 있는 것으로 알려져 있다 (Lee et al., 2006). 잔대는 같은 종이라 하더라도 개체 간에 다양한 형태적 특성을 나타내고 그 유전형질 또한 다양하다고 보고되었다 (Lee et al., 1988; Lee, 1989). 예로부터 알려진 약용식물인 잔대 (사삼)의 효능은 주로 폐 기능에 도움을 주 며, 강한 해독능력이 있다고 전해지고 있다 (Shin and Seo, 2000).
현재까지 약용식물에 대한 연구는 물질분석을 통한 이차대 사산물의 이용과 같은 성분에 대한 연구와 DNA barcode기법 을 통한 종판별이나 randomly amplified polymorphic DNA (RAPD)를 이용한 유연관계 분석 등의 연구가 주로 이루어 졌다. 최근 들어 한약재의 기원이 되는 식물에 대해 분자수준 에서의 분류학적 연구가 활발해지고 있으며 유전정보의 중요 성이 대두되면서 next generation sequencing (NGS)을 통한 유전체 분석 및 유전적 다양성분석과 이를 기초로 하는 분자 육종의 필요성이 대두되고 있다 (Eu et al., 2009; Lee et al., 2010).
분자마커는 생물마다 가지고 있는 DNA의 염기서열 정보에 기반하여 중합효소연쇄반응 (polymerase chain reaction, PCR) 결과물의 길이 차이를 비교하거나 염기서열의 차이를 이용하 여 종 혹은 집단을 비교 분석하는 방법이다. 좋은 마커의 요 건으로는 다형성을 가지고 있으며 공우성적으로 검출할 수 있 고, 대상으로 하는 식물의 유전체 전체에 고르게 분포하며 변 이를 탐색하는데 있어 저렴한 비용으로 짧은 시간에 분석이 가능할 뿐 아니라 재현성이 높게 나타나는 것이 좋다.
분자마커의 대표적인 종류로는 RAPD, restriction fragment length polymorphism (RFLP), amplified fragment length polymorphism (AFLP), single nucleotide polymorphism (SNP), simple sequence repeat (SSR) 등이 있다. 이중에서 SSR마커는 식물체의 유전체에 존재하는 단순반복 염기서열의 반복횟수의 차이를 이용하는 마커이며 다른 마커보다 다형성 정도가 높고 공우성을 나타내기 때문에 검정 계통의 이형접합 도 여부를 쉽게 확인할 수 있는 등의 장점이 있다 (McCouch et al., 1997; Coburn et al., 2002). 실제로 국제식물신품종보호 연맹 (International Union for the Protection of New Varieties of Plants, UPOV) 내의 분자생물학실 무작업반 (Working Group on Biochemical and Molecular Techniques and DNAProfiling in Particular, BMT) 내에서도 품종식별에 SSR마커가 가장 효과적임을 제안한 바 있다 (UPOV-BMT, 2002).
최근 들어 한약재의 기원이 되는 식물 종을 대상으로 분자 수준에서 식물 분류학적 측면의 연구가 많이 이루어지고 있으 며 (Eu et al., 2009; Lee et al., 2010), 종 다양성 및 종간 계통 분류학적 유연관계 분석을 위해 특정한 유전자 부위의 염 기서열이나 유전체를 이용한 DNA fingerprinting을 이용한 연 구가 다수 보고되었다 (Kress et al., 2005; Weising et al., 2005).
최근 NGS 기술의 발전은 대량의 마커 발굴을 가능하게 하 였으며 (Mardis, 2008; Wang et al., 2009), 그 결과 옥수수, 밀, 벼 등 주요 식량작물이나 채소, 화훼에서는 육종기술의 발 달과 마커의 분자 표지 개발에 관한 연구가 활발히 이루어졌 다 (Nybom, 2004; Henry, 2012; Kilian and Graner, 2012). 그러나, 약용작물의 종내 다양성에 대한 연구는 더덕 (Kim et al., 2016)과 같은 주요 약용작물을 제외하면 분자수준의 연구 가 매우 미미한 실정이다.
본 연구에서는 국내의 여러 지역에 걸쳐 자생하며 재배, 유 통되고 있는 잔대를 대상으로 NGS를 통한 SSR마커를 개발 하고 이를 이용하여 잔대의 유전적 다양성을 분석하여 향후 육종에 필요한 연구 기반을 마련하고자 수행하였다.
재료 및 방법
1. SSR 구간 탐색 및 프라이머 제작
본 실험의 NGS 분석에는 강원도 평창에서 수집된 유전자 원 PC001에 속하는 한 개체를 임의로 선택하였으며, SSR마 커의 효율성 검정과 유전다양성 분석을 위한 재료는 평창, 보 은, 임실, 진안, 나주, 곡성, 화순 등 7 개 지역에서 뿌리를 수집 후 농촌진흥청 국립원예특작과학원 인삼특작부에서 증식 한 총 23 유전자원을 사용하였다 (Table 1).
NGS는 Illumina Hiseq 2500 플랫폼 (Illumuna, San Diego, CA, USA)을 이용하여 수행하였으며, 이를 통해 얻은 핵산조각을 Gil 등 (2017)의 방법에 따라 SOAPdenovo2 version 2.04의 SOAPec을 이용하여 error를 보정한 후 SOAPdenovo2를 이용하여 조합하였다. Microsatellites, transposable elements, rDNAs 등과 같은 반복서열은 RepeatMasker version 4.0.5과 RepeatModeler version 1.0.8 ( http://www.repeatmasker.org/ )을 이 용하여 분석하였다. 또한 SSR Finder ( ftp://ftp.gramene.org/pub/ gramene/arch-ives/software/scripts/ssr.pl )를 사용하여 SSR 구간 을 탐색하였다. SSR 구간을 탐색하기 위해 2 가지의 변수를 고려하였다. 첫 번째는 2 개에서 6 개의 염기쌍 (base pairs) 이 직렬로 최소 5 회 이상 반복되는 염기서열을 우선 선택하였 고, 두 번째는 반복되는 motif에 변이가 있는 것은 제외하였다. Primer3 program (Untergasser et al., 2012)을 이용하여 SSR 프라이머 서열을 다음과 같은 조건에서 디자인하였다. 길이는 18 bp에서 26 bp (최적 길이 23 bp)이고 GC함량은 50%에서 70% (최적 60%)로 하였으며, Tm값은 55℃에서 62℃ (최 적 58℃)로 설정하였다. PCR product의 길이는 150 bp에서 250 bp 사이가 되게 설계하되, 프라이머쌍 사이에 서로 다른 motif의 반복구간이 들어간 것들은 모두 제외하였다.
2. 다형성 SSR 프라이머 선발
본 연구에서 수집된 유전자원 중 5 개 지역 (평창, 보은, 나주, 임실, 화순)에서 각각 1 개체씩을 임의로 선별하고 genomic DNA를 추출한 후, 이를 동량으로 혼합하는 genomic DNA pooling 방법을 이용하여 혼합된 genomic DNA를 95 개의 프라이머에 적용하였다. 그 후, Fragment Analyzer Automated CE system (Advanced Analytical Technologies, Ankeny, IA, USA) 기기를 이용하여 Quant-iT PicoGreen dsDNA reagent kit, 1-500 bp (Invitrogen Co., Carlsbad, CA, USA)로 전기영동을 수행하였다. 이후, 증폭된 밴드는 PROSize version 2.0 software (Advanced Analytical Technologies, Ankeny, IA, USA) 프로그램을 이용하여 분석 하였다. 이를 바탕으로, 결과 값이 뚜렷하고, PCR product의 증폭 길이가 예상되었던 수치와 비슷하며, 또한 2 개 이상의 밴드를 보이는 프라이머를 선별하였다.
3. SSR 마커의 효율성 검증
선발된 마커를 수집된 자원에 적용하여 개발된 마커의 효율 성을 검정하였다. 이를 위해서 선발된 프라이머를 총 23 개체 에 각각 적용하였고, 증폭된 PCR 산물을 DNA Fragment Analyzer Automated CE System (Advanced Analytical Technologies, Ankeny, IA, USA)을 이용하여 분석하였다. 그 결과를 마커 분석 프로그램인 Power Marker software (Version 3.23) (Botstein et al., 1980; Liu and Muse 2005) 를 이용하여 선발된 SSR 마커의 효율성을 분석하였다.
결과 및 고찰
1. 염기서열 분석 및 SSR 구간 탐색과 프라이머 제작
본 연구에서는 Illumina Hiseq 2500 platform을 이용하여 염기서열 분석을 수행하였다. Paired-end sequencing 방법을 이용하여 총 342,487,502 개의 read를 포함하는 총 34.59 Gbp의 염기서열 정보를 생산하였고, GC의 비율은 41.37% 였다. 생산된 염기서열로부터 총 357,211 개의 scaffold를 얻 었고, N50은 1,459 bp였다.
이렇게 얻어진 scaffold로부터 SSR 구간을 검색하고 primer 를 제작하였다. 검색된 SSR구간은 총 84,106 개로 assembly 된 전체 염기서열의 0.73%를 차지하였으며, 그 중 dinucleotide가 70,497 개, tri-nucleotide가 9,998 개, tetranucleotide가 1,263 개, penta-nucleotide가 752 개, hexanucleotide가 757 개, hepta-nucleotide가 87 개를 차지하였다 (Table 2, Table 3).
탐색된 SSR 구간에서 서로 다른 SSR motif가 없도록 14,133 개의 SSR 프라이머를 설계하였으며, 이 중에서 임의로 tri-nucleotide 31 개, tetra-nucleotide 40 개, penta-nucleotide 24 개를 포함한 총 95 개의 프라이머 세트를 제작하였다.
2. 다형성을 보이는 SSR 프라이머 선발
제작된 95 개의 프라이머 세트 중 5 개체를 이용하여 다형 성을 조사한 결과 총 39 개의 프라이머 세트가 선발되었다 (Table 4). 선발된 39 개의 프라이머 세트를 이용하여 23 개 의 자원에서 유전적 다양성을 분석한 결과 allele의 수가 2 개 에서 12 개로 나타나 명확한 다형성을 보였으며 (Table 5), 이 SSR 프라이머 세트는 잔대 [Adenophora triphylla var. japonica (Regel) H. Hara]의 SSR 마커로 활용하기에 매우 적합하다고 판단되었다. 이들 마커의 motif 종류는 tri motif 11 개, tetra motif 17 개, penta motif 11 개 였으며, 결과적 으로 motif 별로 고르게 마커가 개발되었음을 알 수 있었다.
3. SSR 마커의 효율성 검증
개발된 마커의 효율성을 검증하기 위하여 23 개체에 적용하 여 얻은 총 263 개의 allele을 분석하였다. Frequency of major alleles (MAF)은 0.17 - 0.87 (평균 0.46) 이었고, genotype number (GN)는 의 값은 2 - 18 (평균 9.9)였으며, heterozygosity (HE)는 0.23 - 0.88 (평균 0.68)이었다. Observed heterozygosity (HO)의 값은 0.24 - 1.00 (평균 0.59)였으며, polymorphism information content (PIC)의 값은 0.20 - 0.87 (평균 0.64)이었 다. PIC 값이 가장 컸던 마커는 Atri00959였으며, 이 마커는 PIC값과 HE 뿐만 아니라 GN도 높게 나타났다 (Table 5). 위 결과를 바탕으로 적용된 39 개의 마커는 잔대의 유전자원 분 석에 적합하다고 사료되었다.
39 개의 SSR마커를 이용하여 얻은 결과를 바탕으로 국내 각 지역에서 수집된 잔대 유전자원 23 개에서 유전자형을 분 석하여 계통학적 분석을 한 결과 수집지역별로 그룹이 만들어 지지 않고 섞여서 만들어지는 것을 알 수 있었다 (Fig. 1). 또 한, 한 지역에서 수집된 자원이라도 다시 세부 그룹으로 나뉘 어져, 잔대는 유전적으로 다양하다는 것을 잔대 유전체 정보 를 바탕으로 개발된 SSR 마커를 이용하여 확인할 수 있었다. 또한, 개발된 마커의 평균 PIC 값이 0.64인 것으로 볼 때 본 연구에서 개발된 잔대의 SSR 마커는 유전자형의 다양성을 확 인하는데 우수한 마커임을 알 수 있었다.
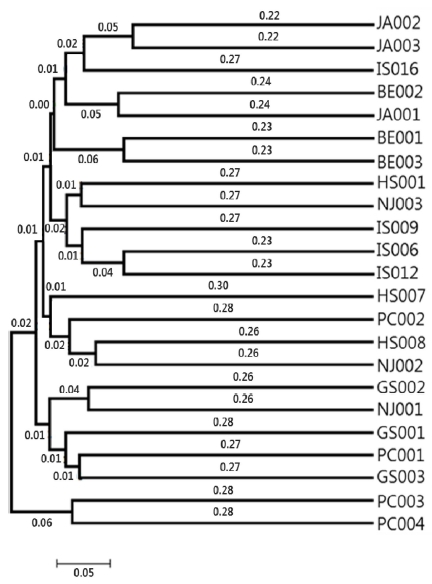
Phylogenetic tree for 23 Adenophora triphylla collections using 39 SSR markers.PC; Gangwondo Pyeongchang-gun 2015, BE; Chungcheongbukdo Boeun-gun 2015, IS; Jeollabuk-do Imsil-gun 2016, JA; Jeollabuk-do Jinan-gun 2015, NJ; Jeollanam-do Naju-si 2016, GS; Jeollanam-do Gokseong-gun 2015, HS; Jeollanam-do Hwasungun 2016. The genetic distances between strains were calculated based on the shared allelic methods (Jin and Chakraborty, 1994) using PowerMarker v3.25 software. The values on each branch indicate the length of the branch. The scale bar indicates the branch length.
지금까지 잔대 (사삼)에서는 항산화와 비만 등에 관련된 성 분물질 분석, 약리활성에 대한 연구가 꾸준하게 이루어져 왔 지만 (Lee et al., 2015; Kim, 2016), 잔대의 유전적 다양성 이나 신품종 육성을 위한 유전적 연구는 RAPD나 DNA barcoding 등을 통해 이루어진 속간 유연관계의 포괄적인 규 명에 국한되었고, 더구나 잔대속의 경우 속간 유전적 분자표 지의 연구나 종내 다양성에 대한 연구는 매우 한정적이어서 (Eddie et al., 2003; Cheon and Yoo, 2013), 확보한 잔대 유전자원의 다양성 확인 및 체계적 관리, 계통 분류 등에 많 은 어려움이 있었다.
본 연구에서 개발된 SSR 마커는 잔대의 유전체 정보를 기 반으로 한 것으로써, 잔대의 유전적 다양성을 염기서열 수준 에서 밝히는데 매우 유용하다는 것을 알 수 있었다. 또한, 이 를 이용하여 국내 잔대 유전자원의 다양성을 성공적으로 파악 함으로써, 잔대의 체계적인 계통 분류 및 분자 육종에 필요한 기본적 자료를 제공하였다. 본 연구에서 개발된 마커는 앞으 로 국내외 잔대 유전자원의 확보 및 다양성 분석, 우수 형질 의 잔대 품종 개발에도 도움을 줄 것으로 사료된다.
감사의 글
본 연구는 농촌진흥청 연구사업(과제번호: PJ0102882017, PJ01102202)의 지원에 의해 이루어진 것으로 이에 감사드립 니다.
References
- D. Botstein, R.L. White, M. Skolnick, R.W. Davis, Construction of a genetic linkage map in man using restriction fragment length polymorphisms., Am. J. Hum. Genet, (1980), 32, p314-331.
-
K.S. Cheon, K.O. Yoo, Phylogeny of Hanabusaya (Campanulaceae), a Korean endemic, based on ITS sequences of nuclear ribosomal DNA., J. Syst. Evol, (2013), 51, p704-714.
[https://doi.org/10.1111/jse.12039]

- J.R. Coburn, S.V. Temnykh, E.M. Paul, S.R. McCouch, Design and application of microsatellite marker panels for semiautomated genotyping of rice(Oryza sativa L.)., Crop Sci, (2002), 42, p2092-2099.
-
W.M. Eddie, T. Shulkina, J. Gaskin, R.C. Haberle, R.K. Jansen, Phylogeny of Campanulaceae S. Str. inferred from ITS sequences of nuclear ribosomal DNA., Ann. Mo. Bot. Gard, (2003), 90, p554-575.
[https://doi.org/10.2307/3298542]

- G.S. Eu, M.R. Park, S.J. Yun, Internal transcribed spacer(ITS) regions reveals phylogenic relationships of Rubus species cultivated in Korea., Hanguk Yakyong Changmul Hakhoe Chi, (2009), 17, p165-172.
-
J. Gil, Y. Um, S. Kim, O.T. Kim, S.C. Koo, C.S. Reddy, S.C. Kim, C.P. Hong, S.G. Park, H.B. Kim, D.H. Lee, B.H. Jeong, J.W. Chung, Y. Lee, (2017), Development of genome-wide SSR markers from Angelica gigas Nakai using next generation sequencing., Genes, 8, p238, http://www.mdpi.com/2073-4425/8/10/238/htm.
[https://doi.org/10.3390/genes8100238]

-
R.J. Henry, Next-generation sequencing for understanding and accelerating crop domestication., Brief. Funct. Genomics, (2012), 11, p51-56.
[https://doi.org/10.1093/bfgp/elr032]

- L. Jin, R. Chakraborty, Estimation of genetic distance and coefficient of gene diversity from single-probe multilocus DNA fingerprinting data., Mol. Biol. Evol, (1994), 11, p120-127.
-
B. Kilian, A. Graner, NGS technologies for analyzing germplasm diversity in genebanks., Brief. Funct. Genomics, (2012), 11, p38-50.
[https://doi.org/10.1093/bfgp/elr046]

-
H.K. Kim, Adenophora remotiflora protects human skin keratinocytes against UVB-induced photo-damage by regulating antioxidative activity and MMP-1 expression., Nutr. Res. Pract, (2016), 10, p371-376.
[https://doi.org/10.4162/nrp.2016.10.4.371]

- S. Kim, J.H. Jeong, H. Chung, J.H. Kim, J. Gil, J. Yoo, Y. Um, O.T. Kim, T.D. Kim, Y.Y. Kim, D.H. Lee, H.B. Kim, Y. Lee, Simple sequence repeat marker development from Codonopsis lanceolata and genetic relation analysis., J. Plant Biotechnol, (2016), 43, p181-188.
-
W.J. Kress, K.J. Wurdack, E.A. Zimmer, L.A. Weigt, D.H. Janzen, Use of DNA barcodes to identify flowering plants., Proc. Natl. Acad. Sci. USA, (2005), 102, p8369-8374.
[https://doi.org/10.1073/pnas.0503123102]

- D.R. Lee, Y.S. Lee, B.K. Choi, H.J. Lee, S.B. Park, T.M. Kim, H.J. Oh, H.Y. Yang, J.W. Suh, Roots extracts of Adenophora triphylla var. japonica improve obesity in 3T3-L1 adipocytes and high-fat diet-induced obese mice., Asian Pac. J. Trop. Med, (2015), 8, p898-906.
- J.H. Lee, I.H. Jo, J.W. Lee, C.G. Park, K.H. Bang, H.S. Kim, C.B. Park, Molecular authentication of Scrophularia herbs by PCR-RFLP based on rpl-5 region of mitochondrial DNA., Hanguk Yakyong Changmul Hakhoe Chi, (2010), 18, p173-179.
- J.K. Lee, Taxonomy of the genus Adenophora (Adenophorae) in Korea. Master Thesis, (1989), Sungkyunkwan University.
-
S.T. Lee, Y.J. Chung, J.K. Lee, A palynotaxonomic study of the Korean Campanulaceae., Korean J. Plant Taxon, (1988), 18, p115-131.
[https://doi.org/10.11110/kjpt.1988.18.2.115]

- S.Y. Lee, S.K. Lee, J.K. Lee, K.H. Kim, S.K. Lee, Cultural characteristics and genetic diversity of Rhizina undulata isolates by random amplified polymorphic DNA(RAPD)., J. Korean For. Soc, (2006), 95, p388-392.
-
K.J. Liu, S.V. Muse, PowerMarker: An integrated analysis environment for genetic marker analysis., Bioinformatics, (2005), 21, p2128-2129.
[https://doi.org/10.1093/bioinformatics/bti282]

- E.R. Mardis, Next-generation DNA sequencing methods., Annu. Rev. Genomics Hum. Genet, (2008), 9, p387-402.
- S.R. McCouch, X. Chen, O. Panaud, S. Temnykh, Y. Xu, Y.G. Cho, N. Huang, T. Ishii, M. Blair, Microsatellite marker development, mapping and applications in rice genetics and breeding., Plant Mol. Biol, (1997), 35, p89-99.
-
H. Nybom, Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants., Mol. Ecol, (2004), 13, p1143-1155.
[https://doi.org/10.1111/j.1365-294x.2004.02141.x]

- D.H. Shin, Y.B. Seo, Bibliographical studies on the shashen(Adenophora triphylla var. japonica Hara.)., Journal of Daejeon Oriental Medicine, (2000), 8, p107-122.
-
A Untergasser, I Cutcutache, T Koressaar, J Ye, BC Faircloth, M Remm, SG Rozen, (2012), Primer3-new capabilities and interfaces.
[https://doi.org/10.1093/nar/gks596]

- UPOV-BMT, C/36/10 Progress report of the 36th session of the technical committee, the technical working parties and working group on biochemical and molecular techniques and DNA-profiling in particular, (2002), Geneva, Switzerland.
-
Z. Wang, M. Gerstein, M. Snyder, RNA-Seq: A revolutionary tool for transcriptomics., Nat. Rev. Genet, (2009), 10, p57-63.
[https://doi.org/10.1038/nrg2484]

- K. Weising, H. Nybom, K. Wolff, G. Kahl, DNA fingerprinting in plants: Principles, methods, and applications, 2nd ed, Boca Raton, FL, USA, CRC Press, (2005), p21-146.
