
오미자과 식물 종 판별을 위한 엽록체 기반 InDel 마커의 개발
 ; Jin Su Gil3
; Jin Su Gil3 ; Chang Pyo Hong4
; Chang Pyo Hong4 ; Sang Ho Kang5 ; Soo Jin Kwon6 ; Hyo Jin Kim7 ; Chang Kug Kim8 ; Jung Ho Lee9 ; Yi Lee10, †
; Sang Ho Kang5 ; Soo Jin Kwon6 ; Hyo Jin Kim7 ; Chang Kug Kim8 ; Jung Ho Lee9 ; Yi Lee10, †
This is an open access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
The three Schisandracae species, Schisandra chinensis, Schisandra repanda, and Kadsura japonica, which are used as raw materials for the production of herbal medicines, are readily confused. However, of these three species, only S. chinensis is formally registered as a medicinal herb in the Korean Pharmacopeia. In order to facilitate the clear identification of S. chinensis, S. repanda, and K. japonica, we developed insertion or deletion (InDel) markers by comparing the chloroplast sequences of the three species.
Genomic DNA was extracted from the leaves of S. chinensis, S. repanda, and K. japonica, and next generation sequencing (NGS) analysis was performed using the Illumina HiSeq-2500 platform. Chloroplast sequences for the three species were obtained and compared to identify polymorphic loci. Primers were designed for those InDel loci showing polymorphism in all three species, and four InDel markers were accordingly developed and successfully used for species discrimination.
In this study, we developed four markers that can be used to discriminate S. chinensis, S. repanda, and K. japonica at the molecular level.
Keywords:
Kadsura japonica, Schisandra chinensis, Schisandra repanda, Chloroplast, Insertion or Deletion Marker서 언
오미자는 오미자과 (Schisandraceae)에 속하는 덩굴성 식물이다. 오미자과는 Schisandra 속, Illicium 속, Kadsura 속 등 3 속을 포함하며 (APG III, APG IV), 이들 3 속은 대한민국에 분포하고 있다 (Lee, 1979; APG, 2009, 2016). Schisandra 속에는 낙엽활엽 덩굴성 식물인 오미자 [Schisandra chinensis (Turcz.) Baill.]와 흑오미자 [(Schisandra repanda (Siebold & Zucc.) Radlk.)] 등 2 종의 식물이 있다 (Lee, 1979).
흑오미자의 학명은 S. repanda이나 S. nigra라는 이명을 갖고 있으며 암수 딴그루인 식물이다 (Kim et al., 1999). Kadsura 속에는 상록활엽 덩굴성 식물인 남오미자 [Kadsura japonica (L.) Dunal] 1 종의 식물이 있다 (Lee, 1979). Illicium 속에는 붓순나무 (I. anisatum) 1 종이 우리나라에 자생하고 있다.
대한민국 약전에서 오미자는 잘 익은 오미자 (S. chinensis) 열매로 규정하고 있으며 쉬잔드린 (C24H32O7 :MW. 432.51), 고미신 A (C23H28O7 :MW. 416.46) 및 고미신 N (C23H28O6 : MW. 400.47)의 합이 0.7% 이상 함유되어야 한다고 되어 있으나 흑오미자 및 남오미자는 한약재 원료로써의 사용이 규정되지 않고 있다 (KFDA, 2012).
붓순나무의 열매는 다른 3 종의 식물과 현저하게 다른 형태를 가지고 있으나 한약재로 이용되는 오미자는 흑오미자 및 남오미자의 건조된 열매와 육안에 의한 판별이 어려워 원료의 혼·오용 문제가 야기될 수 있으며 (Kim et al., 1999), 이를 방지하기 위해 분자생물학적 기법에 의한 판별이 필요하다.
지금까지 우리나라에서 오미자에 대한 연구는 주로 오미자 열매에서 생리활성 및 효능 중심으로 진행되었으며 (Han et al., 2019), 분자 생물학적으로 오미자과의 종 판별에 관한 연구로는 RAPD (random amplified polymorphic DNA) 유래 SCAR (sequence characterized amplified region) marker 및 multiplex-PCR 기법 개발, nrDNA-ITS 분자마커를 이용한 오미자 종 감별 등이 보고되어 있다 (Gao et al., 2003; Moon et al., 2010; Kim et al., 2012; Lee et al., 2013). SCAR marker 기반의 multiplex-PCR을 이용할 경우 다수의 primer를 사용해야하며 증폭이 제대로 이뤄지지 않을 수 있는 단점이 있다 (Henegariu et al., 1997; Lee et al., 2013). 또한 SNP (single nucleotide polymorphism)는 InDel (insertion or deletion) 마커에 비해 분석 시 고가의 장비가 필요하기 때문에 최소한의 실험장비로 크기에 따라 쉽게 구별할 수 있는 InDel 마커가 선호되고 있다 (Väli et al., 2008; Kim et al., 2012; Li et al., 2014; Moghaddam et al., 2014; Lv et al., 2016).
NGS (next generation sequencing) 기술의 발전으로 sequencing과 assembly의 비용과 작업의 복잡성이 줄었으며 식물의 유전 및 육종 연구에 사용할 수 있는 마커를 개발할 수 있는 염기서열을 제공하며 (Moore et al., 2006; Varshney et al., 2009a; Varshney et al., 2009b), 이러한 NGS 기술 바탕으로 엽록체 게놈은 농업에서 품종을 식별하는데 유용한 도구로 사용된다 (Daniell et al., 2016).
식물에서 주로 모계 유전을 하는 엽록체 DNA는 핵 DNA 염기 서열에 비해 진화가 느린 것으로 알려져 있어 식물의 계통학적 연구에 사용되기에 적합하며 (Wolfe et al., 1987; Clegg et al., 1994; Ngan et al., 1999), 이러한 엽록체에서 유전적 변이를 발견하고 엽록체 유전체를 기반으로 한 종의 판별이 보고되고 있다 (Liu et al., 2012; Kim et al., 2015; Park et al., 2017; Song et al., 2017).
기존 종 식별을 위한 엽록체 기반 InDel 마커 연구 사례는 Angelica polymorpha의 엽록체 게놈의 염기서열을 분석하여 InDel 마커를 개발하여 종 식별에 사용되었다 (Park et al., 2019). 이외에도 벼 (Steele et al., 2008), 토마토 (Liu et al., 2017), 차나무 (Liu et al., 2019) 에서 InDel 마커를 품종의 식별에 이용한 보고가 있었다.
본 연구에서는 국내에 자생하고 있는 오미자, 흑오미자 및 남오미자에서 획득한 엽록체 염기서열 정보를 바탕으로 종간의 다형성을 확인할 수 있는 InDel 마커를 개발하였다. 이렇게 개발된 마커는 혼용되기 쉬운 3 종의 오미자를 쉽게 구분하고 한약재 유통과정의 원료 혼·오용을 방지하며 오미자, 흑오미자 및 남오미자의 계통학적 관계를 분석하거나 새로운 품종을 개발하는 데에도 이용될 수 있을 것으로 기대된다.
재료 및 방법
1. 실험재료
본 실험에서 사용된 오미자 (Schisandraceae)는 전라북도농업기술원 약용자원연구소에서 육성중인 만생종, 중생종, 조생종, 재래종 각 3 개체씩 총 12 개체에서 잎을 채취하였으며 흑오미자 암, 수그루와 남오미자는 3 개체씩 9 개체를 제주특별자치도 농업기술원에서 채취하여 -70℃의 초저온 냉동고에 보관하여 DNA 추출에 사용하였다 (Table 1).
2. DNA 추출
DNA 추출을 위해 21 개체의 잎 시료를 액체질소로 급냉시키고 막자사발을 이용하여 분쇄한 후 DNeasy Plant Mini Kit (QIAGEN GmbH, Hilden, Germany)를 사용하여 제조사에서 제시하는 방법에 따라 추출하였다.
추출된 DNA는 1% agarose gel에서 전기영동 하여 확인한 후 Nanodrop (Thermo Fisher Scientific Inc., Waltham, MA, USA) 기기를 이용하여 농도를 측정한 후 nuclease-free water를 이용하여 5 ng/㎕로 희석하였다.
3. InDel 마커 탐색
오미자의 엽록체 염기서열 확보를 위해 추출한 오미자, 흑오미자 및 남오미자의 DNA를 Illumina HiSeq 2500 platform (Illumina Inc., San Diego, CA, USA)을 사용하여 염기서열을 분석하였다. 생산된 염기서열은 CLC Genomics Workbench 11.0 (QIAGEN GmbH, Hilden, Germany) 프로그램을 통해 조립한 뒤 InDel 구간을 탐색하여 프라이머를 제작하였다. 프라이머의 조건은 길이는 18 bp - 22 bp, 온도는 50℃ - 60℃, G/C비율은 50%, product size range는 150 bp - 400 bp로 하였다.
4. PCR (polymerase chain reaction) 및 genotyping
PCR에 사용된 혼합용액 조성의 PCR mixture 총 부피는 20㎕로 5 ng/㎕ genomic DNA 2㎕, 2 × Taq mix (Dongsheng Biotech Co., Ltd., Guangzhou, China) 10㎕, nucleasefree water 6 ㎕, 5 μM forward 및 reverse primer 각각 1 ㎕로 구성하였고 T100 Thermal Cycler (Bio-Rad Inc., Hercules, CA, USA)를 사용하여 증폭하였다.
PCR 반응은 pre-denaturation 94℃ 3 분, denaturation 94℃ 30 초, annealing 54℃ - 55℃ 30 초, extension 72℃ 1분 총 34 회 반복하였으며 final extension 72℃, 10 분의 조건으로 수행하였다. PCR 증폭 산물은 3% agarose gel에서 120 V로 30 분 수행 하였으며 Gel DocTM XR+ Gel Documentation System (Bio-Rad Inc., Hercules, CA, USA)에서 증폭산물의 생성을 확인하였다.
오미자, 흑오미자 및 남오미자 자원 총 21 개체의 PCR 수행 후 생성된 증폭 산물은 DNA Fragment Analyzer Automated CE System (Advanced Analytical Technologies Inc., Ankeny, IA, USA)을 이용하여 길이 분석을 수행하였다. 분석된 data는 PROSize 3.0 프로그램 (Advanced Analytic Technologies Inc., Ankeny, IA, USA)을 이용해 사이즈를 측정하였다.
결과 및 고찰
1. 엽록체 기반 InDel 마커 개발
CLC Genomics Workbench ver. 11.0 프로그램 (QIAGEN GmbH, Aarhus, Denmark)을 이용하여 de novo assembly를 통해 조립된 오미자 (Schisandraceae) 염기서열로부터 sequencing depth와 BLAST (Basic Local Alignment Search Tool) 분석을 통하여 엽록체의 염기서열을 선발하고 이들 염기서열을 비교하여 InDel 구간을 탐색하였다. 총 4 개의 InDel 구간이 선발되어 해당 구간에서 primer를 디자인하였다 (Table 2).
2. 오미자과 식물의 종 판별
제작된 4 개의 프라이머 세트를 오미자 12 개체, 흑오미자 6 개체, 남오미자 3 개체에 적용하여 PCR 분석을 수행하였고 PCR 산물을 fragment analyzer를 이용하여 분석하여 size를 측정하였다.
그 결과 4 개의 마커는 모두 오미자, 흑오미자, 남오미자를 판별을 할 수 있었으며 SCcpInDel-1의 경우 오미자 서열을 기준으로 흑오미자는 24 bp, 남오미자는 7 bp의 염기서열이 결실되었으며, SCcpInDel-2는 오미자 서열 기준으로 흑오미자는 148 bp, 남오미자는 156 bp의 염기가 결실되었고, SCcpInDel-3 의 경우에는 오미자 서열을 기준으로 흑오미자는 20 bp의 염기가 첨가되었고, 남오미자는 33 bp의 염기가 결실되어 있었으며, SCcpInDel-4의 경우에는 오미자 서열 기준으로 흑오미자에서 27 bp, 남오미자에서 71 bp의 염기가 결실되어 있었다 (Fig. 1, Table 3).
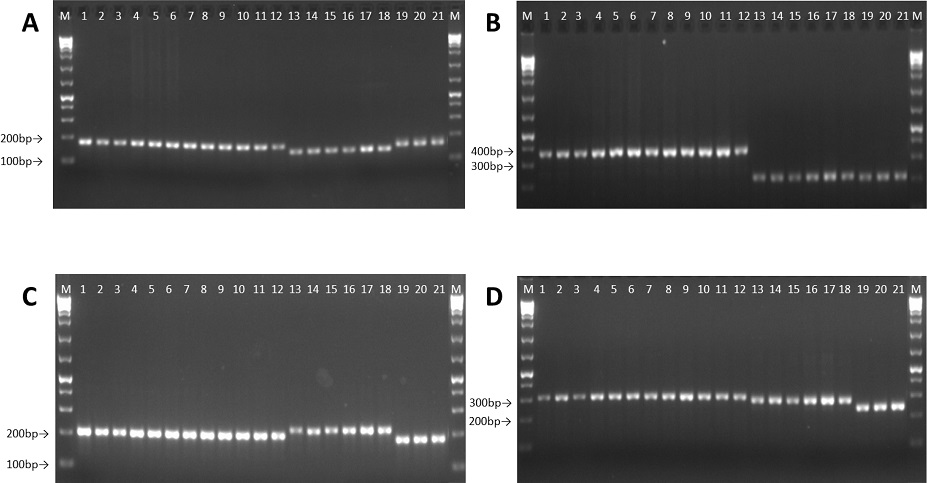
PCR analysis of 21 Schisandraceae (S. chinensis, S. repanda, K. japonica) plants using 4 InDel markers. A; SCcpInDel-1, B; SCcpInDel-2, C; SCcpInDel-3, D; SCcpInDel-4. M; 1 kb ladder plus size marker, lane 1; Sc-L1, lane 2; Sc-L2, lane 3; Sc-L3, lane 4; Sc-N1, lane 5; Sc-N2, lane 6; Sc-N3, lane 7; Sc-E1, lane 8; Sc-E2, lane 9; Sc-E3, lane 10; Sc-N1, lane 11; Sc-N2, lane 12; Sc-N3, lane 13; Sr-F1, lane 14; Sr-F2, lane 15; Sr-F3, lane 16; Sc-M1, lane 17; Sc-M2, lane 18; Sc-M3, lane 19; Kj-1, lane 20; Kj-2, and lane 21; Kj-3.
또한 SCcpInDel-1 과 SCcpInDel-2 마커는 각 염기 서열 차이가 크지 않아 agarose gel에서 분석하는데 한계가 있어 fragment analyzer를 이용하여 분석해야 하지만, SCcpInDel-3과 SCcpInDel-4 마커는 agarose gel에서 육안으로 차이를 확인할 수 있어 SCcpInDel-1과 SCcpInDel-2에 비해 빠른 시간에 유전자형을 확인할 수 있었다. 기존 오미자과 종판별 연구사례 (5Gao et al., 2003; Moon et al., 2010; Kim et al., 2012; Lee et al., 2013)에서 사용된 다소 복잡한 방법에 비해 이번 연구를 통해 개발된 SCcpInDel-3 과 SCcpInDel-4 마커는 빠른 시간 안에 오미자과 식물을 판별할 수 있을 것으로 보인다.
본 연구에서 개발된 4 개의 마커는 국내에 자생하고 있는 오미자과 식물인 오미자, 흑오미자 및 남오미자를 충분히 구분할 수 있었으며 PCR 증폭산물의 길이 차이가 커서 여러 가지 종이 혼합되어 있는 경우에도 어떤 종이 혼합되어 있는지를 판별할 수 있을 정도의 판별 능력을 보였다. 이 결과는 기존 오미자과 종판별 연구 사례 (Gao et al., 2003; Moon et al., 2010; Kim et al., 2012; Lee et al., 2013)에서 해결할 수 없었던 혼합물의 분석 등 많은 부분을 보완할 수 있을 것으로 보인다. 이 마커들은 혼용되기 쉬운 3 종의 오미자과 식물을 쉽게 구분하고 한약재 유통과정의 원료 혼·오용을 방지하며 오미자과 식물의 계통학적 관계를 분석하거나 새로운 품종을 개발하는 데에도 이용될 수 있을 것으로 기대된다.
Acknowledgments
본 연구는 농촌진흥청 연구사업(과제번호: PJ01349002)의 지원에 의해 이루어진 결과로 이에 감사드립니다.
References
-
Angiosperm Phylogeny Group(APG). (2009). An update of the Angiosperm Phylogeny Group classification for the orders and families of flowering plants: APG III. Botanical Journal of the Linnean Society. 161:105-121.
[https://doi.org/10.1111/j.1095-8339.2009.00996.x]

-
Angiosperm Phylogeny Group(APG). (2016). An update of the Angiosperm Phylogeny Group classification for the orders and families of flowering plants: APG IV. Botanical Journal of the Linnean Society. 181:1-20.
[https://doi.org/10.1111/boj.12385]

-
Clegg MT, Gaut BS, Learn JGH and Morton BR. (1994). Rates and patterns of chloroplast DNA evolution. Proceedings of the National Academy of Sciences. 91:6795-6801.
[https://doi.org/10.1073/pnas.91.15.6795]

-
Daniell H, Lin CS, Yu M and Chang WJ. (2016). Chloroplast genomes: Diversity, evolution, and applications in genetic engineering. Genome Biology. 17:134. https://link.springer.com/article/10.1186/s13059-016-1004-2, (cited by 2020 Oct 3).
[https://doi.org/10.1186/s13059-016-1004-2]

- Gao J, Wang Y, Qiao C and Chen D. (2003). Ribosomal DNA ITS sequences analysis of the Chinese crude drug fructus schisandrae sphenantherae and fruits of Schisandra viridis. China Journal of Chinese Materia Medica. 28:706-710.
-
Han SH, Jang JK, Ma KH, Kim YJ, Kim SM, Lee HJ and Hong CO. (2019). Selection of superior resources through analysis of growth characteristics and physiological activity of Schisandra chinensis collection. Korean Journal of Medicinal Crop Science. 27:9-16.
[https://doi.org/10.7783/KJMCS.2019.27.1.9]

-
Henegariu O, Heerema NA, Dlouhy SR, Vance GH and Vogt PH. (1997). Multiplex PCR: Critical parameters and step-by-step protocol. Biotechniques. 23:504-511.
[https://doi.org/10.2144/97233rr01]

-
Kim JH, Byeon JH, Park HS, Lee JH, Lee SW, Cha SW and Cho JH. (2015). Authentication of traded traditional medicine ogapi based on nuclear ribosomal DNA internal transcribed spacers and chloroplast DNA sequences. Korean Journal of Medicinal Crop Science. 23:489-499.
[https://doi.org/10.7783/KJMCS.2015.23.6.489]

-
Kim JS, Jang HW, Kim JS, Kim HJ and Kim JH. (2012). Molecular identification of Schisandra chinensis and its allied species using multiplex PCR based on SNPs. Genes and Genomics. 34:283-290.
[https://doi.org/10.1007/s13258-011-0201-3]

- Kim PG, Lee KY, Kim SH and Han SS. (1999). Foliar characteristics and photosynthetic efficiency of three species of Schisandraceae trees distributed in Korea. Korean Journal of Agricultural and Forest Meteorology. 1:90-96.
- Korea Food and Drug Administration(KFDA). (2012). The Korean pharmacopoeia. Korea Food and Drug Administration. Chungwon, Korea. p.76-77.
- Lee CB. (1979). Coloured flora of Korea. Hyangmoonsa. Seoul, Korea. p.375-376.
-
Lee YM, Moon BC, Ji YU, Seo HS and Kim HK. (2013). Development of RAPD-derived SCAR markers and multiplex-PCR for authentication of the Schisandrae Fructus. Korean Journal of Medicinal Crop Science. 21:165-173.
[https://doi.org/10.7783/KJMCS.2013.21.3.165]

-
Li Y, Liu B, Reif JC, Liu Y, Li H, Chang R and Qiu LU. (2014). Development of insertion and deletion markers based on biparental resequencing for fine mapping seed weight in soybean. The Plant Genome. 7:1-8.
[https://doi.org/10.3835/plantgenome2014.04.0014]

-
Liu S, An Y, Tong W, Qin X, Samarina L, Guo R, Xia X and Wei C. (2019). Characterization of genome-wide genetic variations between two varieties of tea plant(Camellia sinensis) and development of InDel markers for genetic research. BMC Genomics. 20:935. https://link.springer.com/article/10.1186/s12864-019-6347-0, (cited by 2020 Sep 20).
[https://doi.org/10.1186/s12864-019-6347-0]

-
Liu X, Geng X, Zhang H, Shen H and Yang W. (2017). Association and genetic identification of loci for four fruit traits in tomato using indel markers. Frontiers in plant science. 8:1269. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5515879/, (cited by 2020 Sep 20).
[https://doi.org/10.3389/fpls.2017.01269]

-
Liu Y, Zhang L, Liu Z, Luo K, Chen S and Chen K. (2012). Species identification of Rhododendron(Ericaceae) using the chloroplast deoxyribonucleic acid psbA-trnH genetic marker. Pharmacognosy Magazine. 8:29-36.
[https://doi.org/10.4103/0973-1296.93311]

-
Lv Y, Liu Y and Zhao H. (2016). mInDel: A high-throughput and efficient pipeline for genome-wide InDel marker development. BMC Genomics. 17:290. https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-016-2614-5, (cited by 2020 Sep 19).
[https://doi.org/10.1186/s12864-016-2614-5]

-
Moghaddam SM, Song Q, Mamidi S, Schmutz J, Lee R, Cregan P, Osorno JM and McClean PE. (2014). Developing market class specific InDel markers from next generation sequence data in Phaseolus vulgaris L. Frontiers in Plant Science. 5:185. https://www.frontiersin.org/articles/10.3389/fpls.2014.00185/full, (cited by 2020 Sep 19).
[https://doi.org/10.3389/fpls.2014.00185]

- Moon BC, Ji YU, Seo HS, Lee AY, Chun JM and Kim HK. (2010). Molecular authentication of schisandrae fructus and analysis of phylogenetic relationship based on nrDNA-ITS sequences. Korea Journal of Herbology. 25: 47-54.
-
Moore MJ, Dhingra A, Soltis PS, Shaw R, Farmerie WG, Folta KM and Soltis DE. (2006). Rapid and accurate pyrosequencing of angiosperm plastid genomes. BMC Plant Biology. 6:17. https://link.springer.com/article/10.1186/1471-2229-6-17, (cited by 2020 Sep 19).
[https://doi.org/10.1186/1471-2229-6-17]

-
Ngan F, Shaw P, But P and Wang J. (1999). Molecular authentication of Panax species. Phytochemistry. 50:787-791.
[https://doi.org/10.1016/S0031-9422(98)00606-2]

-
Park IK, Yang SY, Kim WJ, Song HJ, Lee HS, Lee HO, Lee JH, Ahn SN and Moon BC. (2019). Sequencing and comparative analysis of the chloroplast genome of Angelica polymorpha and the development of a novel Indel marker for species identification. Molecules. 24:1038. https://www.mdpi.com/1420-3049/24/6/1038, (cited by 2020 Sep 20).
[https://doi.org/10.3390/molecules24061038]

-
Park SI, Hwang BK, Gil JS, Chung H, Kim HB, Kim OT, Kim SC, Koo SC, Um Y and Lee Y. (2017). Determination of the origin of Angelica roots using Angelica gigas chloroplast based SSR markers. Korean Journal of Medicinal Crop Science. 25:361-366.
[https://doi.org/10.7783/KJMCS.2017.25.6.361]

-
Song Y, Chen Y, Lv J, Xu J, Zhu S, Li M and Chen N. (2017). Development of chloroplast genomic resources for Oryza species discrimination. Frontiers in Plant Science. 8:1854. https://www.frontiersin.org/articles/10.3389/fpls.2017.01854/full?report=reader, (cited by 2020 Sep 19).
[https://doi.org/10.3389/fpls.2017.01854]

-
Steele KA, Ogden R, McEwing R, Briggs H and Gorham J. (2008). InDel markers distinguish Basmatis from other fragrant rice varieties. Field Crops Research. 105:81-87.
[https://doi.org/10.1016/j.fcr.2007.08.001]

-
Väli Ü, Brandström M, Johansson M and Ellegren H. (2008). Insertion-deletion polymorphisms(indels) as genetic markers in natural populations. BMC Genetics. 9:8. https://link.springer.com/article/10.1186/1471-2156-9-8, (cited by 2020 Sep 19).
[https://doi.org/10.1186/1471-2156-9-8]

-
Varshney RK, Close TJ, Singh NK, Hoisington DA and Cook DR. (2009a). Orphan legume crops enter the genomics era. Current Opinion in Plant Biology. 12:202-210.
[https://doi.org/10.1016/j.pbi.2008.12.004]

-
Varshney RK, Nayak SN, May GD and Jackson SA. (2009b). Next-generation sequencing technologies and their implications for crop genetics and breeding. Trends in Biotechnology. 27:522-530.
[https://doi.org/10.1016/j.tibtech.2009.05.006]

-
Wolfe KH, Ll WH and Sharp PM. (1987). Rates of nucleotide substitution vary greatly among plant mitochondrial, chloroplast, and nuclear DNAs. Proceedings of the National Academy of Sciences. 84:9054-9058.
[https://doi.org/10.1073/pnas.84.24.9054]

