
황기의 유전체 기반 SSR 마커 개발
 ; Yurry Um2
; Yurry Um2 ; Yi Lee3
; Yi Lee3 ; Yoon Jeong Lee4
; Yoon Jeong Lee4 ; Sung Cheol Koo5
; Sung Cheol Koo5 ; Woo Tae Park6
; Woo Tae Park6 ; Jang Hoon Kim7
; Jang Hoon Kim7 ; Yun Chan Huh8
; Yun Chan Huh8 ; Youn Ho Moon9
; Youn Ho Moon9
This is an open access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Although Astragalus membranaceus is a major medicinal herb used for food and medicinal purposes, genome-based research for its exploration is insufficient. The introduction of the genetic breeding system for medicinal crops, based on genomic analysis, will have a great impact not only on the establishment of the origin but also on understanding the existing traditional breeding system.
A. membranaceus from five accessions of Poongsung, Asung, Jecheon, Sancheong, and Jilin from China were used, which are cultivated in the test field. The produced nucleotide sequences were assembled de novo to obtain 450,449 contigs, and the repeat sequences were extracted using the selection criteria, two to six nucleotide sequences repeated five time or more. Finally, we obtained 147,766 simple sequence repeat (SSR) marker candidates. Using the CLC genomics workbench program, 949 SSR markers showing mutations for Astragalus in the 5 accessions were selected. From the designed primer sets, 99 were randomly selected and applied to the genomic DNA which was extracted from five cultivars and pooled. Ten primer sets showing more than two bands were finally selected as SSR markers, and were used for the genetic relationship analysis in 81 strains of A. membranaceus.
The data of this study can be used as a marker when registering A. membranaceus varieties through pure line selection and line breeding in the future. It is thought that it can greatly contribute to securing intellectual property rights for domestic Astragalus breeding varieties.
Keywords:
Astragalus membranaceus, Next Generation Sequencing, Simple Sequence Repeat서 언
한국에서 재배되는 황기는 단너삼이라고도 하며 콩과의 다년생 초본식물로 학명은 Astragalus membranaceus, 약명은 Astragali Radix로 사용한다. 황기는 한국과 몽골, 중국 동북부, 북서부, 서북부에서 오랫동안 전통 한약재로 이용되어 왔다 (Fu et al., 2014; Li et al., 2018).
우리나라에서는 황기를 주로 식품과 한약재로서 재배하고 있으며, 그 뿌리는 원뿌리가 곧게 뻗으며 외피는 황갈색이지만 잘라 보면 둘레는 유백색이고 속은 황백색을 띤다. 황기의 뿌리는 독성이 없어 안전하면서도 다양한 약리 효능을 가지고 있기 때문에 식용과 약용으로 모두 사용할 수 있다. 황기는 전통적으로 피로, 식욕감퇴, 자연발한, 호흡곤란 등의 증세를 치료하고 쇠약해진 기운을 회복시켜주는 약재로 많이 사용되어 왔으며, 이외에도 항염증 및 항고혈압, 간장보호, 항산화, 항바이러스, 심장혈관 보호, 면역증진 활성, 항노화 등의 효능이 보고된 바 있다 (Zhang et al., 1984; Kajimura et al., 1996; Wang and Feng, 2000; Dong et al., 2003; Kim et al., 2003, 2007; Lee et al., 2003; Lei et al., 2003; Du et al., 2012).
분자 마커는 유전적 다양성 특성화, 유전자 합성 경로에 관한 연구, QTL (quantitative-trait locus) mapping 및 진화 연구와 같은 다양한 영역에서 널리 사용된다 (Moose and Mumm, 2008). 특히 PCR을 활용한 SNP, SSR 등의 분자마커를 활용하면 적은 양의 DNA를 이용하여 식물의 생장과 상관없이 다양한 조직에서 안정적으로 마커 탐색이 가능하며 비교적 적은 비용으로 빠른 시간 안에 분석이 이루어진다는 장점을 가진다 (Jo et al., 2013; Um et al., 2016).
최근 NGS 기술의 발전은 대량의 마커 발굴을 가능하게 하였으며 (Mardis, 2008; Wang et al., 2009), 그 결과 옥수수, 밀, 벼 등 주요 식량작물이나 채소, 화훼에서는 육종 기술의 발달과 마커의 분자 표지 개발에 관한 연구가 활발히 이루어졌다 (Nybom, 2004; Henry, 2012; Kilian and Graner, 2012). 그러나, NGS 기법을 활용한 약용작물의 유전 다양성에 대한 연구는 더덕 (Kim et al., 2016), 당귀 (Gil et al., 2017), 도라지 (Kim et al., 2020)와 같은 주요 약용작물을 제외하면 분자 수준의 연구가 매우 미흡한 실정이다.
Li 등 (2017)은 황기의 전사체 분석 연구를 통해 황기의 유효성분인 calycosin과 calycosin-7-O-β-D-glucoside의 생합성 관련 유전자의 기능을 밝힌 바 있으나 이외의 전사체나 단백질체에 관한 연구 보고는 알려진 바 없다.
국내에서는 황기 재배지에서 토양의 물리적 특성에 따른 습해 정도와 재배 적지 기준 설정, 바실러스 속 미생물에 의한 황기의 생육 촉진 및 토양병해 방제효과 등과 같은 재배관리기술 연구, 황기 추출물의 항당뇨 및 지질개선 효능 등 성분물질 분석, 약리효능 평가에 대한 연구가 꾸준히 진행 중에 있으나 유전학, 계통학적 연구는 미흡한 실정이다 (Lee et al., 2021).
따라서 본 연구에서는 국내에서 널리 재배되며 이용하고 있는 황기를 대상으로 NGS 분석을 통한 SSR 마커를 개발하여 황기의 유전적 다양성을 분석하고 향후 분자육종에 필요한 연구 기반을 구축하고자 하였다.
재료 및 방법
1. 황기 시료
본 연구에서 high depth로 WGS (Whole genome sequencing)을 수행한 황기 재료는 국립원예특작과학원 인삼특작부 약용작물과에서 재배중인 황기의 품종 ‘풍성’을 사용하였다.
다형성 SSR 프라이머 선발을 위한 시료는 위와 마찬가지로 약용작물과 시험포지에서 재배중인 황기 품종으로 아성, 지역수집종인 제천, 산청 그리고 국외 수집종인 중국 길림을 사용하였으며 선발된 SSR 마커의 재현성 및 유전다양성 분석을 위한 재료로는 약용작물과에서 수집한 계통과 교배로 얻어진 81 계통의 잎 시료를 사용하였다.
2. SSR 탐색 및 프라이머 제작
NGS는 Illumina Hiseq 2500 플랫폼 (Illumuna, SanDiego, CA, USA)을 이용하여 수행하였으며, 이를 통해 얻은 핵산조각을 Gil 등 (2017)의 방법에 따라 SOAPdenovo2 version 2.04의 SOAPec을 이용하여 error를 보정한 후 SOAPdenovo2를 이용하여 조합하였다.
Microsatellites, transposable elements, rDNAs 등과 같은 반복서열은 RepeatMasker version 4.0.5과 RepeatModeler version 1.0.8을 이용하여 분석하였다. 또한 SSR finder를 사용하여 SSR 구간을 탐색하였다. SSR 구간을 탐색하기 위해 2 가지의 변수를 고려하였다. 첫 번째는 2 개에서 6 개의 염기쌍 (base pairs)이 직렬로 최소 5 회 이상 반복되는 염기서열을 우선 선택하였고, 두 번째는 반복되는 motif에 변이가 있는 것은 제외하였다. Primer3 program (Untergasser et al., 2012)을 이용하여 SSR 프라이머 서열을 다음과 같은 조건에서 디자인하였다.
길이는 18 bp에서 26 bp (최적 길이 23 bp)이고 GC 함량은 50% 이상을 기준으로 하였으며, Tm 값은 55℃에서 62℃ (최적 조건 58℃)로 설정하였다. PCR product의 길이는 150 bp에서 250 bp 사이가 되게 설계하되, 모든 반복 서열 영역을 제외한 나머지 부분에서 프라이머를 제작하였으며 프라이머쌍 사이에 서로 다른 motif의 반복 구간이 들어간 것들은 모두 제외하였다. 최종적으로 Blastn tool을 사용하여 프라이머의 신규성을 확인하였다.
2. 다형성 SSR 프라이머 선발
연구에서 선발된 5 개 황기 유전자원 (풍성, 아성, 제천, 산청, 길림)에서 각각 1 개체씩을 임의로 선별하고 genomic DNA를 추출한 후, 이를 동량으로 혼합하는 genomic DNA pooling 방법을 이용하여 혼합된 genomic DNA를 99 개의 프라이머에 적용하였다.
Fragment Analyzer Automated CE system (Advanced Analytical Technologies, Ankeny, IA, USA) 기기를 이용하여 Quant-iT PicoGreen dsDNA reagent kit, 1 - 500 bp (Invitrogen Co., Carlsbad, CA, USA)로 전기영동을 수행하였다.
증폭된 밴드는 PROSize version 2.0 software (Advanced Analytical Technologies Inc., Ankeny, IA, USA) 프로그램을 이용하여 분석하였다. 이를 바탕으로, 결과값이 뚜렷하고, PCR product의 증폭 길이가 예상되었던 수치와 비슷하며, 또한 2개 이상의 밴드를 보이는 프라이머를 선별하였다.
3. SSR 마커의 재현성 및 유전다양성 검정
선발된 마커를 수집된 자원에 적용하여 개발된 마커의 효율성을 검정하였다. 이를 위해서 선발된 프라이머를 총 81 개체에 각각 적용하였고, 증폭된 PCR 산물을 DNA Fragment Analyzer Automated CE System (Advanced Analytical Technologies Inc., Ankeny, IA, USA)을 이용하여 분석하였다. 그 결과를 마커 분석 프로그램인 Power Marker software (Version 3.23) (Liu and Muse, 2005) 를 이용하여 선발된 SSR 마커의 효율성을 분석하였다.
결과 및 고찰
1. 황기 염기서열 분석 및 SSR 구간 탐색과 프라이머 제작
본 연구에서는 Illumina Hiseq 2500 platform을 이용하여 황기의 염기서열 분석을 수행하였다. Paired-end sequencing 방법을 이용하여 총 844,014,134 개의 read를 포함하는 총 42.06 Gbp의 염기서열 정보를 생산하였고, GC의 비율은 38.48%로 나타났다. 국내에서 황기 유전체 연구를 수행한 바 없었기 때문에 de novo 어셈블리를 수행하였고 k-mer 주파수 스펙트럼의 분석은 판독의 17 k-mer 깊이 분포에서 피크 (k-mer 깊이: 12/22)를 나타내었으며, 유전체 크기는 1,535,632,662 bp로 나타났다. 이는 게놈 전체에 걸쳐 높은 이형접합성을 시사한다.
de novo 어셈블리 결과 생성된 contig 및 scaffolds의 수는 총 450,449 개 였고, N50은 2,861 bp, N90은 783 bp로 나타났다 (Table 1). 생성된 contig 및 scaffold 중 가장 긴 contig 및 scaffold의 길이는 46,758 bp 였다 (Table 2).
RepBase database를 사용하여 반복 서열을 찾아내는 방법결과, 황기 유전체의 12.27%에서 반복 서열을 확인할 수 있었고 자가 유전체 상동성 검색을 통한 종 특이적 반복 서열을 찾아내는 방법으로 분석한 결과 42.05%의 반복 서열을 확인 할 수 있었다. 이처럼 reference based와 de novo based 방법으로 찾아낸 반복 서열의 병합 결과는 43.67%로 분석되었다 (Table 3).
황기 유전체에서 분석된 총 SSR의 양상을 분석한 결과 염기서열 길이에 상관없이 2 번 반복되는 것은 77.02%, 3 번 반복되는 것은 19.04%, 4 번 반복되는 것은 2.67%로 같은 서열이 2 번 반복되는 양상을 보이는 SSR이 가장 큰 비율을 차지하고 있었다 (Table 4).
또한 SSR motif 분석 결과 염기서열 2 개 (2 bp repeat)가 반복되는 di-nucleotide 비율이 8.88%, tri-nucleotide 9.62%, tetra-nucleotide 10.29%, penta-nucleotide 7.81%, hexa-nucleotide 11.65% 로 6 개 염기서열이 반복되는 SSR의 수가 가장 큰 비율을 차지하는 것을 알 수 있었다 (Table 5).
반복되는 염기서열의 유형을 분석하였을 때는 AG (AG/GA/TC/CT)가 48.5%로 가장 많은 비율을 차지하였고 두 번째 많은 비율을 차지하는 유형은 AT (AT/TA)로서 35.62%에 해당 하였다 (Table 6). 탐색된 황기 SSR 중에서 중복되는 SSR motif가 없도록 10,101 개의 SSR 프라이머를 설계하였다.
2. SSR 프라이머 선발
풍성 황기를 제외한 4 종 (아성, 제천, 산청, 길림)의 gDNA를 추출하여 각 5 Gbp 염기서열을 생산하였다. 생산된 각 계통의 염기서열을 CLC genomic walkbench 를 이용하여 어셈블리하였고 in silico 상에서 앞서 설계된 10,101 개 프라이머를 이용하여 다양성 분석을 수행하였고 그 결과 949 개의 polymorphic한 프라이머를 선발할 수 있었다.
949 개의 SSR중에서 반복 서열이 3 개인 tri-nucleotide 이상이면서 반복 서열 주변에 Indel이 없는 SSR을 선발한 결과 99 개의 마커가 분류되었다. 이들 마커의 motif 종류는 tri motif 56 개, tetra motif 32 개, penta motif 4 개, hexa motif 7 개였으며, 결과적으로 motif 별로 고르게 마커가 개발 되었음을 알 수 있었다.
3. 유전체 기반 SSR 마커의 효율성 검정
해당 마커를 이용하여 품종 육성 중인 황기 계통 81 개 DNA에 대해 유전자형 검정한 결과 10 종의 황기 SSR에서 높은 재현성을 확인할 수 있었다 (Table 7).

Characterization of 10 polymorphic single sequence repeat (SSR) markers validated in 81 A. membranaceus accessions.
개발된 마커의 효율성을 검증하기 위하여 81 개체에 적용하여 분석한 결과, frequency of major alleles (MAF)은 0.23 - 0.61 (평균 0.45)이었고, genotype number (GN)의 값은 2.0 - 15.0 (평균 8.9)였으며, heterozygosity (HE)는 0.50 - 0.87 (평균 0.70)이었다. Observed heterozygosity (HO)의 값은 0.02 - 0.74 (평균 0.50)였으며, polymorphism information content (PIC)의 값은 0.38 - 0.85 (평균 0.66)이었다. PIC 값이 가장 컸던 마커는 Am-gSSR14였으며, 이 마커는 PIC값과 HE 뿐만 아니라 GN도 높게 나타났다 (Table 8).
10 개의 SSR 마커를 이용하여 얻은 결과를 바탕으로 육성 중인 황기 유전자원 81 개에서 유전자형을 분석하여 계통도를 작성한 결과 자원별로 그룹이 이루어지지 않고 분리되는 것을 확인하였다 (Fig. 1). 따라서 개발된 황기 SSR 마커는 유전다 양성을 확인하는데 우수한 마커임을 알 수 있었다.
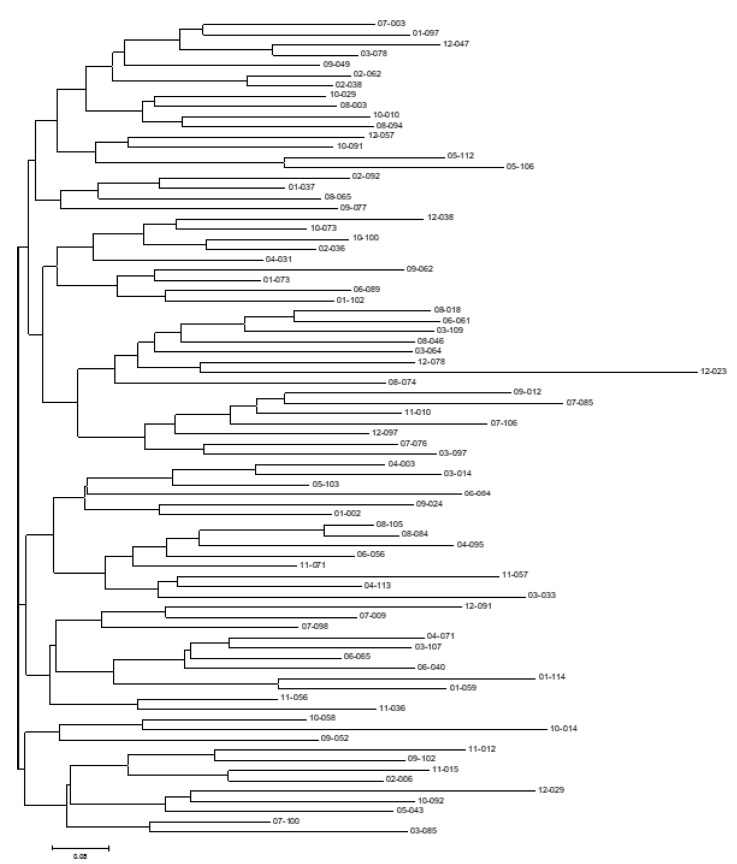
Dendrogram generated using neighbor joining cluster analysis based on genetic diversity of 81 A. membranaceus accessions.
황기 (A. membranaceus)의 유전학적 연구는 전무한 실정이었으나 최근 중국에서 엽록체 DNA의 유전체 전체 게놈이 완성 및 분석된 바 있다 (Lei et al., 2016; Wang et al., 2016). 또 Tian 등 (2021)에 의해 Astragalus 속 3 종의 엽록체 DNA가 완성되면서 엽록체 DNA상의 SSR을 분석한 것이 보고되었으나 우리나라에서 재배되는 황기 (A. membranaceus)에 대한 유전자 마커는 개발되어 있지 않다.
본 연구에서 개발된 황기의 SSR 마커는 차세대염기서열정보 분석 기법을 활용한 유전체 정보를 기반으로 한 것으로써, 황기의 유전정보 확보 및 염기서열 수준에서의 유전적 다양성 분석에 의의가 있다. 또한 유전체 분석을 통해 특정 DNA 영역에 국한되지 않고 다양한 유전좌위에 적용할 수 있다는 장점을 가지고 있다. 또한, 이를 이용하여 국내·외 황기유전자원의 다양성을 분석함으로써, 황기 자원의 정확하고 체계적인 계통 분류 및 집단 선발 등과 같은 분자 육종에 필요한 기본적 자료를 제공할 수 있다. 본 연구에서 개발된 SSR마커는 향후 국내외 황기 유전자원의 확보 및 다양성 분석, 우수 형질의 품종 개발에도 도움을 줄 것으로 사료 된다.
Acknowledgments
본 연구는 농촌진흥청 연구사업(과제번호: PJ014402022021)의 지원에 의해 이루어진 결과로 이에 감사드립니다.
References
-
Dong TTX, Ma XQ, Clarke C, Song ZH, Ji ZN, Lo CK and Tsim KWK. (2003). Phylogeny of Astragalus in China: Molecular evidence from the DNA sequences of 5S rRNA spacer, ITS, and 18S rRNA. Journal of Agricultural and Food Chemistry. 51:6709-6714.
[https://doi.org/10.1021/jf034278x]

-
Du X, Zhao B, Li J, Cao X, Diao MK, Feng H, Chen X, Chen Z and Zeng X. (2012). Astragalus polysaccharides enhance immune responses of HBV DNA vaccination via promoting the dendritic cell maturation and suppressing Treg frequency in mice. International Immunopharmacology. 14:463-470.
[https://doi.org/10.1016/j.intimp.2012.09.006]

-
Fu J, Wang Z, Huang L, Zheng S, Wang D, Chen S, Zhang H and Yang S. (2014). Review of the botanical characteristics, phytochemistry, and pharmacology of Astragalus membranaceus (Huangqi). Phytotherapy Research. 28:1275-1283.
[https://doi.org/10.1002/ptr.5188]

-
Gil JS, Um YR, Kim SR, Kim OT, Koo SC, Reddy CS, Kim SC, Hong CP, Park SG, Kim HB, Lee DH, Jeong BH, Chung JW and Lee Y. (2017). Development of genome-wide SSR markers from Angelica gigas Nakai using next generation sequencing. Genes. 8:238. https://www.mdpi.com/2073-4425/8/10/238, (cited by 2021 Oct 20).
[https://doi.org/10.3390/genes8100238]

-
Henry RJ. (2012). Next-generation sequencing for understanding and accelerating crop domestication. Briefings in Functional Genomics. 11:51-56.
[https://doi.org/10.1093/bfgp/elr032]

-
Jo IH, Bang KH, Kim YC, Kim JU, Shin MR, Moon JY, Noh BS, Hyun DY, Kim DH, Cha SW and Kim HS. (2013). Analysis of mitochondrial DNA sequence and molecular marker development for identification of Panax species. Korean Journal of Medicinal Crop Science. 21:91-96.
[https://doi.org/10.7783/KJMCS.2013.21.2.91]

-
Kajimura K, Takagi Y, Ueba N, Yamasaki K, Sakagami Y, Yokoyama H and Yoneda K. (1996). Protective effect of Astragali radix by intraperitoneal injection against Japanese encephalitis virus infection in mice. Biological and Pharmaceutical Bulletin. 19:855-859.
[https://doi.org/10.1248/bpb.19.855]

-
Kilian B and Graner A. (2012). NGS technologies for analyzing germplasm diversity in genebanks. Briefings in Functional Genomics. 11:38-50.
[https://doi.org/10.1093/bfgp/elr046]

-
Kim JE, Kang SH, Park SG, Yang TJ, Lee Y, Kim OT, Chung OS, Lee JH, Choi JP, Kwon SJ, Lee KP, Ahn BO, Lee DJ, Yoo SI, Shin IG, Um Y, Lee DY, Kim GS, Hong CP, Bhak J and Kim CK. (2020). Whole-genome, transcriptome, and methylome analyses provide insights into the evolution of platycoside biosynthesis in Platycodon grandiflorus, a medicinal plant. Horticulture Research. 7:112. https://www.nature.com/articles/s41438-020-0329-x, (cited by 2021 Oct 20).
[https://doi.org/10.1038/s41438-020-0329-x]

-
Kim JH, Park SY, Lim HK, Park AY, Kim JS, Kang SS, Youm JR and Han SB. (2007). Quantitative evaluation of radix Astragali through the simultaneous determination of bioactive isoflavonoids and saponins by HPLC/UV and LC-ESI-MS/MS. Bulletin of the Korean Chemical Society. 28:1187-1194.
[https://doi.org/10.5012/bkcs.2007.28.7.1187]

-
Kim SR, Jeong JH, Chung H, Kim JH, Gil JS, Yoo JM, Um Y, Kim OT, Kim TD, Kim YY, Lee DH, Kim HB and Lee Y. (2016). Simple sequence repeat marker development from Codonopsis lanceolata and genetic relation analysis. Journal of Plant Biotechnology. 43:181-188.
[https://doi.org/10.5010/JPB.2016.43.2.181]

-
Lee EJ, Choi H, Yoon WC, Kim YS, Song BN, Lee MY, Park BR, Lee SH, Choi JH and Park SY. (2021). Anti-diabetic and lipid profile effect of Astragalus membranaceus(Fisch.) Bunge fermented by Aspergillus awamori in db/db mice. Korean Journal of Medicinal Crop Science. 29:263-272.
[https://doi.org/10.7783/KJMCS.2021.29.4.263]

-
Lee ES, Hyeon GS, Kim YI, Chang JK and An TJ. (2021). Establishment of Soil Suitability and Assessment of Wet Injury in Astragalus membranaceus(Fisch.) Bunge by Soil Properties. Korean Journal of Medicinal Crop Science. 29:273-281.
[https://doi.org/10.7783/KJMCS.2021.29.4.273]

-
Lee GW, Yi JH, Mo YM, Hong JE, Jo TH and Lim SJ. (2021). Effect of Bacillus spp. on growth promotion and control of soil borne diseases of Astragalus membranaceus Bunge. Korean Journal of Medicinal Crop Science. 29:99-109.
[https://doi.org/10.7783/KJMCS.2021.29.2.99]

-
Lee YS, Han OK, Park CW, Suh SI, Shin SW, Yang CH, Jeon TW, Lee ES, Kim KJ, Kim SH, Yoo WK and Kim HJ. (2003). Immunomodulatory effects of aqueous extracted Astragali radix in methotrexate treated mouse spleen cells. Journal of Ethnopharmacology. 84:193-198.
[https://doi.org/10.1016/S0378-8741(02)00298-2]

- Lei H, Wang B, Li WP, Yang Y, Zhou AW and Chen MZ. (2003). Anti-aging effect of astragalosides and its mechanism of action. Acta Pharmacologica Sinica. 24:230-234.
-
Lei W, Ni D, Wang Y, Shao J, Wang X, Yang D, Wang J, Chen H and Liu C . (2016). Intraspecific and heteroplasmic variations, gene losses and inversions in the chloroplast genome of Astragalus membranaceus. Scientific Reports. 6:21669. https://www.nature.com/articles/srep21669, (cited by 2021 Oct 20).
[https://doi.org/10.1038/srep21669]

-
Li J, Lee YH, Denton MD, Feng Q , Rathjen JR, Qu Z and Adelson DL. (2017). Long read reference genome-free reconstruction of a full-length transcriptome from Astragalus membranaceus reveals transcript variants involved in bioactive compound biosynthesis. Cell Discovery. 3:17031. https://www.nature.com/articles/celldisc201731, (cited by 2021 Oct 20).
[https://doi.org/10.1038/celldisc.2017.31]

-
Li R, Yin M, Yang M, Chu S, Han X, Wang M and Peng H. (2018). Developmental anatomy of anomalous structure and classification of commercial specifications and grades of the Astragalus membranaceus var. mongholicus. Microscopy Research and Technique. 81:1165-1172.
[https://doi.org/10.1002/jemt.23111]

-
Liu K and Muse S. (2005). PowerMarker: An integrated analysis environment for genetic marker analysis. Bioinformatics. 21:2128-2129.
[https://doi.org/10.1093/bioinformatics/bti282]

-
Mardis ER. (2008). Next-generation DNA sequencing methods. Annual Review of Genomics and Human Genetics. 9:387-402.
[https://doi.org/10.1146/annurev.genom.9.081307.164359]

-
Moose SP and Mumm RH. (2008). Molecular plant breeding as the foundation for 21st century crop improvement. Plant Physiology. 147:969-977.
[https://doi.org/10.1104/pp.108.118232]

-
Nybom H. (2004). Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants. Molecular Ecology. 13:1143-1155.
[https://doi.org/10.1111/j.1365-294X.2004.02141.x]

-
Tian C, Li X, Wu Z, Li Z, Hou X and Li FY. (2021). Characterization and comparative analysis of complete chloroplast genomes of three species from the genus Astragalus(Leguminosae). Frontiers in Genetics. 12:705482. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8378255/, (cited by 2021 Oct 20).
[https://doi.org/10.3389/fgene.2021.705482]

-
Um Y, J in ML, Kim OT, Kim YC, Kim SC, Ch a SW, Chung KW, Kim S, Chung CM and Lee Y. (2016). Identification of Korean ginseng(Panax ginseng) cultivars using simple sequence repeat markers. Plant Breeding and Biotechnology. 4:71-78.
[https://doi.org/10.9787/PBB.2016.4.1.71]

-
Untergasser A, Cutcutache I, Koressaar T, Ye J, Faircloth BC, Remm M and Rozen SG. (2012). Primer3-new capabilities and interfaces. Nucleic Acids Research. 40:e115. https://academic.oup.com/nar/article/40/15/e115/1223759?login=true, (cited by 2017 Oct 20).
[https://doi.org/10.1093/nar/gks596]

-
Wang B, Chen H, Ma H, Zhang H, Lei W, Wu W, Shao J, Jiang M, Zhang H, Jia Z and Liu C. (2016). Complete plastid genome of Astragalus membranaceus(Fisch.) Bunge var. membranaceus. Mitochondrial DNA Part B. 1:517-519.
[https://doi.org/10.1080/23802359.2016.1197057]

- Wang XJ and Feng P. (2000). Antioxidant activity of Qizhu tang. Acta Pharmacologica Sinica. 21:1141-1144.
-
Wang Z, Gerstein M and Snyder M. (2009). RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10:57-63.
[https://doi.org/10.1038/nrg2484]

-
Zhang WJ, Hufnagl P, Binder BR and Wojta J. (2003). Antiinflammatory activity of astragaloside IV is mediated by inhibition of NF-κB activation and adhesion molecule expression. Thrombosis and Haemostasis. 90:904-914.
[https://doi.org/10.1160/TH03-03-0136]

- Zhang YD, Wang YL, Shen JP and Li DX. (1984). Effect of astragaloside IV on inflammatory and hypertension. Acta Pharmacologica Sinica. 19:333-337.