rDNA FISH를 이용한 큰조롱과 넓은잎큰조롱의 세포유전학적 연구
This is an open access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Dried tuberous roots of Cynanchum wilfordii are known to relieve menopause symptoms. However, the dried roots of C. wilfordii are morphologically similar to those of C. auriculatum, which makes it difficult to distinguish when used as a medicine. Various comparative studies have focused on chemical or molecular analysis of these roots. However, the differences between the two species at the cytogenetic level based on chromosome structure and composition remain to be elucidated.
For chromosome slides, the roots were fixed in 8-hydroxyquinoline, digested with enzyme mixture, and spread on slides. 5S and 45S rDNA were used as cytogenetic markers for the analysis of nuclear genomes by FISH. The chromosome number of the two species was 2n = 22, with a relatively short length, 1.13 ㎛ - 4.24 ㎛ and 1.00 ㎛ - 3.42 ㎛ with respect to each other. Both species represent one pair of 5S and 45S rDNA signal on chromosome 1, at the proximal region and peri-centromeric region, respectively.
These preliminary cytogenetic data using FISH in C. wilfordii and C. auriculatum could be valuable for the comprehension of Cynanchum genome history.
Keywords:
Cynanchum wilfordii, Cynanchum auriculatum, Chromosome, Fluorescence in situ hybridization서 언
큰조롱 [Cynanchum wilfordii (Maxim.) Fook. f.]과 넓은잎큰조롱 (Cynanchum auriculatum Royle ex Wight)은 협죽도과 (Apocynaceae) 박주가리아과 (Asclepiadoideae)에 속하는 다년생식물로 한국과 중국에서 약용작물로 활용되어왔다 (Chase et al., 2016).
동북아시아 지역의 양지바른 산이나 들에 분포하며, 건조된 덩이뿌리가 다양한 질병을 치료하는 데 사용되어 왔다. 큰조롱의 덩이뿌리를 백수오라 하며, 백모 (白毛), 강장 (强壯), 보혈 (補血), 신경쇠약 (神經衰弱), 동맥경화 (動脈硬化) 등에 효능이 보고되어있다 (Hwang et al., 1999; Kim et al., 2003; Shan et al., 2006). 넓은잎큰조롱의 덩이뿌리는 우피소 (牛皮消) 또는 이엽우피소라 하여 항암 (抗癌), 보간 (保肝), 고지혈증 (高脂血症) 등을 치료하는 데 사용되어왔다 (Ham et al., 2007; Peng et al., 2008). 그러나 한국소비자원 (Korea Consumer Agency)과 미국 식약처 (Food and Drug Administration)는 이엽우피소의 추출물로부터 유산의 위험성이 높아진다는 연구보고서를 바탕으로 독성 식물 리스트에 등재하고 사용을 금지하고 있다 (Han and Luan, 1984; Lu et al., 1998).
최근, 국내에서는 약용작물로부터 추출된 건강기능식품의 수요가 급격히 증가하고 있다. 특히, 백수오는 갱년기 증상을 완화하는 효과가 밝혀지며 주목받고 있다 (Chang et al., 2012). 그러나, 백수오와 형태적으로 유사한 이엽우피소는 높은 생산성을 바탕으로 정확한 감별법이 부재한 시장에 혼용되고 있다 (Jiang et al., 2011). 또한, 백수오는 주로 추출물이나 분말 형태로 가공되어 판매되기 때문에 정확한 분별 기준의 부재는 소비자의 혼란을 초래하고 있다 (Song et al., 2004; Kim et al., 2015). 따라서 다방면의 연구를 통한 기초 정보의 축적은 필수적이라 할 수 있다.
큰조롱과 넓은잎큰조롱을 구분하기 위한 약리적 (Lee et al., 2013; Lee and Lee, 2015), 분자유전학적 (Kim et al., 2013; Kim et al., 2018), 형태적 (Kim et al., 2014; Doh et al., 2015; Nam and Chung, 2015) 분야에서는 이미 연구가 진행되었으나, 게놈의 특성을 밝히는데 유용한 세포유전학적 접근을 통한 연구는 염색체 수가 2n = 22라는 정보만이 밝혀져 있으며 (Albers and Meve, 2001; Yamashiro et al., 2002) 세부 연구정보는 부재한 실정이다.
FISH (fluorescence in situ hybridization)를 적용한 세포유전학적 접근은, 형광물질의 탐침을 통한 마커의 위치를 가시화하여 게놈 구조, 계통학적 관계 및 배수성 연구 등에서 활용되고 있다 (Tessadori et al., 2004; Devi et al., 2005; Ohmido et al., 2010). 특히, 종간 유연관계의 비교 분석에 활용되어 분류학적 관계를 규명하고 유전자의 이동 과정을 추적함으로써 게놈의 이해도를 높이는 데 도움을 주고 있다 (Jiang and Gill 2006; Chester et al., 2010). 주로 식물에 보편적으로 존재하며 높은 반복 빈도를 보이고, 서로 다른 유전자좌에 존재하는 5S와 45S rDNA를 마커로 활용한다 (Kato et al., 2004; Pellerin et al., 2019). 따라서 본 연구는 두 종의 세포유전학적 데이터를 확인하여 기초 데이터를 확보하고, 유전체 연구를 위한 기반을 마련하여 백미꽃속 게놈에 대한 이해도를 높이고자 하였다.
재료 및 방법
1. 식물재료
큰조롱 [Cynanchum wilfordii (Maxim.) Fook. f.]과 넓은잎큰조롱 (이엽우피소, Cynanchum auriculatum Royle ex Wight)의 종자는 농촌진흥청 원예특작과학원 인삼특작부 (충북 음성)에서 분양받았으며, 분양받은 종자를 24 시간 침종 후, wagner pot에 파종하여 발근을 유도하여 실험에 사용하였다.
증거표본 확보를 위하여 각각의 식물체를 석엽표본으로 만들어 식물표본관에 수납하였다 (Voucher No: KWNU No. 2019035 & KWNU No. 2019036).
2. 염색체 슬라이드 준비
염색체 슬라이드 준비는 Kato 등 (2004)의 방법을 참고하였다. 근단은 2 mM 8-hydroxyquinoline에 침지하여 12℃에서 4 시간 처리하였고, Carnoy`s solution (absolute ethanol : glacial acetic acid, 3 : 1, v/v)에 12 시간 처리하여 70% ethanol에 보관하며 실험 재료로 사용하였다.
고정한 근단의 분열조직을 절단하여 효소 혼합액 (4% cellulase onzuka R-10, 1% pectolylase Y-23, 100 mM citric buffer)에 침지시켜 37℃에서 60 분 처리하였다. 이후 분열조직을 Carnoy`s solution으로 수세하여 끝이 뭉뚝한 바늘로 분쇄하고 원심분리하여 세포를 가라앉혔다. 마지막으로 90% aceto-ethanol (9 : 1, v/v)로 다시 혼합한 후, 슬라이드에 퍼트리고 상온에서 건조시켰다. 광학현미경 하에서 분열상이 양호한 염색체를 선발하였고 이를 FISH에 이용하였다.
3. FISH
Probe 제작과 FISH는 Waminal과 Kim (2012)에 기술된 방법을 참고하여 수행하였다. 5S와 45S rDNA 서열은 Son 등 (2010, 2012)에서 기술한 서열을 활용하였다. 5S와 45S rDNA는 각각 digoxigenin-11-dUTP (Apexbio Tech, Houston, TX, USA)과 biotin-16-dUTP (Roche, Basel, Switzerland)로 label 되었다. 혼성화 혼합액 (50% formamide, 10% dextran sulfate, 2 × salin sodium citrate buffer, probes)은 90℃에서 5 분간 변성시킨 후 급냉시켰다. 슬라이드에 혼성화 혼합액을 적하하여, 37℃ humid chamber (Sanyo Electric Co., Ltd., Tokyo, Japan)에서 18 시간 이상 처리하였다.
슬라이드는 42℃의 20% formamide와 detection buffer에서 5 분씩 수세한 후, 1% Cy3-streptavidin (BioLegen, SanDiego, CA, USA)와 anti-digoxigenin-FITC (Sigma-Aldrich Co., St. Louis, MO, USA) 혼합액을 슬라이드에 첨가하여 37℃에서 1 시간 이상 반응시켰다. 슬라이드는 detection buffer에서 5 분씩 3 번 그리고 70%, 90%, 100% 에탄올에서 각각 3 분씩 수세하였다. Vectashield를 포함한 4,6-diamidino-2-phenylindole (DAPI, Vector Laboratories Inc., Burlingame, CA, USA)를 슬라이드에 도포하여 커버 슬립을 덮은 후, 공초점 현미경 (LSM 880, Carl Zeiss, Oberkochen, Germany) 으로 signal을 확인하고 사진을 촬영하였다. Adobe Photoshop CS6를 이용하여 이미지를 편집하였고, 염색체의 길이는 Image J (Wayne rasband, Bethesda, MD, USA)를 사용하여 분석하였다.
결과 및 고찰
핵형분석은 종간 유연관계뿐만 아니라 종의 기원 및 다양성을 판단하기에 유용한 방법으로 활용된다 (Bhat and Wani, 2017). 관련된 정보는 염색체 수, 크기, 형태, satellite의 위치, 그리고 banding 패턴 등이 있다 (Guerra, 2008).
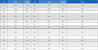
본 연구에서는 DAPI 염색을 통해 중기 상태의 염색체를 선발하여 이전 연구보다 정확한 염색체의 수, 크기 등 특징들을 확인하였고, Fig. 1에 나타내었다. 두 종 모두 5 개 이상의 세포를 분석하였으며, 길이에 따라 각 염색체에 번호를 붙이고 길이를 측정하여 Table 1에 나타내었다.

DAPI staining on somatic metaphase chromosomes.(A) C. wilfordii (2n = 22) and (B) C. auriculatum (2n = 22). Scale bars = 2 ㎛.
큰조롱 [Cynanchum wilfordii (Maxim.) Fook. f.]과 넓은잎큰조롱 (Cynanchum auriculatum Royle ex Wight)을 포함한 백미꽃속 (Cynanchum)에 포함된 대부분의 식물 염색체 수는 2n = 22로 밝혀져 있다 (Albers and Meve, 2001; Yamashiro et al., 2002).
본 연구에서도 두 종 모두 2n = 2x = 22로, 기본 염색체 수는 x = 11인 이배체 식물임을 재확인하였다. 두 종의 경우, 핵형 수준에서는 염색체의 길이로 차이를 확인할 수 있었다. 큰조롱은 염색체 길이의 범위가 1.31 ㎛ - 4.24 ㎛로 확인되었다. 가장 긴 1 번 염색체는 평균 길이가 3.78 ± 0.39 ㎛, 가장 짧은 11 번 염색체의 평균 길이는 1.36 ± 0.08 ㎛로 확인되었다. 넓은잎큰조롱의 염색체 길이 범위는 1.00 ㎛ - 3.42 ㎛로 확인되었다. 가장 긴 1 번 염색체는 평균 길이는 2.81 ± 0.49 ㎛, 가장 짧은 11 번 염색체의 평균 길이는 1.27 ± 0.21 ㎛로 확인되었다. 11 쌍의 염색체 모두 큰조롱의 염색체가 넓은잎큰조롱보다 평균적으로 0.2 ㎛ 정도 더 길이가 긴것을 확인하였다.
핵형은 동원체를 기준으로 단완과 장완의 비율로 분석하지만, 이번 연구에서는 일반적인 염색 방법이나 DAPI 염색으로 정확한 중기 염색체상을 얻지 못하였다. 그 외 satellite 염색체 위치 또는 banding 패턴 등에서는 차이를 확인할 수 없었다.
두 종에 대한 정확한 세포유전학적 분석을 위해 5S와 45S rDNA 탐침을 이용하여 FISH를 수행하였고, 그 결과를 Fig. 2에 나타내었다. 큰조롱과 넓은잎큰조롱의 5S와 45S rDNA locus는 모두 1 번 염색체에서 1 쌍씩 확인되었다 (Fig. 3). 두 종 모두 5S rDNA는 단완의 말단에 위치하고, 바로 밑 부분인 일차 협착 구간에서 45S rDNA를 확인하였다.

Fluorescence in situ hybridization patterns on mitotic metaphase chromosomes.(A) C. wilfordii (2n = 22) and (B) C. auriculatum (2n = 22). Red arrows indicate 45S rDNA locus (red signals) and yellow arrows indicate 5S rDNA locus (green signals). Scale bars = 2 ㎛.

Karyotypic idiograms of C. wilfordii and C. auriculatum using FISH from the metaphase chromosome.(A) C. wilfordii and (B) C. auriculatum. Each species has only one 5S rDNA (green) and 45S rDNA (red) homologous pair. Scale bar = 2 ㎛.
5S와 45S rDNA는 리보솜 구성 성분으로 식물에 보편적으로 존재하며, 반복적으로 존재하는 특성이 있다. 5S rDNA는 rDNA 복제 수와 위치의 변이가 다양하여 식물 종간 유연관계와 진화를 추정하는 데 유용하게 활용되고 있다 (Maluszynska and Heslop-Harrison, 1993; Castilho and Heslop-Harrison, 1995). 또한, 45S rDNA도 모든 식물 종의 염색체상에서 1 쌍 이상이 존재하고 (Maluszynska and Heslop-Harrison, 1991), 위치나 개수의 변이가 매우 다양하여 같은 속 내에서도 5S rDNA와 동일한 위치에서 나타나지 않기 때문에 (Kim et al., 2006), 5S와 45S rDNA의 위치를 확인하여 유전자가 구별되는 진화 과정을 확인할 수 있는 마커로써 유용하게 활용되고 있다 (Martins and Galetti, 1999; Mantovani et al., 2005).
백미꽃속의 염색체에 관한 연구는 인도와 아프리카 자생종을 대상으로 수행되었는데 2n = 22 롤 보고된 것이 대부분이었다 (Meve and Liede, 1994; Liede, 1996). 아프리카 분포종인 C. virens의 염색체 수를 2n = 24로 보고하고 있는데, 이것은 1 번 염색체의 2 차 협착구간을 오 동정한 것으로 판단된다.
또한, Kannan과 Thoppil (2018)은 인도 자생 백미꽃속 두 종의 염색체를 관찰하고 1 번 염색체 상에 2 차 협착구간이 있음을 밝혔으며, 염색체의 크기와 염색체 수의 증가, 핵형 formula를 가지고 종의 진화 경향을 추정하면서 염색체의 크기가 작은 종이 좀 더 원시적인 종이라고 하였다. 한편, 큰 조롱과 넓은잎큰조롱의 식별을 위한 분자계통학적 연구에서 ITS sequence를 비교한 결과, 단지 5 bp만 차이가 나는 결과를 얻어 이것은 두 종을 구별하는 marker로서는 유용성이 떨어짐을 밝혔다 (Sato-Masumoto et al., 2017). 그러나 Kim 등 (2018)은 두 종을 구별할 수 있는 marker로서 3개의 InDel 구간을 밝혔다. 즉, 큰조롱 (C. wilfordii)은 trnQ 와 psbK gene 사이에 InDel을 가지고 있었고, 넓은잎큰조롱 (C. auriculatum)은 rps2-rpoC2 구간과 psaJ-rpl33 구간에서 InDels을 나타내었다.
이러한 결과는 큰조롱과 넓은잎큰조롱을 구별하는 유용한 marker라고 생각되며, 이 두 종을 구별하기 위해서는 꽃요소의 표현 형질과, 엽록체 gene의 InDel 구간을 확인하는 분자적 접근방법이 가장 효율적이라고 사료된다.
큰조롱과 넓은잎큰조롱의 FISH 결과에서는 유전자좌의 개수, 위치 및 배열이 모두 유사하게 나타났다. 이는 두 종이 유사한 진화 과정을 거쳤음을 추정할 수 있고, 염색체 재배열로 인한 유전자의 소실과 같은 변이가 나타나지 않았음을 확인할 수 있었다. 세포분열 단계에 따른 염색체 응축 정도의 차이 등을 고려해 보아도 5S와 45S rDNA을 이용한 분석으로는 두 종을 분류하기 위한 형질로 활용하기 어렵다고 판단되었다. 향후 genome sequencing을 통해 동원체에 특이적인 DNA 마커를 활용한 FISH를 적용하면 세포유전학적 수준에서 정확한 분석이 가능할 것이다.
재배 농민과 소비자가 큰조롱과 넓은잎큰조롱을 구분하는데 적용하기에는 어려운 방법일 수 있다. 현재까지 큰조롱과 넓은잎큰조롱을 분별할 수 있는 가장 빠르고 정확한 방법은 형태학적 수준에서 명확한 차이를 규명해 주는 것으로 판단된다. 특히 Kim 등 (2014)의 연구에서 밝힌 꽃의 형질에서 암술의 주두 (stigma)에 나타나는 돌기물, 부화관 (corona lobe)의 형태, 화분괴 구상체의 비율 등을 활용하는 것이 효율적일 것이라 생각된다.
본 연구에서 수행된 큰조롱과 넓은잎큰조롱의 핵형 및 rDNA를 이용한 FISH 분석은 게놈구조를 이해하고 유전자의 물리적 위치를 확인하는 기초 연구로써 두 종의 세포유전학적 기초자료로 유용하게 이용될 것이다. 특히 차세대 염기서열 분석 (next generation sequencing, NGS)을 통한 data를 바탕으로, 백미꽃속 내에서 높은 빈도로 존재하는 marker를 확보한다면 역동적인 게놈의 진화 과정에 대한 이해를 높여갈 수 있을 것으로 생각한다.
References
-
Albers F and Meve U. (2001). A karyological survey of Asclepiadoideae, Periplocoideae, and Secamonoideae, and evolutionary considerations within Apocynaceae s.l. Annals of the Missouri Botanical Garden. 88:624-656.
[https://doi.org/10.2307/3298637]

-
Bhat TA and Wani AA. (2017). Chromosome structure and aberrations. Fluorescence in situ hybridization(FISH) and its applications. Springer. New Delhi, India. p.343-367.
[https://doi.org/10.1007/978-81-322-3673-3]

-
Castilho A and Heslop-Harrison JS. (1995). Physical mapping of 5S and 18S–25S rDNA and repetitive DNA sequences in Aegilops umbellulata. Genome. 38:91-96.
[https://doi.org/10.1139/g95-011]

-
Chang A, Kwak BY, Yi KT and Kim JS. (2012). The effect of herbal extract(EstroG-100) on pre-, peri- and post-menopausal women: A randomized double-blind, placebo-controlled study. Phytotherapy Research. 26:510-516.
[https://doi.org/10.1002/ptr.3597]

-
Chase MW, Christenhusz MJM, Fay MF, Byng JW, Judd WS, Soltis DE, Mabberley DJ, Sennikov AN, Soltis PS, Stevens PF. (2016). An update of the angiosperm phylogeny group classification for the orders and families of flowering plants: APG IV. Botanical Journal of the Linnean Society. 181:1-20.
[https://doi.org/10.1111/boj.12385]

-
Chester M, Leitch AR, Soltis PS and Soltis DE. (2010). Review of the application of modern cytogenetic methods(FISH/GISH) to the study of reticulation(Polyploidy/Hybridisation). Genes. 1:166-192.
[https://doi.org/10.3390/genes1020166]

- Devi J, Ko JM and Seo BB. (2005). FISH and GISH: Modern cytogenetic techniques. Indian Journal of Biotechnology. 4:307-315.
-
Doh EJ, Kim JH, Choi GY, Lee SH, Song HJ, Ju YS and Lee GS. (2015). Microscopic identification-keys for Cynanchum wilfordii radix and Cynanchum auriculatum radix. Korean Journal of Herbology. 30:65-69.
[https://doi.org/10.6116/kjh.2015.30.4.65.]

-
Guerra M. (2008). Chromosome numbers in plant cytotaxonomy: Concepts and implications. Cytogenetic and Genome Research. 120:339-350.
[https://doi.org/10.1159/000121083]

- Ham IH, Lee JY, Yoon YJ, Yang GS, Tae J, Bu YM, Kim HC and Choi HY. (2007). Effects of Cynanchum spp. on the hyperlipidemia in rats induced by Triton WR-1339. Korean Journal of Herbology. 22:279-286.
- Han J and Luan DH. (1984). Sow abortion caused by feeding Cynanchum auriculatum. Animal Husbandry and Veterinary Medicine. 16:266-266.
-
Hwang BY, Kim SE, Kim YH, Kim HS, Hong YS, Ro JS, Lee KS and Lee JJ. (1999). Pregnane glycoside multidrug-resistance modulators from Cynanchum wilfordii. Journal of Natural Products. 62:640-643.
[https://doi.org/10.1021/np980479x]

-
Jiang J and Gill BS. (2006). Current status and the future of fluorescence in situ hybridization(FISH) in plant genome research. Genome. 49:1057-1068.
[https://doi.org/10.1139/g06-076]

-
Jiang Y, Choi HG, Li Y, Park YM, Lee JH, Kim DH, Lee JH, Son JK, Na MK and Lee SH. (2011). Chemical constituents of Cynanchum wilfordii and the chemotaxonomy of two species of the family Asclepiadacease, C. wilfordii and C. auriculatum. Archives of Pharmacal Research. 34:2021-2027.
[https://doi.org/10.1007/s12272-011-1203-z]

-
Kannan BN and Thoppil JE. (2018). Cytogenetic evaluation of two species of Cynanchum L.(Apocynaceae) from South India: A possible clue to evolution. Phytotaxa. 357:198-206.
[https://doi.org/10.11646/phytotaxa.357.3.4]

-
Kato A, Lamb JC and Birchler JA. (2004). Chromosome painting using repetitive DNA sequences as probes for somatic chromosome identification in maize. Proceedings of the National Academy of Sciences of the United States of America. 101:13554-13559.
[https://doi.org/10.1073/pnas.0403659101]

- Kim HK, Kim YA, Lee AY and Ko BS. (2003). Pattern analysis of Cynanchi wilfordii Radix and Polygoni multiflori Radix. Korean Journal of Pharmacognosy. 34:278-281.
-
Kim KH, Kim YS, Kim MR, Lee HY, Lee KH, Kim JH, Seong RS, Kang TS, Lee JH and Jang YM. (2015). Development of primer sets for the detection of Polygonum multiflorum, Cynanchum wilfordii and C. auriculatum. Journal of Food Hygiene and Safety. 30:289-294.
[https://doi.org/10.13103/JFHS.2015.30.3.289]

-
Kim MJ, Kim IJ, Choi SY, Han DH, Kim YH, Lim SC, Kim TJ, Nam SY, Song BH, Oh BU and Park CG. (2014). Comparison of Cynanchum wilfordii, C. auriculatum, Metaplexis japonica and Polygonum multiflorum by morphological characters. Korean Journal of Medicinal Crop Science. 22:113-120.
[https://doi.org/10.7783/KJMCS.2014.22.2.113]

- Kim MK, Wang H, Kim YJ, Sathiyamoorthy S, Kwon WS and Yang DC. (2013). Molecular authentication by multiplex-PCR of three similar medicinal plant species: Cynanchum wilfordii, Cynanchum auriculatum and Polygonum multiflorum(Fallopia multiflorum). Journal of Medicinal Plants Research. 7:2584-2589.
- Kim SY, Bang JW and Lee JK. (2006). Cytogenetic analysis using mitosis, meiosis chromosomes and bicolor fluorescence in situ hybridization of Bupleurum latissimum Nakai. Korean Journal of Medicinal Crop Science. 14:354-359.
-
Kim YG, Choi HJ, Shin JW, Jo AR, Lee KE, Cho SS, Hwang YP and Choi CY. (2018). Molecular discrimination of Cynanchum wilfordii and Cynanchum auriculatum by InDel markers of chloroplast DNA. Molecules. 23:1337. https://www.mdpi.com/1420-3049/23/6/1337, (cited by 2020 May 20).
[https://doi.org/10.3390/molecules23061337]

-
Lee BJ and Lee KJ. (2015). Discrimination and proper use of Polygoni multiflori Radix, Cynanchi wilfordii Radix, and Cynanchi auriculati Radix in Korea: A descriptive review. Evidence-based Complementary and Alternative Medicine. 2015: 827380. https://www.hindawi.com/journals/ecam/2015/827380/, (cited by 2020 May 20).
[https://doi.org/10.1155/2015/827380]

-
Lee HS, Choi JH, Kim YE, Kim IH, Kim BM and Lee CH. (2013). Effects of the Cynanchum wilfordii ethanol extract on the serum lipid profile in hypercholesterolemic rats. Preventive Nutrition and Food Science. 18:157-162.
[https://doi.org/10.3746/pnf.2013.18.3.157]

-
Liede S. (1996). A revision of Cynanchum(Asclepiadaceae) in Africa. Annals of the Missouri Botanical Garden. 83:283-345.
[https://doi.org/10.2307/2399864]

- Lu C, Zhang X, Zhai C, Wu L and Jiang Z. (1998). Toxicological study of food safety of Baishouwu(Cynanchum auriculatum Royle ex Wight). Journal of Nanjing Railway Medical College. 4:1998-2004.
-
Maluszynska J and Heslop-Harrison JS. (1991). Localization of tandemly repeated DNA sequences in Arabidopsis thaliana. The Plant Journal. 1:159-166.
[https://doi.org/10.1111/j.1365-313X.1991.00159.x]

-
Maluszynska J and Heslop-Harrison JS. (1993). Molecular cytogenetics of the genus Arabidopsis: In situ localization of rDNA sites, chromosome numbers and diversity in centromeric heterochromatin. Annals of Botany. 71:479-484.
[https://doi.org/10.1006/anbo.1993.1063]

-
Mantovani M, Abel LDDS and Moreira-Filho O. (2005). Conserved 5S and variable 45S rDNA chromosomal localisation revealed by FISH in Astyanax scabripinnis(Pisces, Characidae). Genetica. 123:211-216.
[https://doi.org/10.1007/s10709-004-2281-3]

- Martins C and Galetti PM. (1999). Chromosomal localization of 5S rDNA genes in Leporinus fish(Anostomidae, Characiformes). Chromosome Research. 7:363-367.
-
Meve U and Liede S. (1994). Cynanchum crassipedicellatum(Asclepiadaceae), a new and unusual succulent from Madagascar. Novon. 4:276-279.
[https://doi.org/10.2307/3391655]

-
Nam BM and Chung GY. (2015). A palynological study of the subfamily Asclepiadoideae(Apocynaceae s. l.) in Korea. Korean Journal of Plant Taxonomy. 45:183-191.
[https://doi.org/10.11110/kjpt.2015.45.2.183]

-
Ohmido N, Fukui K and Kinoshita T. (2010). Recent advances in rice genome and chromosome structure research by fluorescence in situ hybridization(FISH). Proceedings of the Japan Academy, Series B Physical and Biological Sciences. 86:103-116.
[https://doi.org/10.2183/pjab.86.103]

-
Pellerin RJ, Waminal NE and Kim HH. (2019). FISH mapping of rDNA and telomeric repeats in 10 Senna species. Horticulture, Environment and Biotechnology. 60:253-260.
[https://doi.org/10.1007/s13580-018-0115-y]

-
Peng Y, Li Y, Liu X, Zhang J and Duan J. (2008). Antitumor activity of C-21 steroidal glycosides from Cynanchum auriculatum Royle ex Wight. Phytomedicine. 15:1016-1020.
[https://doi.org/10.1016/j.phymed.2008.02.021]

-
Sato-Masumoto N, Uchikura T, Sugiwaki H, Yoshimura M, Masada S, Atsumi T, Watanabe M, Tanaka N, Uchiyama N, Amakura Y and Hakamatsuka T. (2017). Survey on the original plant species of crude drugs distributed as Cynanchi wilfordii Radix and its related crude drugs in the Korean and Chinese markets. Biological and Pharmaceutical Bulletin. 40:1693-1699.
[https://doi.org/10.1248/bpb.b17-00226]

-
Shan L, Liu R, Shen Y, Zhang W, Zhang C, Wu D, Min L, Su J and Xu X. (2006). Gastroprotective effect of a traditional chinese herbal drug “Baishouwu” on experimental gastric lesions in rats. Journal of Ethnopharmacology. 107:389-394.
[https://doi.org/10.1016/j.jep.2006.03.022]

-
Son JH, Park KC, Kim TW, Park YJ, Kang JH and Kim NS. (2010). Sequence diversification of 45S rRNA ITS, trnH-psbA spacer, and matK genic regions in several Allium species. Genes and Genomics 32:165-172.
[https://doi.org/10.1007/s13258-009-0849-0]

-
Son JH, Park KC, Lee SI, Jeon EJ, Kim HH and Kim NS. (2012). Sequence variation and comparison of the 5S rRNA sequences in Allium species and their chromosomal distribution in four Allium species. Journal of Plant Biology 55:15-25.
[https://doi.org/10.1007/s12374-011-9185-4]

- Song KS, Shin CG and Ju YS. (2004). External and internal morphological standard of original plants and herbal states in Polygoni multiflori and Cynanchi wilfordii radix. Korean Journal of Herbology. 19:55-66.
-
Tessadori F, van Driel R and Fransz P. (2004). Cytogenetics as a tool to study gene regulation. Trends in Plant Science. 9:147-153.
[https://doi.org/10.1016/j.tplants.2004.01.008]

-
Waminal NE and Kim HH. (2012). Dual-color FISH karyotype and rDNA distribution analyses on four Cucurbitaceae species. Horticulture, Environment and Biotechnology. 53:49-56.
[https://doi.org/10.1007/s13580-012-0105-4]

- Yamashiro T, Yokota M, Denda T, Tateishi Y, Yokoyama J and Maki M. (2002). Chromosome numbers of Japanese Asclepiadaceae. Acta Phytotaxonomica et Geobotanica. 53:143-152.