
강원도 영월 산에서 발견된 거대 백수오 추정 식물자원의 형태 및 유전적 특성
1서울대학교 농림생물자원과학부 식물유전체육종연구소 박사과정생2서울대학교 농림생물자원과학부 식물유전체육종연구소 석사과정생
3서울대학교 농림생물자원과학부 식물유전체육종연구소 연구원
4농촌진흥청 국립원예특작과학원 인삼과 연구원
5서울대학교 농림생물자원과학부 식물유전체육종연구소 석사과정생
6서울대학교 농림생물자원과학부 식물유전체육종연구소 박사과정생
7서울대학교 농림생물자원과학부 식물유전체육종연구소 박사과정생
8서울대학교 농림생물자원과학부 식물유전체육종연구소 연구원
9강원도 영월군 영월읍 하송로 93 약초채취자
10농촌진흥청 국립원예특작과학원 약용작물과 연구사
11농촌진흥청 국립원예특작과학원 약용작물과 연구관
12서울대학교 농림생물자원과학부 식물유전체육종연구소 교수
2Master’s degree student, Department of Agriculture, Forestry and Bioresources, Seoul National University, Seoul 08826, Korea
3Researcher, Department of Agriculture, Forestry and Bioresources, Seoul National University, Seoul 08826, Korea
4Researhcer, Department of Herbal Crop Research, NIHHS, RDA, Eumseong 27709, Korea
5Master’s degree student, Department of Agriculture, Forestry and Bioresources, Seoul National University, Seoul 08826, Korea
6Doctor’s degree student, Department of Agriculture, Forestry and Bioresources, Seoul National University, Seoul 08826, Korea
7Doctor’s degree student, Department of Agriculture, Forestry and Bioresources, Seoul National University, Seoul 08826, Korea
8Researcher, Department of Agriculture, Forestry and Bioresources, Seoul National University, Seoul 08826, Korea
9Herbalist, Yeongwol 26237, Korea
10Researcher, Department of Herbal Crop Research, NIHHS, RDA, Eumseong 27709, Korea
11Researcher, Department of Herbal Crop Research, NIHHS, RDA, Eumseong 27709, Korea
12Professor, Department of Agriculture, Forestry and Bioresources, Seoul National University, Seoul 08826, Korea
This is an open access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
A giant root that resembled the root of Cynanchum species (Giant Cynanchum) was found on a mountain in Gangwon Province, Korea, in March 2021. The root was 10 m long and weighted 20 kg. Its morphological characteristics highly resembled those of Cynanchum wilfordii (Cw) and Cynanchum auriculatum (Ca).
The giant root collected in Gangwon Province has been permanently exhibited at the National Institute of Horticultural and Herbal Science, Rural Development Administration, Eumsung, Korea. The plants were clonally propagated for further research at the Seoul National University farm. Seven molecular markers were applied to the root for accurate species authentication. However, the species could not be clearly identified because the results of seven markers yielded contradicting results : one Cw-type, two Ca-types, and four unidentified types. The internal transcribed spacer 2 (ITS2) of the nuclear ribosomal RNA coding gene and four plastid DNA barcoding regions (matK, rbcL, trnL-trnF, and psbA-psbH) were analyzed for sequence comparison and phylogenetic tree construction. The results showed that the species was close to Ca. However, the results obtained form the phylogenetic tree are not definite because Cynanchum species were not monophyletic
The species of the giant root is assumed to be close to Ca, but additional research is necessary to clarify the exact species and its origin. Moreover, this root might be a valuable plant resource for the bio-industry.
Keywords:
Cynanchum wilfordii, C. auriculatum, Germplasm, Giant Roots서 언
박주가리아과 (Asclepiadoideae)에 속하는 백미꽃속 (Cynanchum)은 전 세계적으로 약 200 여종이 보고되었으며 지중해, 유럽의 열대, 아시아의 아열대 및 온대 등 다양한 지역에 분포하고 있다 (Wu et al., 1991). 국내에는 백미꽃 (C. atratum), 산해박 (C. paniculatum), 양반풀 (C. thesioides), 큰조롱 (C. wilfordii) 등을 포함한 약 14 분류군의 백미꽃속 식물들이 서식하고 있는 것으로 알려져 있다 (Park et al., 2020).
백미꽃속 식물들은 생육형에 따라 큰조롱, 덩굴박주가리 등과 같은 덩굴성 식물과 백미꽃, 산해박, 양반풀 등의 직립성 식물까지 다양한 생육형을 가지고 있으며, 꽃의 형태와 화서 (inflorescence), 잎의 형태에서도 다양한 외부형태적 특징을 보인다 (Khanum et al., 2016). 백미꽃속 식물들은 공통적으로 줄기에 흰색 또는 미색을 띠는 라텍스 (latex) 성분의 유액을 함유하고 있으며, 열매의 형태도 모두 골돌과 (follicle)인 특징이 있다 (Khanum et al., 2016).
백미꽃속 식물들은 한국과 중국에서 전통 의약품으로 사용되어 왔으며, 스테로이드, 벤젠 및 파생 물질, 테르펜, 소량의 알칼로이드 등, 400 개 이상의 유용물질들을 포함하는 것으로 확인되었다. 최근 이러한 백미꽃 속 식물의 다양한 화학적 성분에 기인한 생리 활성 평가 및 약리효과 구명을 위한 연구가 활발하게 수행되고 있으며, 특히 항암, 항염증, 항바이러스, 식욕 억제 등의 효과를 나타내는 것으로 확인되었다 (Han et al., 2018). 따라서 백미꽃 속 식물이 약리학 및 의학 분야에서 유용한 생물자원으로 활용될 수 있을 것으로 전망되며, 이들의 산업적 연구 및 개발의 중요성이 확대되고 있다 (Han et al., 2018).
한반도 자생 식물인 C. wilfordii (큰조롱)는 산업적으로 사용되는 대표적인 백미꽃속 식물이며, 한약(생약)규격집에서는 큰조롱의 덩이뿌리를 백수오 (白首烏)라는 이름의 생약으로 규정하고 있다. 전통적으로 백수오는 근골 강화, 노화 억제, 자양 강장 등을 목적으로 한약재로 사용되어왔고, 최근 연구에서는 항산화 및 항균, 항간질, 항암효과를 가지고 있다고 보고되었다 (Kim et al., 2005; Choi et al., 2016; Li et al., 2016). 특히, 최근에는 여성 갱년기 증상을 완화시키는 효과에 기인하여, 건강기능식품의 기능성 원료로도 각광받고 있다 (Chang et al., 2012).
중국에 주로 분포하는 넓은잎큰조롱 (C. auriculatum Royle ex Wight)은 큰조롱의 대표적인 근연식물로서 이엽우피소 (耳葉牛皮消)라는 이름으로 알려져 있다. 한편 환경부 국립생물자원관에서 발간하는 국가생물종목록 2021년에는 넓은잎큰조롱을 C. boudieri로 규정하고 있고 C. auriculatum은 국명을 부여하고 있지 않아 학명과 국명의 정립이 필요하다 (Park et al., 2020). 국내에는 1990년대 이전에 도입되어 2000년대 초반까지는 ‘도입 백수오’ 등으로 불리며 농촌진흥청에서 재배방법 확립을 위한 연구가 수행되었고, 국내 백수오 시장 안정화를 위해 넓은잎큰조롱의 개발 및 소득 작물로서의 입식이 필요하다는 주장들이 제기되기도 하였다 (Kim et al., 2005; Nam et al., 2008).
2015년 가짜 백수오라는 오명을 얻은 넓은잎큰조롱은 당시 심각한 독성을 가지고 있는 것으로 언론에 보도되었으나, 2017년 식품의약품안전처에 의해 실시된 큰조롱 및 넓은잎큰조롱의 독성 검사에 따르면, 넓은잎큰조롱 열수 추출물의 독성시험 결과 1 일 섭취량 2,000 mg/kg까지 증량시킨 고용량 투여에서는 독성이 확인되었으나 1 일 섭취량 1,000 mg/kg의 용량 투여의 경우 큰조롱과 마찬가지로 독성을 나타내지 않는 것으로 보고 되었다 (Yu et al., 2022). 또한 넓은잎큰조롱 추출물의 간 보호 효능 등이 보고된 바 있어 넓은잎큰조롱의 독성과 효능에 대한 추가적인 연구가 요구된다 (Chen et al., 2019; Cui et al., 2019; Wang et al., 2021; Zhuang et al., 2021).
중국에서는 넓은잎큰조롱을 큰조롱과 같은 유용한 생약으로서 식품과 의약품 소재로 이용되고 있으며, 항암, 항우울, 항산화 등의 약리 효과를 가지고 있는 것으로 보고되고 있다 (Shan et al., 2005; Peng et al., 2008; Chen et al., 2019; Wang et al., 2021). 이러한 약리적 효능으로 인해 넓은잎큰조롱 자원의 중요성이 강조되고 있으며, 대사체와 약리 효능뿐 아니라 전사체, 유전체를 포괄하는 다양한 연구 결과들이 보고되고 있다 (Kim and Kim, 2019; Zhang et al., 2020).
넓은잎큰조롱은 1992년 생물 다양성 협약 (CBD, Convention on Biological Diversity)과 2014년 나고야 의정서 (Nagoya Protocol)의 정식 발효 이전에 이미 국내로 도입되었기 때문에 국내 생물자원으로써 활용될 수 있는 중요한 자원식물이다. 그러나 산업 자원으로서의 가능성에도 불구하고 넓은잎큰조롱은 대한민국 식품공전에 식품 원료로 인정되지 않아 식품소재로서의 사용은 여전히 제한되고 있으며 이에 대한 대책 마련이 필요하다 (MFDS, 2021).
본 연구에 사용된 거대백수오는 2021년 3월 강원도 영월군 약초 채집가에 의해 채집된 것으로, 뿌리의 크기가 큰조롱과 형태적으로 유사하고 전례 없이 거대한 크기를 가지고 있다. 그러나 뿌리에서 넓은잎큰조롱에서 발견되는 특징인 하얀 유액이 나와 특정 종 판별이 필요한 상황이었다. 이에 본 연구에서는 강원도 영월에서 발견된 거대한 식물을 ‘거대백수오’라 명명하고, 기 보고된 종 판별 분자표지들을 적용해 이 식물의 정확한 종 식별을 위한 분석을 수행하였다. 나아가 대표적인 식물 DNA 바코딩 지역을 대상으로 한 계통수 분석을 통해 명확한 종 판별, 기원 규명 및 식물자원으로서의 활용성 확대를 위한 연구를 수행하고자 하였다.
재료 및 방법
1. 식물 자원
2021년 3월 강원도 영월군 약초 채집가에 의해 발견되어 가칭 ‘거대백수오’로 명명된 식물자원 (이하 거대백수오)의 잔뿌리 일부를 분양받아 경기도 수원시 소재 서울대학교 농장에서 영양번식 시키고 형태적 특성을 조사하였다.
각 자원들은 경운 깊이 30 cm, 재식 간격 50 cm로 정식 하였으며, 흑색 비닐을 피복해 재배하였다. 큰조롱, 넓은잎큰조롱, 거대백수오는 동일 조건에서 재배되었다. 엽형의 비교는 거대백수오, 큰조롱, 넓은잎큰조롱의 영양 번식체에서 초엽이 발생한 후 90 일이 경과한 시점에 가장 큰 잎을 기준으로 수행하였다.
2. DNA 추출 및 분자표지 유전자검사
거대백수오, 큰조롱, 넓은잎큰조롱의 어린 잎을 액체질소를 사용하여 마쇄하였다. DNA 추출은 GeneAll Plant SV mini Kit (GeneAll Biotechnology, Seoul, Korea)을 사용하여 제조사의 실험방법에 따라 수행하였다.
추출된 각 DNA에 큰조롱와 넓은잎큰조롱 종판별 검사법에 사용되는 분자표지 7 개 (Table 1)를 적용하여 polymerase chain reaction (PCR)을 진행하였다. PCR 반응 시약은 inclone PCR kit (Inclone biotech, Seongnam, Korea)를 사용했다. PCR 반응액은 멸균수 17.75㎕, 10 × inclone Taq buffer 2.5 ㎕, dNTP Mix (each 10mM) 0.5㎕, forward primer (10 pmole/㎕) ㎕, reverse primer (10 pmole/㎕) 1㎕, 주형 DNA (10 ng/㎕) 1㎕, Inclone Taq polymerase (5 U/㎕) 0.25 ㎕로 조성하였으며, 각 primer 별 PCR 반응 조건은 이전 논문에서 제시한 방법을 따랐다 (Kim et al., 2013; Kim et al., 2017; Park et al., 2020).
Verriti 96 well thermal cycler (Thermo Fisher Scientific Inc., Waltham, MA, USA)를 이용해 PCR 후 증폭 산물은 2% agarose gel (Merck KGaA, Darmstadt, Germany)을 이용해 100 V 조건에서 70 분 동안 Mupid-exU system (Takara Bio Inc., Otsu, Japan)을 이용해 전기 영동한 후 ethidium bromide (Thermo Fisher Scientific Inc., Waltham, MA, USA)로 20 분간 염색하여 Gel documentation system (Lab Tech, Namyangju, Korea)을 이용해 확인하였다.
3. 근연종과의 염기서열 및 계통 분석
추출한 거대백수오의 DNA를 Illumina HiSeq X 플랫폼을 이용해 sequencing하여 염기 서열을 생산하였다 (LabGenomics Co. Ltd., Seongnam, Korea). 생산된 염기서열에서 ASCII code 33 이상의 고품질 서열들만 clc_quality_trim (Qiagen, Hilden, Germany)을 이용해 추출하였다 (Table 2).
거대백수오와 백미꽃속 근연종들과의 염기서열 비교분석을 위해 식물의 대표적인 DNA 바코딩 지역 5 곳 (ITS2, matK, rbcL, trnL-trnF, psbA-trnH)을 선별하였다. 생산된 거대백수오의 염기서열을 NCBI GenBank에 등록된 넓은잎큰조롱의 엽록체 및 핵 리보솜 서열을 참조하여 clc mapper (Version 4.21, Qiagen, Hilden, Germany)를 이용해 mapping하고 (Jang et al., 2016; Lee et al., 2022), 거대백수오의 바코딩 지역 서열을 완성하였다 (Table 3 and 4).
백미꽃속 식물 5 종 [큰조롱 (C. wilfordii), 넓은잎큰조롱 (C. auriculatum), C. bungei, C. boudieri, C. chinense]의 DNA 바코딩 지역 서열정보를 NCBI GenBank로부터 확보하여 거대백수오 염기서열과 함께 비교분석을 수행하였다 (Table 4).
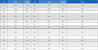
Reference information for five DNA barcoding regions of the Cynanchum species used in the phylogenic analysis.
변이 탐색 및 계통수 분석은 MAFFT (Katoh et al., 2019)를 사용하여 염기서열을 다중 정렬 후, IQ-TREE 프로그램을 사용하여 bootstrap 1,000 반복 조건에서 Maximum-Likelihood (ML) 방법으로 수행하였다 (Nguyen et al., 2015).
결과 및 고찰
1. 거대백수오 뿌리의 발견 및 보존
본 연구에 사용된 거대백수오 추정 식물은 2021년 3월 강원도 영월군에서 백수오와 형태적으로 유사하지만 일반 백수오보다 거대한 크기를 가진 식물 뿌리 (가칭 거대백수오; Giant Cynanchum)가 발견되었다 (Fig. 1A and 1C). 이 식물의 뿌리 길이는 10 m 이상으로 측정되었으며, 생근중은 20 kg이 넘는 것으로 확인되었다. 일반적인 야생 혹은 재배 환경에서는 큰조롱 (C. wilfordii) 단일 개체의 뿌리는 보통 1 kg을 넘기 어렵기 때문에 약초 전문가들은 거대백수오가 약 100년 이상의 수령을 가지고 있을 것으로 추정하였다 (Lee et al., 2022).

The new Cynanchum plant (Giant Cynanchum) which discovered in Yeongwol, Gangwon, Korea.A; the appearance of Giant Cynanchum after excavation. B; the model of the Giant Cynanchum was created using its actual dimensions and form (exhibited in the National Institute of Horticultural and Herbal Science, Rural Development Administration, Eumseong, Korea). C; map and location information (red dot) where Giant Cynanchum was found. D; a vegetatively propagated Giant Cynanchum. E; Leaves of C. wilfordii, C. auriculatum, and Giant Cynanchum.
거대한 길이와 무게를 가진 거대백수오 뿌리는 그 자체로 보존과 활용 가치가 있다고 판단되어 충청북도 음성에 소재한 농촌진흥청 원예특작과학원 산하 인삼특작부 전시관에 실물 모형으로 보존하여 많은 사람들에게 소개되고 있으며 (Fig. 1B), 연구적 활용을 위해 서울대학교 농장에서 유전자원으로 보존 증식하고 있다 (Fig. 1D).
2. 거대백수오의 형태적 특성과 기원종 추정
거대백수오 발견 당시 일부 전문가는 거대백수오 뿌리가 넓은잎큰조롱의 특성인 하얀 진액을 갖고 있기 때문에 큰조롱이 아닌 넓은잎큰조롱일 것으로 판단하였으나, 이외의 형태적 특징들에서 큰조롱과 유사한 특징들도 나타났기 때문에 뿌리 관찰을 통해 거대백수오를 특정종으로 확정짓는 것이 어려운 상황이었다.
지상부 관찰을 위해 서울대학교 농장에서 거대백수오의 종근으로부터 영양번식체를 유도하여 성숙한 개체들의 형태적 특징들을 관찰하였다 (Fig. 1D and 1E). 거대백수오의 엽형은 종횡비가 1 : 1에 가까운 심장형을 띠었으며, 전반적인 길이와 너비가 큰조롱 및 넓은잎큰조롱 보다 우량한 것으로 확인되었다. 거대백수오 잎의 외부 형태는 전반적으로 큰조롱 보다는 넓은잎큰조롱와 유사하나, 잎의 크기가 넓은잎큰조롱에 비해 큰 것으로 관찰되었다.
그러나 같은 백미꽃속에 속하는 국내 큰조롱 자원들의 지상부와 지하부 생육조사 결과에서 많은 형태적 다양성이 보고된 바 있으며 (Lee et al., 2022), 거대백수오에서 확인되는 형태적 특이성 역시 큰조롱나 넓은잎큰조롱의 종내 다양성으로부터 기인했을 가능성을 배제할 수 없어 형태적인 비교 분석 결과만을 기준으로 거대백수오를 특정종으로 동정하거나, 혹은 새로운 분류군으로 보고하는데는 무리가 있다고 판단된다.
3. 거대백수오 기원종 추정을 위한 유전자검사법 적용
거대백수오의 종 규명을 위해 큰조롱 및 넓은잎큰조롱의 엽록체 게놈 유래 종특이 분자표지 7 개를 이용하여 거대백수오의 종판별을 수행하였다 (Table 1 and Fig. 2).

The results of species identification for Giant Cynanchum using PCR analysis.Big; Giant Cynanchum, Cw; C. wilfordii; Ca; C. auriculatum. C1; Kim et al., 2021., C2; Kim et al., 2017, C3; Kim et al., 2013, C4 - C7; Park et al., 2020 (Table 1).
거대백수오의 유전자형은 식품의약품 안전처에서 가장 대표적으로 사용하는 검사법 C1에 의해 큰조롱의 유전자형을 나타냈지만, 또 다른 논문에 보고된 검사법 C2와 C3에서는 넓은잎큰조롱의 유전자형을 나타내었으나, C3에서는 희미하게 큰조롱의 유전자형이 동시 식별되어, 기존에 알려진 유전자 검사법들 간에 상이한 결과를 보여주었다 (Kim et al., 2013; Kim et al., 2017).
또한 본 연구팀에서 개발한 검사법 C4 - C7에서도 다양한 결과들이 확인되었다. 검사법 C5에서는 거대백수오가 넓은잎큰조롱와 같은 유전자형을 보였으며, 검사법 C4, C6, C7에서는 큰조롱 및 넓은잎큰조롱 두 종 모두와 다른 유전자형을 나타냈다 (Fig. 2).
결론적으로 7 가지 검사법마다 거대백수오의 유전자형이 큰조롱 혹은 넓은잎큰조롱의 유전자형을 보이거나, 전혀 다른 유전자형을 나타내는 등 상이한 결과들이 확인되었기 때문에 거대백수오를 큰조롱 혹은 넓은잎큰조롱 중 어느 하나의 종으로 특정할 수 없었다.
4. 거대백수오와 근연종들의 DNA 바코딩 지역 염기서열 및 계통수 분석
백미꽃속 식물들과의 염기서열 비교분석을 위해 거대백수오를 대상으로 약 1.5Gbp의 차세대 염기서열 데이터를 생산하고, 데이터 trimming 과정을 거쳐 약 1.1Gbp의 신뢰도 높은 염기서열들을 확보하였다 (Table 2).
큰조롱의 엽록체 및 핵리보솜 서열을 참조하여 거대백수오의 DNA 바코딩 지역 5 곳 (ITS2, matK, rbcL, trnL-trnF, psbA-trnH)의 염기서열을 완성한 후, 생산된 염기서열을 mapping한 결과 최소 144.11X에서 최대 365.71X까지 분석에 충분한 양의 데이터가 확보되었음을 확인하였다 (Table 3).
백미꽃속 식물 5 종 [큰조롱 (C. wilfordii), 넓은잎큰조롱 (C. auriculatum), C. bungei, C. boudieri, C. chinense]과 거대백수오의 DNA 바코딩 지역 5 곳의 염기서열을 기반으로 계통수 분석을 수행했으며, 서로 완전하게 일치하는 서열들은 병합된 삼각형으로 표시하였다 (Fig. 3)

Phylogenetic analysis based on five barcoding regions for Giant Cynanchum and related species.Con-trees denote the completely identical sequences. Each Cynanchum species is marked with different colored circles (A; ITS2, B; matK, C; rbcL, D; trnL-trnF, E; psbA-trnH).
분석 결과, 거대백수오는 근연종 식물 5 종 모두와 높은 유사성을 보였지만, 그 중에서도 넓은잎큰조롱와 가장 가까운 유연관계를 나타냈다. 4 곳의 DNA 바코딩 지역 (ITS2, matK, rbcL, trnL-trnF)에서 넓은잎큰조롱와 가장 높은 유사성을 보였으며, 특히 trnL-trnF 지역에서 서로 완벽히 동일한 서열을 갖는 것이 확인되었다 (Fig. 3A, 3B, 3C, 3D). 이와는 다르게 psbA-trnH 지역에서는 거대백수오가 C. bungei와 가장 가까운 유연관계를 나타냈다 (Fig. 3E).
백미꽃속 근연종 식물 5 종은 계통수 내에서 단계통이 아닌 분계군을 형성했다 (Fig. 3). 큰조롱는 matK, trnL-trnE 지역에서만 단계통을 형성하였으며, 넓은잎큰조롱는 4 개의 엽록체 유전자 지역에서 단계통을 형성하지 않고 다른 종들과 함께 분계군을 형성하였다 (Fig. 3B, 3C, 3D, 3E). C. bungei는 ITS2 지역의 계통수에서 큰조롱 분계군에 위치하였으며, matK와 trnL-trnH 지역의 계통수에서는 넓은잎큰조롱 분계군 내에 위치하였다. C. bungei와 C. boudieri는 다수의 계통수 내에서 큰조롱와 넓은잎큰조롱의 분계군 내에 혼재되어 위치하였으며, C. chinense는 다른 4 종의 근연식물들과 가장 먼 유연관계를 보여주었다 (Fig. 3).
본 계통수 분석 결과들은 기존 연구에서 보고된 것처럼 백미꽃속 식물들이 비교적 높은 종내 유전적 다양성을 갖고 있음을 보여주며, 그로 인해 종간에 서로 혼재되어 있는 분계군을 형성함으로서 근연종 간의 명확한 종 판별이 어렵다는 것을 보여 준다 (Lee et al., 2022). 또한 큰조롱, 넓은잎큰조롱, C. bungei, C. boudieri 간에는 해당 유전자 지역의 변이가 적어 계통 분석에 필요한 염기서열 정보가 부족했을 것으로 판단된다 (Fig. 3).
따라서, 백미꽃속 내 큰조롱 및 넓은잎큰조롱 근연종들의 보다 정확한 계통유연 관계를 파악하기 위해서는 각 종의 형태 및 유전적 한계에 대한 연구와 보다 넓은 영역의 대규모 유전체 정보를 활용한 포괄적인 분석이 필요할 것으로 생각된다.
5. 거대백수오의 기원 추정
형태적 특성 및 분자표지 기반 유전자형 비교와 계통수 분석을 통해 거대백수오가 근연종들 중에서 넓은잎큰조롱와 가장 유사한 것으로 확인되었지만, 모든 분석 결과들이, 넓은잎큰조롱와 완전히 일치하지는 않았다 (Fig. 1, 2, 3). 다른 근연종인 큰조롱, C. bungei, C. boudieri와도 일부 유전체적 유사성을 보였으며, 이를 통해 본 연구에서 사용된 여러 비교분석 결과들을 바탕으로 거대백수오를 특정 단일종으로 규정하는데는 무리가 있었다.
발견 당시 생육상태를 통해 수십 년 혹은 백 년 이상 생존해왔을 것이라 추정되는 거대백수오의 기원에 대해서 크게 두 가지로 가정해 볼 수 있다.
첫째, 거대백수오가 큰조롱처럼 국내에 존재하고 있던 자생 식물이지만 이번에 처음 발견된 식물자원일 가능성이다. 또 다른 가능성으로는 거대백수오가 여러 비교분석 결과에서 넓은잎큰조롱와 가장 높은 유사성을 보이는 것으로 보아 중국 등 국외에서 도입된 다양한 넓은잎큰조롱 자원 중 일부로서 오래전에 한반도에 유입되어 영월의 야산에서 수십 년 혹은 백 년 이상 존재해온 식물자원일 가능성이다. 후속 연구를 통해 첫 번째 가설을 뒷받침할 근거를 확보할 경우, 신종으로서의 생물 유전자원 등록 및 관련 연구를 통해 생물주권을 확보하는 등 산업적 활용 가치를 발굴할 필요가 있다.
두번째 가설의 근거를 확보할 경우, 거대백수오는 국제생물다양성협약이 체결된 1992년보다 이전에 이미 국내에 도입된 것으로 판단되어 해당 생물자원에 대한 고유 주권을 인정받을 수 있으므로, 생물다양성 협약 및 나고야 의정서 등 여러 국제법에 저촉되지 않으면서 국내에서 활용 가능한 유용한 생물자원으로서 가치를 인정받는 근거가 될 것이다 (Morgera et al., 2014).
따라서, 해당 유용 생물자원에 대한 주권을 확보하고 생물자원의 지속 가능한 이용을 위한 연구가 추가적으로 필요하다. 또한, 거대백수오의 사례에서 확인할 수 있듯이, 종내 다양성을 포함한 다양한 생물학적 요건을 고려한 국내 유용 생물자원의 발굴 방법의 재정립이 필요할 것으로 판단된다.
Acknowledgments
본 연구는 한국연구재단 기초연구사업(MIST, No. 2020R1A2C3007885)과 농촌진흥청 연구사업(과제번호: PJ017134)의 지원에 의해 이루어진 결과로 이에 감사드립니다.
References
-
Chang A, Kwak BY, Yi KT and Kim JS. (2012). The effect of herbal extract(EstroG-100) on pre, peri‐and post‐menopausal women: A randomized double‐blind, placebo‐controlled study. Phytotherapy Research. 26:510-516.
[https://doi.org/10.1002/ptr.3597]

-
Chen WH, Zhang ZZ, Ban YF, Rahman K, Ye BZ, Sun XI, Tan HY, Zheng XH, Liu HY, Xu LC Yan B and Han T. (2019). Cynanchum bungei Decne and its two related species for “Baishouwu”: A review on traditional uses, phytochemistry, and pharmacological activities. Journal of Ethnopharmacology. 243:112110. https://www.sciencedirect.com/science/article/pii/S0378874119307548, (cited by 2022 June 15).
[https://doi.org/10.1016/j.jep.2019.112110]

- Cui WW, Peng YR and Ding YF. (2019). Effects of total C-21 steroidal glucosides from Cynanchum auriculatum on oxidative stress pathway in mice with liver injury. China Journal of Chinese Materia Medica. 44:2960-2965.
-
Han L, Zhou X, Yang M, Zhou L, Deng X, Wei S, Wang W, Wang Z, Qiao X and Bai C. (2018). Ethnobotany, phytochemistry and pharmacological effects of plants in genus Cynanchum Linn.(Asclepiadaceae). Molecules. 23:1194. https://www.mdpi.com/1420-3049/23/5/1194, (cited by 2022 June 10).
[https://doi.org/10.3390/molecules23051194]

-
Jang W, Kim KY, Kim KH, Lee SC, Park HS, Lee JK, Seong RS, Shim YH, Sung SH and Yang TJ. (2016). The complete chloroplast genome sequence of Cynanchum auriculatum Royle ex Wight (Apocynaceae). Mitochondrial DNA Part A. 27:4549-4550.
[https://doi.org/10.3109/19401736.2015.1101557]

-
Katoh K, Rozewicki J and Yamada KD. (2019). MAFFT online service: Multiple sequence alignment, interactive sequence choice and visualization. Briefings in Bioinformatics. 20:1160-1166.
[https://doi.org/10.1093/bib/bbx108]

-
Khanum R, Surveswaran S, Meve U and Liede-Schumann S. (2016). Cynanchum(Apocynaceae: Asclepiadoideae): A pantropical Asclepiadoid genus revisited. Taxon. 65:467-486.
[https://doi.org/10.12705/653.3]

-
Kim CK and Kim YK. (2019). The multipartite mitochondrial genome of Cynanchum auriculatum(Gentianales: Apocynaceae). Mitochondrial DNA Part B 4:3360-3361.
[https://doi.org/10.1080/23802359.2019.1673683]

-
Kim JH, Moon JC, Kang TS, Kwon KS and Jang CS. (2017). Development of cpDNA markers for discrimination between Cynanchum wilfordii and Cynanchum auriculatum and their application in commercial C. wilfordii food products. Applied Biological Chemistry. 60:79-86.
[https://doi.org/10.1007/s13765-017-0252-5]

- Kim MJ, Song BH, Nam SY, Kim IJ, Lee CH and Yun T. (2005). Effects of nonsupporting methods on growth and yield of Cynanchum auriculatum Royle ex Wight. Korean Journal of Medicinal Crop Science. 13:268-272.
- Kim MK, Kim KY, Kim N, Kang JH, Shim YH and Lim NJ. (2021). Composition for distinguishing a plant of genus Cynanchum. Korea. Patent. 10-22840510000. (2021. 07. 26).
- Kim MK, Wang H, Kim YJ, Sathiyamoorthy S, Kwon WS and Yang DC. (2013). Molecular authentication by multiplex-PCR of three similar medicinal plant species: Cynanchum wilfordii, Cynanchum auriculatum and Polygonum multiflorum(Fallopia multiflorum). Journal of Medicinal Plants Research. 4:2584-2589.
-
Lee SH, Kim JS, Park HS, Koo HJ, Waminal NE, Pellerin RJ, Shim HA, Lee HO, Kim E, Park JY, Yu HS, Kim HH, Lee JH and Yang TJ. (2022). Genome structure and diversity among Cynanchum wilfordii accessions. BMC Plant Biology. 22:4. https://link.springer.com/article/10.1186/s12870-021-03390-y, (cited by 2022 June 10).
[https://doi.org/10.1186/s12870-021-03390-y]

-
Mafra I, Ferreira IM and Oliveira MBPP. (2008). Food authentication by PCR-based methods. European Food Research and Technology. 227:649-665.
[https://doi.org/10.1007/s00217-007-0782-x]

- Ministry of Food and Drug Safety(MFDS). (2021). Ministry of food and drug safety notification(No. 2021-54): Food code. Ministry of Food and Drug Safety. Cheongju, Korea. p.22-66.
-
Morgera E, Tsioumani E and Buck, M. (2014). Unraveling the nagoya protocol: A commentary on the nagoya protocol on access and benefit-sharing to the convention on biological diversity. Brill. Leiden, Netherlands. p.1-444.
[https://doi.org/10.1163/9789004217188]

- Nam SY, Kim MJ, Kim IJ, Lee JK, Rho CW, Yun T and Min KB. (2008). Effect of plowing depth on growth and tuber yield in C. auriculatum introduced from china. Korean Journal of Medicinal Crop Science. 16:69-73.
-
Nguyen LT, Schmidt HA, Von Haeseler A and Minh BQ. (2015). IQ-TREE: A fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Molecular Biology and Evolution. 32:268-274.
[https://doi.org/10.1093/molbev/msu300]

-
Park HS, Jayakodi M, Lee SH, Jeon JH, Lee HO, Park JY, Moon BC, Kim CK, Wing RA, Newmaster SG, Kim JY and Yang TJ. (2020). Mitochondrial plastid DNA can cause DNA barcoding paradox in plants. Scientific Reports. 10:6112. https://www.nature.com/articles/s41598-020-63233-y, (cited by 2022 Aug 10).
[https://doi.org/10.1038/s41598-020-63233-y]

- Park JS, An JH, Kim YS, Kim DH, Yang BG and Kim TH. (2020). Database of national species list of Korea: The taxonomical systematics platform for managing scientific names of Korean native species. Journal of Species Research. 9:233-246.
-
Peng Y, Li YB, Liu XD, Zhang JF and Duan JA. (2008). Antitumor activity of C-21 steroidal glycosides from Cynanchum auriculatum Royle ex Wight. Phytomedicine. 15:1016-1020.
[https://doi.org/10.1016/j.phymed.2008.02.021]

-
Shan L, Zhang WD, Zhang C, Liu RH, Su J and Zhou Y (2005). Antitumor activity of crude extract and fractions from root tuber of Cynanchum auriculatum Royle ex Wight. Phytotherapy Research. 19:259-261.
[https://doi.org/10.1002/ptr.1678]

-
Wang L, Cai F, Zhao W, Tian J, Kong D, Sun X, Liu Q, Chen Y, An Y, Wang F, Liu X, Wu Y and Zhou H. (2021). Cynanchum auriculatum Royle ex Wight., Cynanchum bungei Decne. and Cynanchum wilfordii(Maxim.) Hemsl.: Current research and prospects. Molecules. 26:7065. https://www.mdpi.com/1420-3049/26/23/7065, (cited by 2022 June 10).
[https://doi.org/10.3390/molecules26237065]

- Wu Z, Ding L and Zhao S. (1991). Chemical constituents and pharmacological effects of Cynanchum Linn. World Phytomedicine. 6:147-154.
-
Yu CW, Hong SH, Lee JH, Jung KK, Oh JH, Jeong JY, Kwon HJ, Kang JK and Yang JY. (2022). Comparative sub-chronic toxicity studies in rats of two indistinguishable herbal plants, Cynanchum wilfordii(Maxim.) Hemsley and Cynanchum auriculatum Royle ex Wight. Food Science and Biotechnology. 31:759-766.
[https://doi.org/10.1007/s10068-022-01072-5]

-
Zhang M, Hong LZ, Gu MF, Wu CD and Zhang G. (2020). Transcriptome analyses revealed molecular responses of Cynanchum auriculatum leaves to saline stress. Scientific Reports. 10:449. https://www.nature.com/articles/s41598-019-57219-8, (cited by 2022 June 10).
[https://doi.org/10.1038/s41598-019-57219-8]

- Zhuang ZR, Wang ML, Peng YR and Shen MQ. (2021). Effect of C21 steroidal glycoside of Cynanchum auriculatum on liver and kidney fibrosis through TLR4 pathway. China Journal of Chinese Materia Medica. 46:2857-2864.