
일천궁에 발생하는 오이모자이크바이러스의 현장 검출을 위한 Lateral Flow Reverse Transcription Recombinase Polymerase Amplification (LF-RT-RPA) 개발
This is an open access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Quality and quantity of Cheongung (Cnidium officinale Makinoi) is reduced by plant viral infections. Therefore, it is necessary to develop effective detection methods for diagnosis of problematic viruses in Cheongung farms. This study aimed to develop lateral flow reverse transcription recombinase polymerase amplification (LF-RT-RPA) assay for the detection of cucumber mosaic virus (CMV) in C. officinale.
Total RNA was extracted from CMV-infected Cheongung plant and subjected to RT-RPA reaction with RPA_CMV-CP-F1/R1 primers targeting the CMV RNA3 sequence. When specificity was examined using lateral flow Immunostrip, experimental and control lines were generated only in the sample from the CMV-infected plant, while control lines were observed in samples from cnidium vein yellowing virus-1, cnidium vein yellowing virus-2, and apple stem grooving virus-infected Cheongung plants. The optimal temperature and reaction time for LF-RT-RPA assay were determined to be 37oC and 15 min. On-site diagnosis carried out to detect CMV in 7 Cheongung plants showing viral-infection symptoms, confirmed six of the plants infected with CMV.
CMV, one of the prevalent plant viruses, was effectively detected using LF-RT-RPA assay without the need of any equipment on farms. Detection limit for CMV in C. officinale Makinoi was up to 100 fg of total RNA, suggesting that the LF-RT-RPA assay developed in this study is suitable for on-site detection of CMV in Cheongung farms.
Keywords:
Cnidium officinale Makino, Cucumber mosaic virus, Detection, Lateral Flow Immunoassay, On-site, Reverse Transcription-Recombinase Polymerase Amplification서 언
천궁 (Cnidium officinale Makinoi)은 미나리과 (Apiaceae)에 속하는 식물로 뿌리줄기를 한약재에 이용하는 대표적인 고소득 약용작물이며 항산화와 미백효과, 항염 효과, 진통 작용 등 효능이 알려져 있다 (Cho et al., 1996, Oh et al., 2010; Um et al., 2017).
천궁은 기후변화에 예민하여 해발고도가 높은 서늘한 지역에서만 재배가 가능하며, 특히 연작장해로 인해 매년 재배지를 옮겨야 하는 단점으로 재배면적이 감소되고 있다. 천궁의 재배면적은 1997년 789 ha에서 2017년 185 ha로 약 76.5% 감소되었으며, 생산량도 1997년 2,294 M/T (metric ton)이었으나 2017년 1,290 M/T로 약 43.8% 감소한 것으로 발표되었다. (NIHHS, 2017; Kim et al., 2015; Seo et al., 2018; Jung et al., 2019; Yun et al., 2022).
천궁의 정확한 원산지는 확인되지 않았지만 일천궁은 일본에도시대에 중국을 통해 들어온 것으로 알려져 원산지가 일본으로 추정되고 있으며, 토천궁은 원산지가 중국의 사천성 인근으로 추정되고 있다 (Song et al., 2009). 대한약전에 천궁이라고 기재되어 있는 약재는 일천궁을 말하며, 국내 천궁 농가에서 재배하는 천궁은 일천궁이 대다수로 알려져 있다 (Hwang and Yang, 1998; Park, 1998).
일천궁에 발생하는 것으로 알려진 병해로는 Phytoplasma 감염에 의한 빗자루병, Colletotrichum gloeosporioides 감염에 의한 탄저병, Fusarium solani 감염에 의한 시들음병이 대표적으로 보고되었다 (Choi et al., 1985; Jung et al., 2018; Han et al., 2021). 일천궁에 발생한 바이러스병은 apple stem grooving virus (ASGV), cnidium vein yellowing virus-1 (CnVYV-1), cnidium vein yellowing virus-2 (CnVYV-2), cucumber mosaic virus (CMV), cnidium virus 1 (CnV1), cnidium virus 2 (CnV2), cnidium virus X (CnVX)가 보고되었다 (Hord et al., 2001; Yoo et al., 2015; Yang et al., 2018; Honma et al., 2019;Ortega-Acosta et al., 2019; Belete et al., 2022; Chung et al., 2022; Lee et al., 2023; Park et al., 2023).
천궁 감염 식물 바이러스들 중 CMV (한글명으로는 오이모바이크바이러스)는 Bromoviridae 과 (family), Cucumovirus 속 (genus)에 속하는 식물 바이러스이다. CMV의 입자는 약 28 nm 크기의 구형이며 게놈 RNA는 3 개의 분절 게놈으로 구성되어 있다. RNA1은 약 3.4 kb이며 methyltransferase 도메인과 helicase motif를 가지는 약 116 kDa의 1a 단백질을 암호화하고, RNA2는 약 3.1 kb이며 RNA 의존성 RNA 중합효소 (RNA-dependent RNA polymerase) GDD motif를 가지는 약 86 kDa의 2a 단백질과 기주식물의 RNA 침묵 경로 (RNA silencing pathway)의 억제 단백질 (suppressor) 역할을 하는 약 19 kDa의 2b 단백질을 암호화하고 있다 (Palukaitis and Garcia-Arenal, 2003). RNA3은 약 2.3 kb 크기로 구성되어 있으며 31 kDa의 3a 단백질과 24 kDa 크기의 외피 단백질을 암호화한다 (Palukaitis et al., 1992; Bujarski et al., 2019; Bujarski, 2021). CMV는 101과에 속하는 1241 개의 종 (species) 식물을 감염시킬 수 있으며 현재 알려진 식물 바이러스 중 가장 넓은 기주 범위 (host range)를 가지고 있는 것으로 알려져 있다 (Edwardson and Christie, 1991; Palukaitis et al., 1992; Bujarski et al., 2019; Bujarski, 2021). CMV는 다양한 국내 재배 채소 작물, 화훼작물, 과수 작물뿐만 아니라 약용작물과 잡초 등에서 가장 빈번하게 피해를 주는 바이러스이다 (Cho et al., 2007; Kim et al., 2011).
CMV가 감염된 식물들은 모자이크 증상 (mosaic symptom), 괴저 (necrosis), 엽맥 퇴록 (vein clearing), 원형 반점 (ringspot), 황화 (yellowing) 등 다양한 증상이 나타나며, 생육 불량, 과실 품질 저하에 따른 경제적인 피해가 발생하게 되므로 CMV는 농업 현장에서 가장 중요하고 시급한 대책이 필요한 바이러스로 여겨지고 있다 (Cho et al., 2007; Kim et al., 2011). 특히 바이러스에 감염된 묘목은 피해가 장기적으로 나타나며, 묘목을 제거하는 것 이외에는 다른 치료방법이 없다. 또한 CMV에 감염된 천궁으로부터 고추, 가지 등 타작물로 매개충에 의한 전염, 확산이 우려되기 때문에 CMV 감염된 천궁 묘목 및 종근을 조기에 진단하여 감염된 식물은 폐기하고 건전묘로 유지하는 것이 중요하다. (Kim et al., 2011; Yoo et al., 2015; Honma et al., 2019).
Recombinase polymerase amplification (RPA)은 등온 증폭법 중 하나로 핵산 검출기반으로 개발되었다 (Piepenburg et al., 2006). PCR과 기본 원리는 유사하나 RPA에 사용되는 DNA 중합효소는 주형교환활성 (strand-displacement activity)이 있어 기존의 높은 온도에 의한 DNA의 변성(denaturation) 단계는 재조합효소 (recombinase)에 의해 이루어진다. RNA를 주형으로 하는 식물 바이러스의 경우 Reverse transcription RPA (RT-RPA)를 이용하여 증폭할 수 있으며 (Lee et al., 2020), 형광 표지를 이용한 real-time RPA도 식물바이러스 진단 기술로 개발되었다 (Kim and Lee, 2017; Srivastava et al., 2019). Lateral flow immunoassay 기술을 활용한 dipstick을 이용하는 lateral flow RT-RPA (LF-RT-RPA) 방법으로 (Zhao et al., 2019), 바이러스 유전자를 증폭할 경우 36℃ - 42℃ 온도에서 30 분 내에 별도의 장비가 없이 식물 바이러스 진단이 가능하다 (Crannell et al., 2014; Li et al., 2019) (Fig. 1).

Viral symptoms on Cnidium officinale.(A); Cultivation of Cnidium officinale in farms (Yeongyang-gun, Gyeongsangbuk-do, Republic of Korea). (B); Symptomatic leaves of C. officinale (C); Mosaic and yellowing symptoms of a C. officinale plant infected with CMV.
본 연구에서는 기존 RT-RPA 진단에 반드시 필요한 효소인 nfo endonuclease를 사용하지 않아도 되는 새로운 방법을 적용하여 천궁에 발생하는 기주 범위가 넓어 가장 진단이 시급하고 중요한 CMV를 검출할 수 있는 LF-RT-RPA 검출 기술을 개발하고자 연구를 수행하였다.
재료 및 방법
1. 바이러스원
모자이크 병징 또는 황화 병징을 보이는 10 개의 일천궁 식물체들을 경상북도 영양군 농가들로부터 2020년에 채집하였다. 식물체들은 화분에 옮겨 심어 격리된 온실에서 유지하였다 (Fig. 1C).
2. Total RNA 추출과 RT-PCR 반응
채집한 일천궁 식물체들의 잎에서 CMV 감염 여부를 확인하기 위해 RNeasy® Plant Mini kit (Qiagen, Hilden, Germany)를 이용하여 제조회사에서 지시한 방법대로 total RNA를 추출하였다.
일천궁의 잎 조직 (100 ㎎)을 막자사발에 넣고 액체질소를 부은 후 곱게 갈고 2 ㎖ 튜브로 옮겼다. 즉시 450 ㎕의 RLC 용액을 첨가하여 교반한 후 QIAshredder spin column에 옮겨 상온에서 2 분 동안 13,000 rpm으로 원심분리를 실시하였다. 여과된 약 400 ㎕ 상층액을 새로운 1.5 ㎖ 튜브에 옮기고 200 ㎕의 100% 에탄올을 첨가하고 혼합하였다. 약 650 ㎕의 혼합액을 RNeasy Mini spin column에 옮겨 상온에서 15 초 동안 8,000 g로 원심분리 하였다. 여과액은 버리고 700 ㎕의 RW1을 RNeasy Mini spin column에 첨가하여 상온에서 15 초 동안 8,000 × g로 원심분리를 실시하였다. 여과액은 제거하고 500 ㎕의 RPE 용액을 RNeasy Mini spin column에 첨가하여 상온에서 15 초 동안 8,000 × g로 원심 분리하였다. 다시 한번 여과액을 제거하고 500 ㎕의 RPE 용액을 RNeasy Mini spin column에 첨가하여 상온에서 15 초 동안 8,000 × g로 원심분리를 진행하였다. 통과액을 제거하고 RNeasy spin column을 새로운 1.5 ㎖ 튜브에 놓고 50 ㎕의 멸균수를 spin column 막에 직접 첨가한 후 상온에서 15 초 동안 8,000 × g로 원심분리를 하였다. 이후 Epoch Microplate Spectrophotometer (BioTek Inc., Winooski, VT, USA)를 이용하여 Gen5 Take3 Module을 통해 total RNA 농도를 측정 후 –80℃에 보관하여 추후 실험에 사용하였다.
채집한 일천궁에 감염된 바이러스를 진단하기 위해 국내 천궁에 발생이 보고된 바이러스 4 종 (CnVYV-1, CnVYV-2, CMV, ASGV)을 검출할 수 있는 RT-PCR 프라이머 세트를 제작하였으며 식물 mitochondrial NADH dehydrogenase subunit 5 (nad5) 유전자를 특이적으로 증폭시킬 수 있는 프라이머를 RT-PCR용 내부 대조구로 사용하였다 (Lee and Chang, 2006; Table 1).
RT-PCR은 Topscript one-step RT-PCR kit (Enzynomics, Daejeon, Korea)를 이용하여 수행하였다. 반응액의 총량은 5 ㎕ one-step RT-PCR kit, 1 ㎕ 정방향 프라이머 (10 pmole/㎕), 1 ㎕ 역방향 프라이머 (10 pmole/㎕), 1 ㎕ RNA, 13 ㎕ 증류수를 포함하여 총 20 ㎕로 반응시켰다.
RT-PCR 반응은 역전사 과정을 50℃에서 30 분 진행하고 초기 변성을 95℃ 10 분 (1 회) 반응시킨 후 변성을 95℃ 30 초, 프라이머 재결합은 55℃ 30초, 가닥 신장을 72℃ 40 초로 프로그래밍을 하여 40 cycle을 반복하였으며 마지막으로 남은 가닥의 신장을 위해 72℃에서 5 분 동안 반응을 시킨 후 4℃로 종료하였다.
3. 프라이머 세트 제작
LF-RT-PCR를 이용한 CMV 검출용 특이적 프라이머 세트를 제작하기 위해, 국내에서 분리한 CMV 계통 (isolates)을 포함하여 외국에서 보고된 CMV 계통들의 유전자 정보 (NCBI (https://www.ncbi.nlm.nih.gov/)를 이용하여 다중 염기서열 배열 분석 (multiple sequence alignment)을 실시하였다 (Table 2). 다중 염기서열 배열 분석은 EMBL-EBI (https://www.ebi.ac.uk)에서 제공하는 Clustal Omega 소프트웨어를 이용하였다.
CMV 검출을 위한 RT-RPA 프라이머들은 30 - 35 염기 크기 GC content는 30 − 70%로 설정하였으며, 등온으로 증폭되므로 Tm (melting 온도)는 고려하지 않았다. RT-RPA 증폭 산물의 크기는 80 bp − 400 bp로 설정하고 하나의 특정 염기가 길게 이어지거나 특정 염기서열이 반복적으로 증폭되는 것을 피하여 프라이머 세트를 고안하였다.
프라이머의 위치는 다중 염기서열 배열 분석 결과에서 CMV 계통들 간의 변이가 적은 최대한 보존된 염기서열 내에서 이루어졌으며 Integrated DNA technologies (IDT, https://sg.idtdna.com)에서 제공하는 OligoAnalyzer Tool을 이용하여, 위에서 서술된 기준값을 적용하였다. 프라이머 세트들은 전문 염기제작 회사에 의뢰하여 합성하였다 (Cosmogenetech, Seoul, Korea).
최종 RT-RPA 진단용 후보 프라이머들은 CMV RNA1 서열을 이용한 RPA_CMV-RC-F1/R1 세트, CMV RNA2 서열을 이용한 RPA_CMV-RP-F1/R1, CMV RNA3 서열을 이용한 RPA_CMV-CP-F1/R1을 제작하였다 (Table 3).
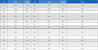
Modified primer sets for detection of CMV using lateral flow reverse transcription recombinase polymerase amplification (LF-RT-RPA).
RT-RPA 진단을 위해 nfo 프로브를 사용하지 않고 lateral flow assay로 RT-RPA 산물을 육안으로 확인하기 위해 각각 정방향 프라이머와 역방향 프라이머 5’ 말단에는 각각 항원으로 사용될 6-carboxyfluorescein (6-FAM)과 biotin을 부착하여 최종 산물의 확인 편리성을 도모하였다 (Table 3 and Fig. 2).
4. LF-RT-PCR 검출 반응
제작된 RT-RPA 프라이머 세트가 CMV를 특이적으로 검출 할 수 있는지 확인하고 최적의 반응 온도와 시간 조건, total RNA 양에 따른 검출 한계를 확인하였다. RT-RPA에는 RPA 반응을 위한 TwistAmp® Basic kit (TwistDX, Cambridge, England)와 역전사를 위한 역전사효소로 RevertAid reverse transcriptase (ThermoScientific, Waltham, MA, USA)를 이용하였다. 반응액은 동결 건조된 TwistAmp® basic pellet이 있는 튜브에 정방향 프라이머 (2 pmole/㎕)와 역방향 프라이머 (2 pmole/㎕) 각각 2.4 ㎕, rehydration buffer 29.5 ㎕, 역전사효소 1 ㎕, 주형 1 ㎕를 포함하여 증류수로 총량을 47.5 ㎕ 로 맞춘 반응 혼합물을 만들었다.
각 반응액의 동시적인 반응 시작을 위해 RPA 반응 시작에 필요한 280 mM 마그네슘아세테이트 용액 2.5 ㎕를 반응 튜브 뚜껑에 첨가하고 36℃ − 42℃에서 30 분 이내로 설정하여 수행하였다. RT-PCR과 RT-RPA의 증폭 산물을 전기영동을 수행하여 확인하였다. 또한 부직포로 둘러싸인 핫팩 (규격: 분말형 50 g, 평균 온도 40℃, 지속시간 10 시간)을 이용하여 위에서 서술한 total RNA를 반응액에 넣은 시료들을 반응시켰다.
전기영동은 2.0% 아가로스 젤에서 확인하였으며 1 × TAE buffer (Enzynomics, Daejeon, Korea), LE Agarose (Lonza, Basel, Switzerland), 6 × DNA loading buffer (Enzynomics, Daejeon, Korea), 100 bp DNA ladder marker (Enzynomics, Daejeon, Korea), ethidium bromide (EtBr)이 사용되었다. 최종 산물의 검출은 Gel Doc XR+ gel documentation system (Bio-Rad, Hercules, CA, USA)을 사용하여 확인하였다. RT-RPA의 증폭 산물을 육안으로 확인하기 위해 lateral flow assay를 이용하였다. Lateral flow immunostrip으로는 Milenia GenLine HybriDetect-1 (Milenia Biotec GmbH, Gießen, Germany)을 사용하였다.
제조사의 매뉴얼에 따라 0.1% PBST 용액으로 RT-RPA 증폭 산물을 최종 2.0% 희석액을 만든 후 lateral flow immunostrip의 샘플 적용 부위에 10 ㎕ 떨어뜨린 후 immunostrip을 100 ㎕의 HybriDetect assay 용액에 담가 대조선 (control line)과 실험선 (test line) 생성 여부를 확인하였다.
결 과
1. CMV 검출을 위한 LF-RT-RPA 검출 특이성
제작한 RT-PCR 프라이머 세트를 이용해 RT-PCR로 진단한 결과 채집한 일천궁 식물체에서 4 가지 바이러스 (CMV, CnVYV-1, CnVYV-2, ASGV)를 비특이적 반응 없이 검출할 수 있었으며, 단독 감염된 식물체는 온실에서 격리하여 유지하였다. CMV에 대한 RT-RPA 검출 특이성을 확인하기 위해 3 개의 프라이머 세트를 이용하여 CMV 단독 감염된 식물에서 total RNA를 추출하여 RT-RPA 반응을 시킨 결과 CMV RNA3 서열을 이용한 RPA_CMV-CP-F1/R1 프라이머 세트에서만 특이성이 관찰되었다 (data not shown).
따라서 이후 실험에서는 RPA_CMV-CP-F1/R1 프라이머들을 사용하여 RT-RPA 실험을 수행하였다. 이 프라이머의 CMV 검출 특이성은 CMV, CnVYV-1, CnVYV-2, ASGV에 단독 감염된 일천궁 식물체의 total RNA를 이용하여 RT-RPA를 수행하여 확인하였다 (Fig. 3A). 오직 CMV에서만 RT-RPA 산물이 아가로스 젤 전기영동에서 확인이 되었으며, CnVYV-1, CnVYV-2, ASGV 감염 식물 시료에서는 검출되지 않았다. RT-PCR로 일천궁 식물체들의 각 바이러스의 존재를 중복으로 확인하여 특이성을 검증하였다 (Fig. 3A).

Detection specificity of the reverse transcription-recombinase polymerase amplification (RT-RPA) combined with a lateral flow immunostrip for the detection of cucumber mosaic virus (CMV).A single CMV, Cnidium vein yellowing virus-1 (CnVYV-1), Cnidium vein yellowing virus-2 (CnVYV-2), and apple stem grooving virus (ASGV)-infected Cnidium officinale plants were used as templates for the CMV detection of RT-PCR and LF-RT-RPA assay. (A) RT-PCR and RT-RPA for CMV detection were visualized on 2% agarose gels. The primers used in RT-RPA and RT-PCR were described in Table 1. M, 100bp DNA ladder (Enzynomics, Daejeon, Korea). The mitochondrial NADH dehydrogenase gene (nad5) of Cnidium offinale was used as an internal control (Lee and Chang, 2006). (B) LF-RT-RPA assay showed test line and control line in a sample from CMV-infected C. officinale. A lateral flow immunostrip assay was done with fifty-fold dilutions of RT-RPA product in 1× phosphate buffered saline containing 0.1% Tween 20, according to manufacturer’s instructions (TwistDX, Cambridge, England).
LF immunostrip을 이용하여 특이성을 조사한 결과, CMV에 감염된 일천궁 시료에서만 실험선과 대조선이 생성되었으며, CnVYV-1, CnVYV-2, ASGV 감염 식물 시료에서는 대조선만 생성됨을 확인하였다 (Fig. 3B). 이런 결과는 CMV 검출에 RT-RPA의 특이성이 RT-PCR과 유사한 수준이며 CnVYV-1, CnVYV-2, ASGV와 비특이적 교차반응이 이루어지지 않았다는 것을 의미하며, lateral flow immunostrip을 이용한 검출이 효과적이며 특이적 반응임을 제시해준다.
2. LF-RT-RPA 반응 최적화
천궁에서 CMV 검출을 위한 LF-RT-RPA 최적 반응 온도조건을 확인하기 위해 RT-RPA를 수행하여 전기영동과 lateral flow assay로 확인한 결과, 36℃ − 42℃ (각 1℃ 범위 설정) 모든 실험온도에서 211 bp RT-RPA 산물이 합성되었다. 유사하게 LF immunostrip에서는 모든 실험 온도에서 실험선과 대조선이 생성되었다 (Fig. 4).

Optimization of reaction temperature of reverse transcription-recombinase polymerase amplification assay combined with a lateral flow immunostrip assay (LF-RT-RPA).The LF-RT-RPA was performed on C. officinale leaves infected with CMV for 30 min at 36, 37, 38, 39, 40, 41, 42oC, respectively. (A) RT-RPA products were analyzed in 2.0% agarose gel. M, 100bp DNA ladder (Enzynomics, Daejeon, Korea). (B) The same RT-RPA products were visualized using lateral flow immunostrips. The assay was visualized with a distinct line at the test line of the immunostrip from 15 min onwards.
따라서 36℃ − 42℃ 구간의 온도에서 RPA CMV-CP-F1/R1 프라이머 세트에 의한 증폭의 차이는 확인되지 않았으며 위 범위 온도에서 CMV를 안정적으로 검출할 수 있음을 확인하였다.
CMV 검출을 위한 RT-RPA 반응에 필요한 최적 반응 시간을 확인하기 위해 RT-RPA 반응 시간을 30 분 이내에 7 개 간격을 설정(1 분, 5 분, 10 분, 15 분, 20 분, 25 분, 30 분)하여 분석하였다. LF immunostrip은 15 분 증폭 시간부터 실험선과 대조선이 명확하게 생성되었으며 이런 결과는 같은 실험군을 아가로스겔 전기영동으로 다시 확인하였다 (Fig. 5).

Optimization of reaction time of reverse transcription-recombinase polymerase amplification assay combined with a lateral flow immunostrip assay (LF-RT-RPA).The LF-RT-RPA was performed on C. officinale leaves infected with CMV at 37℃ for 1, 5, 10, 15, 20, 25, 30 min, respectively. (A) RT-RPA products were analyzed in 2.0% agarose gel, M, 100bp DNA ladder (Enzynomics, Daejeon, Korea), (B) The same RT-RPA products were visualized using lateral flow immunostrips. The assay was visualized with a distinct line at the test line of the immunostrip from 15 min onwards. Non-template control (NTC).
LF-RT-RPA를 이용한 CMV 검출할 수 있는 한계를 조사하기 위해 CMV 단독 감염된 일천궁 식물체로부터 total RNA를 추출하여, 시작 농도를 10 나노그램 (nanogram, ng)으로 조정 후 각각 10 분의 1 씩 희석하여 10 펨토그램 (femtogram, fg) 까지 LF-RT-RPA 반응의 주형으로 사용하였다.
LF-RT-PCR 결과는 일천궁 100 fg 수준의 total RNA에서 CMV를 검출할 수 있었으며, 이의 결과는 아가로스 겔 전기 영동으로 증명하였다 (Fig. 6). 이런 결과를 통해 일천궁에서 CMV 검출을 위한 최적 LF-RT-RPA 반응에 필요한 최적 반응 조건을 안정적인 LF-RT-PCR 산물의 검출량과 가급적 검출 가능한 온도 중 낮은 온도로 반응시켜 안정적 산출물을 획득하는 방법 등 종합적으로 고려하여 37℃, 15 분으로 결정하였다.

Determination of detection limit for CMV using reverse transcription-recombinase polymerase amplification assay combined with a lateral flow immunostrip assay (LF-RT-RPA).The starting concentration was 10 ng total RNA (indicated 100) Total RNA of CMV-infected C. officinale was s diluted tenfold serially to a final concentration of 10 fg per microliter (indicated 10-6). RT-RPA was performed at 37℃ for 15 min and the synthesized products were visualized in 2.0% agarose gel (A) or using lateral flow immunostrips (B). The immunostrips with non-template control (NTC) were not visualized any amplified DNA products in 2.0 agarose gel or in any test line. M, 100bp DNA ladder (Enzynomics, Daejeon, Korea).
3. 일천궁 재배농가에서 LF-RT-RPA 이용 CMV 검출
별도의 장비 없이 현장 진단의 효용성을 더 높이기 위해 CMV 단독 감염된 일천궁 시료를 이용하여 LF-RT-RPA 반응을 실온 (25℃), 체온, 핫팩을 이용하여 30 분 동안 비교하였다.
그 결과 실온에서 LF-RT-RPA를 수행했을 때는 CMV를 검출할 수 없었으나, 손으로 쥐고 있는 방법과 핫팩으로 감싸는 방법으로 LF-RT-RPA를 수행했을 경우 thermal cycler에서 반응을 시킨 것과 유사하게 CMV를 검출을 확인할 수 있는 실험선과 대조선이 생성되었다 (Fig. 7). 이러한 결과는 별도의 장비 없이 LF-RT-RPA를 이용하여 CMV를 효과적으로 검출 할 수 있음을 말해준다.

Determination of CMV detection using reverse transcription-recombinase polymerase amplification assay combined with a lateral flow immunostrip assay (LF-RT-RPA) without equipment.The RT-RPA for CMV detection was performed at room temperature (25℃), in hand (approximately 37℃), hand warmer (so called hot pack, approx. 42℃) for 15 min. A programmable thermal cycler (37℃, 15 min) was used for direct comparison as a control. The RT-RPA products were visualized in 2.0% agarose gel (A) or using lateral flow immunostrips (B). The immunostrips with no-template control (NTC) were not visualized any amplified DNA products in 2.0 agarose gel or in any test line of the immunostrips.
LF-RT-RPA을 이용하여 일천궁에서 CMV 검출을 위한 현장 진단을 실시하였다. 경상북도 영양군 소재 일천궁 재배 농가 재배지에서 황화, 모자이크 등 바이러스 의심 증상을 보이는 7 개 일천궁 식물체를 확보하였다. LF-RT-RPA 진단 결과 7 개 중 6 개의 식물체가 CMV에 의해 감염되어 있음을 확인할 수 있었으며, 동일한 시료들에 대하여 RT-PCR로 CMV 감염을 다시 검증한 결과, LF-RT-RPA 결과와 동일하게 6 개의 식물체에서만 CMV가 감염되어 있음을 증명하였다 (Fig. 8).

Application of the reverse transcription-recombinase polymerase amplification assay combined with a lateral flow immunostrip assay (LF-RT-RPA) for the detection of CMV on the leaves of C. officinale in farms.(A)∼(H) Symptomatic leaves of C. officinale plants (7 plants) showed mosaic, necrotic, chlorotic patch, or yellowing symptoms. (I) The samples were subjected to RT-RPA and the RT-RPA products were visualized in 2.0% agarose gel. CMV was detected on 6 samples of the 7 collected C. officinale plants by RT-RPA. All leaves for RT-RPA were confirmed the infection of CMV by RT-PCR. M, 100bp DNA ladder (Enzynomics, Daejeon, Korea), lane 1, NTC (non-template control, a healthy plant), lanes 2∼8, on-site detection samples of the collected C. officinale (named sample number 1 – sample number 7). (J) Lateral flow immunostrip assay showed positive reactions to CMV in on-site detection (lane 3 to lane 8). Meanwhile, the immunostrip for NTC (non-template control) did not have a false-positive band in the test line (lane 1).
고 찰
현재까지 식물 바이러스에 대한 농약이 개발되지 않았기 때문에 식물 바이러스는 조기 진단을 통해 이병주를 빨리 제거하는 것이 최선의 방제법이다 (Jones, 2006). 특히 천궁과 같이 영양번식 또는 뿌리 부분을 이용하는 작물의 경우 해마다 바이러스에 감염된 종묘를 심어서 피해가 증가될 수 있으므로 신속한 바이러스 진단이 중요하다 (Baranwal et al., 2020).
본 연구에서는 일천궁에서 CMV를 검출하기 위한 RT-RPA-LFI 진단 기술을 확립하였으며 이 진단 기술은 간단하고, 신속성과 민감성, 정확성의 강점을 가진 것임을 보여주었다. CMV RNA3 일부분과 특이적인 프라이머 1 쌍을 제작하여 이량체 (primer dimer) 형성을 하지 않아 특이성을 확인하였으며, 이 프라이머 쌍을 이용하여 CMV 검출을 위한 LF-RT-RPA 진단 기술은 3 개 일천궁 감염 바이러스와 비특이적 교차 반응이 나타나지 않았다 (Fig. 2).
대다수의 RT-RPA 진단은 프라이머의 영향을 줄이고 특이성을 더욱 높이고자 고안된 방법으로 nfo 프로브를 사용하여 CMV를 포함한 식물 바이러스 등을 진단하고 있으나 (Piepenburg et al., 2006, Srivastava et al., 2019), 본 연구에서는 nfo 프로브가 없이 1 쌍의 프라이머만 사용하여 특이적으로 천궁에서 CMV 검출 기술을 개발한 점은 기존 연구와의 차별성을 보여준다.
게다가, 실제 농장에서 재배되는 일천궁 잎에서 CMV를 검출한 결과 RT-RPA-LFI 검사의 적용 가능성이 입증되었으며 농장에서 CMV의 존재 여부를 확인하는 것은 RT-RPA 반응을 시작한 후 15 분부터 가능하였다. 본 연구에서 개발한 LF-RT-RPA 방법은 36℃ - 42℃에서 안정적으로 일정한 DNA를 증폭하여 CMV를 검출할 수 있음을 보여주었다 (Fig. 4).
이런 결과는 정밀한 보온 장비 없이 온도의 오차가 있을 수 있는 현장에서도 체온 (손), 핫 팩 등을 이용하여 CMV를 진단할 수 있음을 보여주었다 (Fig. 7). RT-RPA (또는 RPA)는 RT-PCR과 비교해 볼 때 비교적 낮은 27℃ - 44℃에서 DNA 증폭이 가능하므로 현장에서 별도의 보온 장비 없이 바이러스와 병원균 검출을 할 수 있는 사례들과 유사하다 (Crannell et al., 2014, Lillis et al., 2014, Yu et al., 2019).
현재까지 여러 식물 종에서 CMV를 진단할 수 있는 많은 기술이 개발되었다. 이러한 기술들은 agar gel immunodiffusion (Scott, 1968; Rao et al., 1982), western immunoblot (Hsu et al., 1989), immunoelectron microscopy (Francki and Hatta, 1980), double antibody sandwich enzyme-linked immunosorbent assay (DAS-ELISA) 등 혈청학적 진단법 (Clark and Adam, 1977, Davis and Hampton, 1986, Devergne et al., 1981, Wahuyni et al., 1992)이 개발되었다.
유전자를 검출할 수 있는 분자생물학적 진단 기술로는 대표적으로 dot blot hybridization, northern blot hybridization, reverse transcription-PCR (RT-PCR), immuno-capture reverse transcription polymerase chain reaction (IC-RT-PCR), quantitative RT-PCR (RT-qPCR), RT loop-mediated isothermal amplification (RT-LAMP) 등의 진단법이 보고되었다 (Owen and Palukaitis, 1988; Hu et al., 1995; Takeshita et al., 1998; Choi et al., 1999; Sharman et al., 2000; Peng et al., 2012, Srivastava et al., 2019).
이런 기술들은 핵산 검출 민감도가 우수하여 정밀한 진단을 할 수 있다는 장점이 있으나 고가의 장비가 필요하고 숙련된 연구자가 실험실 환경에서 분석해야 하며 농장 등에서 현장 진단에 번거로움이 있다는 단점이 있다 (Stevens et al., 1997; Mekuria et al., 2003).
이러한 단점을 보완하여 현재까지 등온 증폭 기술을 이용한 현장 진단 기술로서 RT-RPA 기술은 주목을 받아왔다. CMV를 포함한 극소량의 식물 바이러스 RNA 검출을 할 수 있는 RT-RPA 기반 분석 기술 개발과 관련된 다수의 논문이 발표되고 있다 (Mekuria et al., 2014; Silva et al., 2014; Zhang et al., 2014; Lodoo et al., 2016; Babu et al., 2018; Lu et al., 2018; Babujee et al., 2019; Kim et al., 2019, 2020, 2022a, 2022b; Srivastava et al., 2019; Lee et al., 2021).
또한 식물 바이러스 검출을 위한 LF-RT-RPA 분석은 TwistDx 키트에 따라 수행되었으며 종단점 판독 방법 (end-point readout method)으로 LF immunostrip assay 방법을 활용한 사례가 보고되었다 (Babu et al., 2018, Kim et al., 2022a and 2022b). 이런 장점에도 불구하고 RT-PCR에 비해 LF-RT-RPA의 단점 중 하나는 시료 1 개당 진단 비용이 일반적인 RT-PCR 비용보다는 높다는 점이다. 진단 비용 절감을 위해 특히 LF immunostrip 제조에 들어가는 재료 중 유리섬유 또는 저렴한 종이로 대체 가능성을 보여준 사례가 발표되었다 (Lobato and O’Sullivan, 2018). 본 연구는 TwitAmp basic kit를 이용하여 천궁에서 CMV RNA를 검출하는 LF-RT-RPA 진단기술 개발에 대한 최초의 보고이다.
Acknowledgments
본 연구는 농촌진흥청 공동연구사업(과제번호: PJ014947022023) 지원에 의해 이루어진 것으로 이에 감사드립니다.
References
-
Babu B, Ochoa-Corona FM and Paret ML. (2018). Recombinase polymerase amplification applied to plant virus detection and potential implications. Analytical Biochemistry. 546:72-77.
[https://doi.org/10.1016/j.ab.2018.01.021]

-
Babujee L, Witherell RA, Mikami K, Aiuchi D, Charkowski AO and Rakotondrafara AM. (2019). Optimization of an isothermal recombinase polymerase amplification method for real-time detection of Potato virus Y, O and N types in potato. Journal of Virological Methods. 267:16-21.
[https://doi.org/10.1016/j.jviromet.2019.02.006]

-
Baranwal VK, Kapoor R, Kumar S and Srivastava N. (2020). Recent advances of virus diagnostics in horticultural crops. In Awasthi LP. (ed.). Applied plant virology: Advances, detection, and antiviral strategies. Academic Press. Cambridge, MA, USA. p.27-37.
[https://doi.org/10.1016/B978-0-12-818654-1.00002-5]

-
Belete MT, Igori D, Kim SE, Lee SH and Moon JS. (2022). Complete genome sequence of cnidium virus 1, a novel betanucleorjadovirus infecting Cnidium officinale. Archives of Virology. 167:973-977.
[https://doi.org/10.1007/s00705-021-05348-9]

-
Bujarski J, Gallitelli D, García-Arenal F, Pallás V, Palukaitis P, Reddy MK and Wang A. (2019). ICTV virus taxonomy profile: Bromoviridae. Journal of General Virology. 100:1206-1207.
[https://doi.org/10.1099/jgv.0.001282]

-
Bujarski J. (2021). Bromoviruses (Bromoviridae). In Encyclopedia of Virology. Elsevier. Amsterdam, Netherlands. p.260-267.
[https://doi.org/10.1016/B978-0-12-809633-8.21563-X]

-
Cho JD, Kim JS, Lee SH, Choi GS and Chung BN. (2007). Viruses and symptoms on peppers, and their infection types in Korea. Research in Plant Disease. 13:75-81.
[https://doi.org/10.5423/RPD.2007.13.2.075]

- Cho SK, Kwon OI and Kim CJ. (1996). Anti-inflammatory and analgesic activities of the extracts and fractions of cnidii rhizoma. Korean Journal of Pharmacognosy. 27:282-287.
-
Choi SK, Choi JK, Park WM and Ryu KH. (1999). RT-PCR detection and identification of three species of cucumoviruses with a genus-specific single pair of primers. Journal of Virological Methods. 83:67-73.
[https://doi.org/10.1016/S0166-0934(99)00106-8]

- Choi YM, Lee SH, Kim JS and Lee EK. (1985). An investigation of undescribed witches' broom symptom disease caused by mycoplasma-like organism on Bupleurum falcatum, Cnidium officinale and Plantago asiatica in Korea. The Korean Journal of Mycology. 13:49-51.
-
Chung BN, Kwon SJ, Yoon JY and Cho IS. (2022). First report of Cnidium officinale as a natural host plant of apple stem grooving virus in South Korea. Plant Disease. 106:338-338.
[https://doi.org/10.1094/PDIS-04-21-0781-PDN]

-
Clark MF and Adams AN. (1977). Characteristics of the microplate method of enzyme-linked immunosorbent assay for the detection of plant viruses. Journal of General Virology. 34:475-483.
[https://doi.org/10.1099/0022-1317-34-3-475]

-
Crannell ZA, Rohrman B and Richards-Kortum R. (2014). Equipment-free incubation of recombinase polymerase amplification reactions using body heat. PLOS One. 9:e112146. https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0112146, (cited by 2023 Nov 18).
[https://doi.org/10.1371/journal.pone.0112146]

-
Davis RF and Hampton RO. (1986). Cucumber mosaic virus isolates seedborne in Pahseolus vulgaris: Serology, host-pathogen relationships, and seed transmission. Phytopathology. 76:999-1004.
[https://doi.org/10.1094/Phyto-76-999]

-
Devergne JC, Cardin L, Burckard J and van Regenmortel MHV. (1981). Comparison of direct and indirect ELISA for detecting antigenically related cucumovirus. Journal of Virological Methods. 3:193-199.
[https://doi.org/10.1016/0166-0934(81)90070-7]

-
Edwardson JR and Christie RG. (1991). Cucumoviruses. In: CRC Handbook of viruses infecting legumes. CRC Press, Boca Raton, FL, USA. p.293-319.
[https://doi.org/10.1201/9781351071192-16]

-
Francki RIB and Hatta T. (1980). Cucumber mosaic virus-Variation and problems of identification. Acta Horticulturae. 110:167-174.
[https://doi.org/10.17660/ActaHortic.1980.110.21]

-
Han K, Lee H, Park YM, Jeon KS, Choen W and Jung CR. (2021). The first report of Fusarium solani causing wilting in Cnidium officinale in Korea. Research in Plant Disease. 27:76-78.
[https://doi.org/10.5423/RPD.2021.27.2.76]

-
Honma H, Tsushima D, Kawakami H, Fujihara N, Tsusaka T, Kawashimo M, Nishimura T and Fuji S. (2019). Complete nucleotide sequence of a new potexvirus, ‘Cnidium virus X’, isolated from Cnidium officinale in Japan. Archives of Virology. 164:1931-1935.
[https://doi.org/10.1007/s00705-019-04261-6]

-
Hord MJ, Garciá A, Villalobos H, Rivera C, Macaya G and Roossinck MJ. (2001). Field survey of Cucumber mosaic virus subgroup I and II in crop plants in Costa Rica. Plant Disease. 85:952-954.
[https://doi.org/10.1094/PDIS.2001.85.9.952]

-
Hsu YC, Yeh TJ and Chang YC. (2005). A new combination of RT-PCR and reverse dot blot hybridization for rapid detection and identification of potyviruses. Journal of Virological Methods. 128:54-60.
[https://doi.org/10.1016/j.jviromet.2005.04.002]

-
Hu JS, Li HP, Barry K, Wang M and Jordan R. (1995). Comparison of dot blot, ELISA and RT-PCR assays for detection of two cucumber mosaic virus isolates infecting banana in Hawaii. Plant Disease. 79:902-906.
[https://doi.org/10.1094/PD-79-0902]

- Hwang J and Yang M. (1998). Comparision of chemical components of Ligusticum chuanzxiong Hort and Cnidium officinale Makino. Analytical Science and Technology. 11:54-61.
-
Jones RAC. (2006). Control of plant virus diseases. Advances in Virus Research. 67:205-244.
[https://doi.org/10.1016/S0065-3527(06)67006-1]

-
Jung CR, Jeong DH, Park HW, Kim HJ, Jeon KS and Yoon JB. (2019). Molecular identification of thrips in two medicinal crops, Cnidium officinale Makino and Ligusticum chuanxiong Hort. Korean Journal of Medicinal Crop Science. 27:17-23.
[https://doi.org/10.7783/KJMCS.2019.27.1.17]

- Jung CR, Kim YS, Park YM, Jeong DH, Jeon YH and Jeon KS. (2018). Selection of natural product extracts against Phoma sp. and Colletotrichum gloesporioides occurred in Korean angelica (Angelica gigas Nakai) and cnidii rhizoma(Cnidium officinale Makino). Proceedings of the Korean Journal of Medicinal Crop Science. 26:30-30.
-
Kim DH, Park JM, Kang SM, Lee SM, Seo CW, Lee IY and Lee IJ. (2015). Distribution characteristics of weeds and vegetation types in Cnidium officinale field. Weed & Turfgrass Science. 4:279-287.
[https://doi.org/10.5660/WTS.2015.4.4.279]

-
Kim DH, Jeong RD, Choi S, Ju HJ and Yoon JY. (2022a). Application of apid and reliable detection of cymbidium mosaic virus by reverse transcription recombinase polymerase amplification combined with lateral flow immunoassay. Plant Pathology Journal. 38:665-672.
[https://doi.org/10.5423/PPJ.FT.10.2022.0147]

-
Kim DH, Park JM, Kang SM, Lee SM, Seo CW, Lee IY and Lee IJ. (2015). Distribution characteristics of weeds and vegetation types in Cnidium officinale field. Weed and Turfgrass Science. 4:279-287.
[https://doi.org/10.5660/WTS.2015.4.4.279]

-
Kim JS, Lee SH, Choi,HS, Kim MK, Kwak HR, Nam M, Kim JS, Choi GS, Cho JD and Cho IS. (2011). Occurrence of virus diseases on major crops in 2010. Research in Plant Disease. 17:334-341.
[https://doi.org/10.5423/RPD.2011.17.3.334]

-
Kim JY and Lee JL. (2017). Development of a multiplex real-time recombinase polymerase amplification(RPA) assay for rapid quantitative detection of Campylobacter coli and jejuni from eggs and chicken products. Food Control. 73:1247-1255.
[https://doi.org/10.1016/j.foodcont.2016.10.041]

-
Kim NK, Kim SM and Jeong RD. (2020). Reverse transcription recombinase polymerase amplification assay for rapid and sensitive detection of barley yellow dwarf virus in oat. Plant Pathology Journal. 36:497-502.
[https://doi.org/10.5423/PPJ.NT.08.2020.0148]

-
Kim NK, Lee HJ, Kim SM and Jeong RD. (2022b). Rapid and visual detection of barley yellow dwarf virus by reverse transcription recombinase polymerase amplification with lateral flow strips. Plant Pathology Journal. 38:159-166.
[https://doi.org/10.5423/PPJ.NT.01.2022.0009]

-
Kim NY, Lee HJ and Jeong RD. (2019). A portable detection assay for apple stem pitting virus using reverse transcription- recombinase polymerase amplification. Journal of Virological Methods. 274:113747. https://www.sciencedirect.com/science/article/abs/pii/S0166093419301375, (cited by 2023 Nov 18).
[https://doi.org/10.1016/j.jviromet.2019.113747]

- Lee CB. (1985). The encyclopeida of Korea plants. Hyang Mun Sa, Seoul, Korea. p.583.
-
Lee DS, Lee HK, Kim SY, Kwon BR, Yang HJ, Park CY and Lee SH. (2023). Complete genome sequences of two apple stem grooving viruses in Cnidium officinale in Korea. Microbiology Resource Announcements. 12:e00902-22. https://journals.asm.org/doi/full/10.1128/mra.00902-22, (cited by 2023 Nov 18).
[https://doi.org/10.1128/mra.00902-22]

-
Lee HJ, Cho IS, Ju HJ and Jeong RD. (2021). Rapid and visual detection of Tomato spotted wilt virus using recombinase polymerase amplification combined with lateral flow strips. Molecular and Cellular Probes. 57:101727. https://www.sciencedirect.com/science/article/abs/pii/S0890850821000347, (cited by 2023 Nov 18).
[https://doi.org/10.1016/j.mcp.2021.101727]

-
Lee HJ, Kim HJ, Lee K and Jeong RD. (2020). Rapid detection of Peach latent mosaic viroid by reverse transcription recombinase polymerase amplification. Molecular and Cellular Probes. 53:101627. https://www.sciencedirect.com/science/article/abs/pii/S0890850820303194, (cited by 2023 Nov 18).
[https://doi.org/10.1016/j.mcp.2020.101627]

- Lee SC and Chang YC. (2006). Multiplex RT-PCR detection of two orchid viruses with an internal control of plant nad5 mRNA. Plant Pathology Bulletin. 15:187-196.
-
Li TT, Wang JL, Zhang NZ, Li WH, Yan HB, Li L, Jia WZ and Fu BQ. (2019). Rapid and visual detection of Trichinella spp. using a lateral flow strip-based recombinase polymerase amplification(LF-RPA) assay. Frontiers in Cellular and Infection Microbiology. 9:1. https://www.frontiersin.org/articles/10.3389/fcimb.2019.00001/full, (cited by 2023 Nov 18).
[https://doi.org/10.3389/fcimb.2019.00001]

-
Lillis L, Lehman D, Singhal MC, Cantera J, Singleton J, Labarre P, Toyama A, Piepenburg O, Parker M and Wood R. (2014). Non-instrumented incubation of a recombinase polymerase amplification assay for the rapid and sensitive detection of proviral HIV-1 DNA. PLOS One. 9:e108189. https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0108189, (cited by 2023 Nov 18).
[https://doi.org/10.1371/journal.pone.0108189]

-
Lobato IM and O’Sullivan CK. (2018). Recombinase polymerase amplification: Basics, applications and recent advances. Trends Analytical Chemistry. 98:19-35.
[https://doi.org/10.1016/j.trac.2017.10.015]

-
Londoño MA, Harmon CL and Polston JE. (2016). Evaluation of recombinase polymerase amplification for detection of begomoviruses by plant diagnostic clinics. Virology Journal. 13:48. https://link.springer.com/article/10.1186/s12985-016-0504-8, (cited by 2023 Nov 18).
[https://doi.org/10.1186/s12985-016-0504-8]

-
Lu Y, Yao B, Wang G and Hong N. (2018). The detection of ACLSV and ASPV in pear plants by RT-LAMP assays. Journal of Virological Methods. 252:80-85.
[https://doi.org/10.1016/j.jviromet.2017.11.010]

-
Mekuria TA, Zhang S and Eastwel KC. (2014). Rapid and sensitive detection of Little cherry virus 2 using isothermal reverse transcription-recombinase polymerase amplification. Journal of Virological Methods. 205:24-30.
[https://doi.org/10.1016/j.jviromet.2014.04.015]

- National Institute of Horiticultural and Herbal Science(NIHHS). (2017). Impact assessment based on climate change scenarios (RCP) in apple, grape, mandarin, ginseng, cnidium, and Korean Angelica. National Institute of Horiticultural and Herbal Science. Rural Development Administration. Wanju, Korea. p.89-125.
- Oh YJ, Seo HR, Choi YM and Jung DS. (2010). Evaluation of antioxidant activity of the extracts from the aerial parts of Cnidium officinale Makino. Korean Journal of Medicinal Crop Science. 18:373-378.
-
Ortega-Acosta C, Ochoa-Martínez DL and Hernández-Morales J. (2019). First report of cucumber mosaic virus subgroup I and II with satellite RNA infecting Nicotiana glauca in Mexico. Journal of Plant Pathology. 101:801. https://link.springer.com/article/10.1007/s42161-019-00270-1, (cited by 2023 Nov 18).
[https://doi.org/10.1007/s42161-019-00270-1]

-
Owen J and Palukaitis P. (1988). Characterization of cucumber mosaic virus. I. Molecular heterogeneity mapping of RNA3 in eight CMV strains. Virology. 166:495-502.
[https://doi.org/10.1016/0042-6822(88)90520-X]

-
Palukaitis P and García-Arenal F. (2003). Cucumoviruses. Advances in Virus Research. 62:241-323.
[https://doi.org/10.1016/S0065-3527(03)62005-1]

-
Palukaitis P, Roossinck MJ, Dietzgen RG and Francki RIB. (1992). Cucumber mosaic virus. Advances in Virus Research. 41:281-348.
[https://doi.org/10.1016/S0065-3527(08)60039-1]

-
Park YC, Kim KK, Jun HJ, Kang EH, Lee SR, Ahn JK, Seo HK, Lee SH and Hoe, KL. (2023). Complete genome sequence of cnidium virus 2, a novel cytorhabdovirus isolated from Cnidium officinale in South Korea. Archives of Virology. 168:170. https://link.springer.com/article/10.1007/s00705-023-05784-9, (cited by 2023 Nov 18).
[https://doi.org/10.1007/s00705-023-05784-9]

- Park YK. (1998). The study on antioxidative effects and quality comparison of Ligusticum chuanxiong and Cnidium officinale (Ⅰ). The Korea Journal of Herbology. 13:103-114.
-
Peng J, Shi M, Xia Z, Huang J and Fan Z. (2012). Detection of Cucumber mosaic virus isolates from banana by one step reverse transcription loop-mediated isothermal amplification. Archives of Virology. 157:2213–2217.
[https://doi.org/10.1007/s00705-012-1376-x]

-
Piepenburg O, Williams CH, Stemple DL and Armes NA. (2006). DNA detection using recombination proteins. PLOS Biology. 4:e204. https://journals.plos.org/plosbiology/article?id=10.1371/journal.pbio.0040204, (cited by 2023 Nov 18).
[https://doi.org/10.1371/journal.pbio.0040204]

-
Rao ALN, Hatta T and Francki RIB. (1982). Comparative studies on tomato aspermy virus and cucumber mosaic viruses VII. Serological relationships reinvestigated. Virology. 116:318-326.
[https://doi.org/10.1016/0042-6822(82)90423-8]

-
Scott HA. (1968). Serological behavior of cucumber mosaic virus(strain Y) and the virus protein. Virology. 34:79-90.
[https://doi.org/10.1016/0042-6822(68)90010-X]

- Seo YJ, Nam HH, Jang WC, Kim JS and Lee BY. (2018). Effect of meteorological factors on evapotranspiration change of Cnidium officinale Makino. Korean Journal of Agricultural and Forest Meteorology. 20:366-375.
-
Shaman M, Gambley CF, Oloteo EO, Abgona RVJ and Thomas JE. (2000). First record of natural infection of abaca(Musa textilis) with Banana bract mosaic potyvirus in the Philippines. Australian Plant Pathology. 29:69. https://link.springer.com/article/10.1071/AP00012, (cited by 2023 Nov 18).
[https://doi.org/10.1071/AP00012]

-
Silva G, Bömer M, Nkere C, Kumar PL and Seal SE. (2014). Rapid and specific detection of yam mosaic virus by reverse-transcription recombinase polymerase amplification. Journal of Virological Methods. 222:138-144.
[https://doi.org/10.1016/j.jviromet.2015.06.011]

- Song IG, An BR, Seo BI and Park SJ. (2009). Molecular marker to identify and origin of cnidii rhizoma from Korea and China. The Korea Journal of Herbology. 24:1-8.
-
Srivastava N, Kapoor R, Kumar R, Kumar S, Saritha RK, Kumar S and Baranwal VK. (2019). Rapid diagnosis of cucumber mosaic virus in banana plants using a fluorescence-based real-time isothermal reverse transcription-recombinase polymerase amplification assay. Journal of Virological Methods. 270:52-58.
[https://doi.org/10.1016/j.jviromet.2019.04.024]

-
Stevens M, Hull R and Smith HG. (1997). Comparison of ELISA and RT-PCR for the detection of beet yellows closterovirus in plants and aphids. Journal of Virological Methods. 68:9-16.
[https://doi.org/10.1016/S0166-0934(97)00103-1]

-
Takeshita M, Suzuki M, Kuwata S and Takanami Y. (1998). Involvement of cucumber mosaic cucumovirus RNA2 and RNA3 in viral systemic spread in radish plant. Archives of Virology. 143:1109-1117.
[https://doi.org/10.1007/s007050050359]

-
Um JN, Min JW, Joo KS and Kang HC. (2017). Antioxidant, anti-wrinkle activity and whitening effect of fermented mixture extracts of Angelica gigas, Paeonia lactiflora, Rehmannia chinensis and Cnidium officinale. Korean Journal of Medicinal Crop Science. 25:152-159.
[https://doi.org/10.7783/KJMCS.2017.25.3.152]

-
Wahyuni WS, Dietzgen RG, Hanada K and Francki RIB. (1992). Serological and biological variation between and within subgroup I and II strains of cucumber mosaic virus. Plant Pathology. 41:282-297.
[https://doi.org/10.1111/j.1365-3059.1992.tb02350.x]

- Yang HJ, Lee HK, Lee DS, Kim SY, Kwon B, Oh J, An C, Kwon DY, Lim S, Moon JS and Lee SH. (2018). Survey of virus occurrence in Cnidium officinale and Ligustucum chuanxiong. Proceedings of the Korean Journal of Pesticide Science. 304- 304.
-
Yoo RH, Zhao F, Lim S, Igori D, Lee SH and Moon JS. (2015). The complete nucleotide sequence and genome organization of lychnis mottle virus. Archives of Virology. 160:2891-2894.
[https://doi.org/10.1007/s00705-015-2501-4]

-
Yun YB, Um Y, Huh JH, Son HJ, Song YG and Lee KC. (2022). Changes in growth chracteristics and physiological activity of Cnidium officinale Makino according to the Climate Change Scenario(RCP). Korean Journal of Medicinal Crop Science. 30:450-461.
[https://doi.org/10.7783/KJMCS.2022.30.6.450]

-
Zhang S, Ravelonandro M, Russell P, McOwen N, Briard P, Bohannon S and Vrient A. (2014). Rapid diagnostic detection of plum pox virus in prunus plants by isotheral AmplifyRP using reverse transcription-recombinase polymerase amplification. Journal of Virological Methods. 207:114-120.
[https://doi.org/10.1016/j.jviromet.2014.06.026]

-
Zhao C, Sun F, Li X, Lan Y, Du L, Zhou T and Zhou Y. (2019). Reverse transcription-recombinase polymerase amplification combined with lateral flow strip for detection of rice black- streaked dwarf virus in plants. Journal of Virological Methods. 263:96-100.
[https://doi.org/10.1016/j.jviromet.2018.11.001]

-
Zhao L, Cheng J, Hao X, Tian X and Wu Y. (2012). Rapid detection of tobacco viruses by reverse transcription loop- mediated isothermal amplification. Archives of Virology. 157: 2291-2298.
[https://doi.org/10.1007/s00705-012-1441-5]

- Zhao XJ, Deng YJ, Wei ZL, Zhang W, Huang GL, Li B, Chen DX, Qing L and Sun XC. (2016). Development of a RT- LAMP method for the rapid detection of cucumber mosaic virus from vegetables. Acta Horticulturae Sinica. 43:1203-1210.
